大学共同利用機関法人 情報・システム研究機構
データサイエンス共同利用基盤施設
ライフサイエンス統合データベースセンター
小野 浩雅
[email protected]
2019年8月28日(水)
AJACS宮崎2 @ 宮崎大学 木花キャンパス 情報基盤センター
これは統合データベース講習会 AJACS宮崎2「遺伝子発現データの生物学的解釈を助けるデータベース・ウェブツールの使い方」の講習資料です。
講習会全体のプログラムはこちらです。
© 2019 小野 浩雅, CC-BY-4.0
本講習だれでも自由に使うことができる公共データベースやウェブツールを活用して、研究のさまざまな場面で調べることの多い個々の遺伝子発現データを簡単に調べるための方法と基礎知識について学びます。続いて、数百~数千におよぶ遺伝子セットについて、生物学的な解釈をする方法とその結果の考察を実践します。
今回の講習では、コンピュータを使って以下の内容について説明します。
-
個々の遺伝子の発現プロファイルを調べる
- RefEx
- 【実習】RefExを使って、組織特異的遺伝子を検索する
- RefEx
-
数十~数千の遺伝子群の生物学的解釈
- ChIP-Atlas
- 【実習】ChIP-AtlasのEnrichment Analysis を使って、興味ある遺伝子リストを制御する可能性の高い転写因子を調べる
- ChIP-Atlas
-
研究現場で頻繁に使われるデータベースやツールを知る
- 統合TV
-
みんなで同時にアクセスするとサイトにつながりにくくなることが予想されます。
- 資料を見ながら自力で進められそうな方はどんどん先に、そうでない方は講師と一緒にすすめていきましょう。
- サイトの反応が悪い時はタイミングをずらして実行してみてください。
- 反応が無いからと言って何度もクリックするとますます繋がらなくなってしまいます。おおらかな気持ちで臨みましょう。
-
Web上で質問・コメントできるフォームを用意していますので、ご活用ください。
- https://www.sli.do (「新しいタブで開く」とよいです)
- ウェブブラウザで sli.do と入力(スマホでもアクセス可能)
- AJ78 と入力
- 質問をする (゚д゚)カンタンー
- 皆さんの質問一覧 https://wall.sli.do/event/3cabini9
-
こんなことは知ってて当たり前だと他の人に思われるような「シロウト」質問を歓迎します。
- 当たり前に感じる質問や一見関係なさそうな質問がでると、「そういう質問をしてもよいのだ」という空気になり、この講義から得られる情報が増え、皆さんの受講満足度が上がります(たぶん)。
- あなたがいま疑問に思ったことは、実は、隣の人やその隣の人もそう思っていることが多いです。
- 知っている人は講師を助けてください。サポート大歓迎です
- でも講師も知らないことは(多々)あります。(以下ループ)
| 統合TVを知っていますか? | 人数 | 割合 |
|---|---|---|
| 知らない | 27 名 | 47 % |
| 知っている | 18 名 | 32 % |
| 回答なし | 12 名 | 21 % |
| 自分で実験して得た、数十〜数千の遺伝子からなる 「遺伝子リスト」(例: 発現差のあった遺伝子など) を持っていますか? |
人数 | 割合 |
|---|---|---|
| これから実験をする・したい | 12 名 | 21 % |
| 公共データを活用する・したい | 7 名 | 12 % |
| 既に持っている | 9 名 | 16 % |
| 大規模発現解析の予定はない | 9 名 | 16 % |
| 回答なし | 20 名 | 35 % |
- さまざまな実験で得られたデータは、論文投稿時などに公共データベース上に登録し、その後誰でも参照可能になるようにすることが義務付けられていることが多いですが、それは遺伝子発現データについても同様です。公共データベースには多種多様な遺伝子発現データが日々大量に登録、蓄積され続けています。
- これらのデータをうまく活用すれば、例えば、予備実験をせずに済む、自分の実験結果を支持する知見が得られる、仮説立案の新たな切り口が得られるなど、多くのメリットがあります。
- 一方で、いざ使おうと思ったときに、その膨大さから、どのデータを選び、詳しく調べればよいのか分かりにくく、利用することが困難で諦めてしまうことが多いです。
- これらの困難さを軽減するために、いくつかの二次的なデータベースやツールが開発されています。これらをうまく使うことで、自分のデータと組み合わせられる遺伝子発現データをすばやく検索、発見し、活用できるようになるでしょう。
-
遺伝子発現解析の基準となるデータを快適に検索できるウェブツール
-
公共DBにある正常組織や細胞株における遺伝子発現データを再利用・整理
-
4つの異なる実験手法(EST、GeneChip、CAGE、RNA-seq)によって得られた正常組織、初代培養細胞、細胞株における遺伝子発現データを検索、閲覧可能
-
このツールでできること
- 正常組織における遺伝子発現データを調べる
- 測定手法による遺伝子発現量の差異を比較する
- 組織特異的遺伝子をワンタッチで検索可能
- 遺伝子発現解析などで見出された不詳な遺伝子群の機能および関係性を調べる
-
RefExで掲載されているデータはすべて再利用可能
- オリジナルデータの再処理方法の詳細はGitHubに
- 再処理済みの発現データやサンプルアノテーション等のすべてのデータはfigshareに
- 「The RefEx analysis」として論文に引用していただいた活用例
- Aberrant IDH3α expression promotes malignant tumor growth by inducing HIF-1-mediated metabolic reprogramming and angiogenesis, Oncogene, (22 December 2014) | doi:10.1038/onc.2014.411 @ Figure 6
- がん研究者が、発現解析実験で見出した数百個の治療標的・候補遺伝子の絞込みに使えないか検討した。
- これらの候補遺伝子の正常組織における発現量が低ければ、治療標的とした場合に悪影響・副作用が小さくなると仮説した。
- 実際に、これらの遺伝子の発現量をRefExで確認し、追加確認実験の優先順位付けを効率的に行うことができた。
- その他RefExを引用した論文の一覧はこちらでご覧いただけます。
- Hiromasa Ono, Osamu Ogasawara, Kosaku Okubo, Hidemasa Bono RefEx, a reference gene expression dataset as a web tool for the functional analysis of genes Scientific Data, 4:170105 DOI: 10.1038/sdata.2017.105
- 川路 英哉、粕川 雄也、坊農 秀雅、小野 浩雅 「FANTOM5データを誰でも活用できる形に」 Scientific Data誌著者インタビュー (平成29年8月29日) https://www.natureasia.com/ja-jp/scientificdata/papers-from-japan/fantom5
- 小野 浩雅・坊農 秀雅 「遺伝子発現解析の基準となるデータを快適に検索できるウェブツールRefEx」 ライフサイエンス新着論文レビュー (平成29年9月5日) DOI: 10.7875/first.author.2017.093
- 統合TV 「RefExの使い方」DOI: 10.7875/togotv.2014.009
- 【統合TV】RefExの使い方
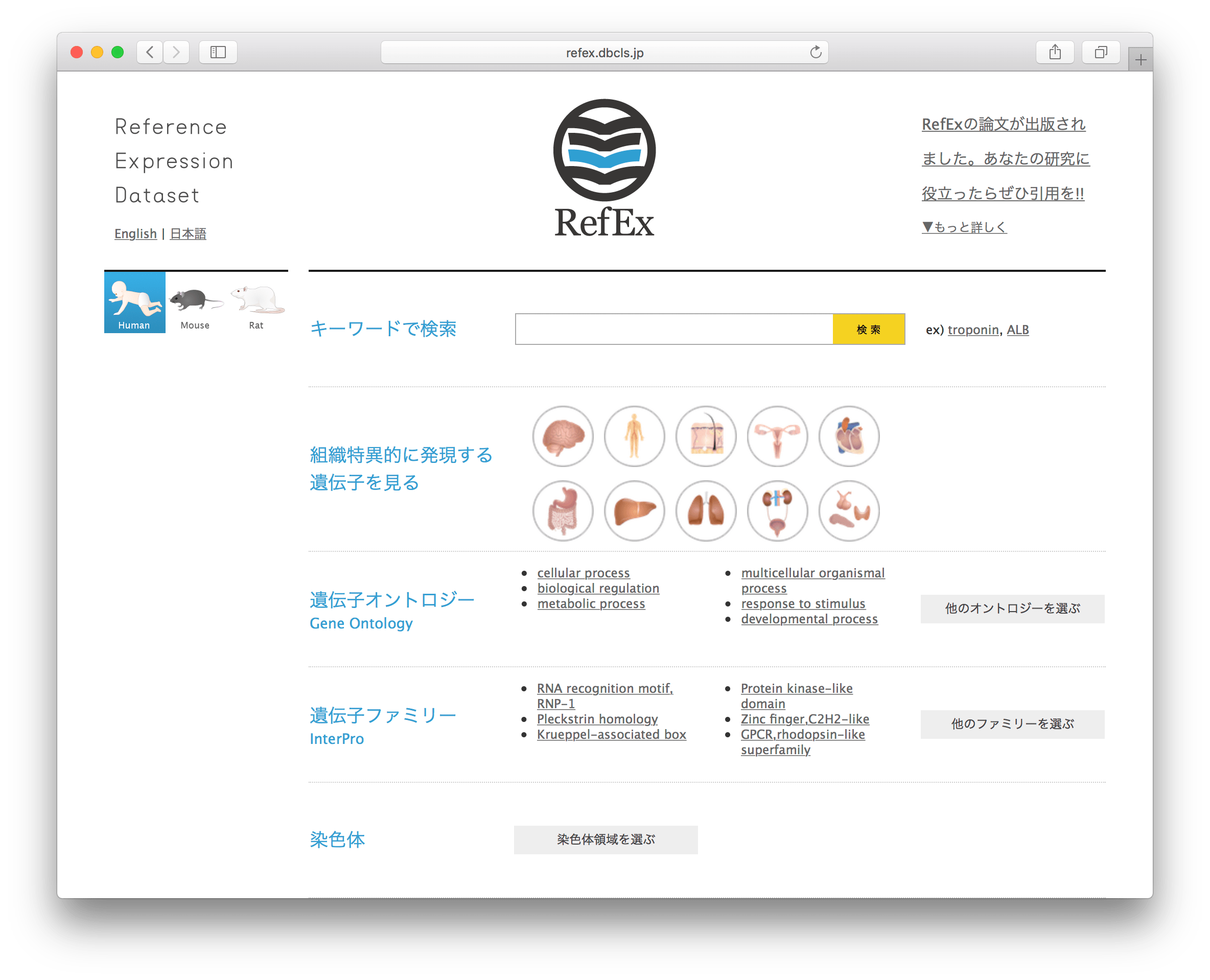
- https://refex.dbcls.jp/ を開きます。
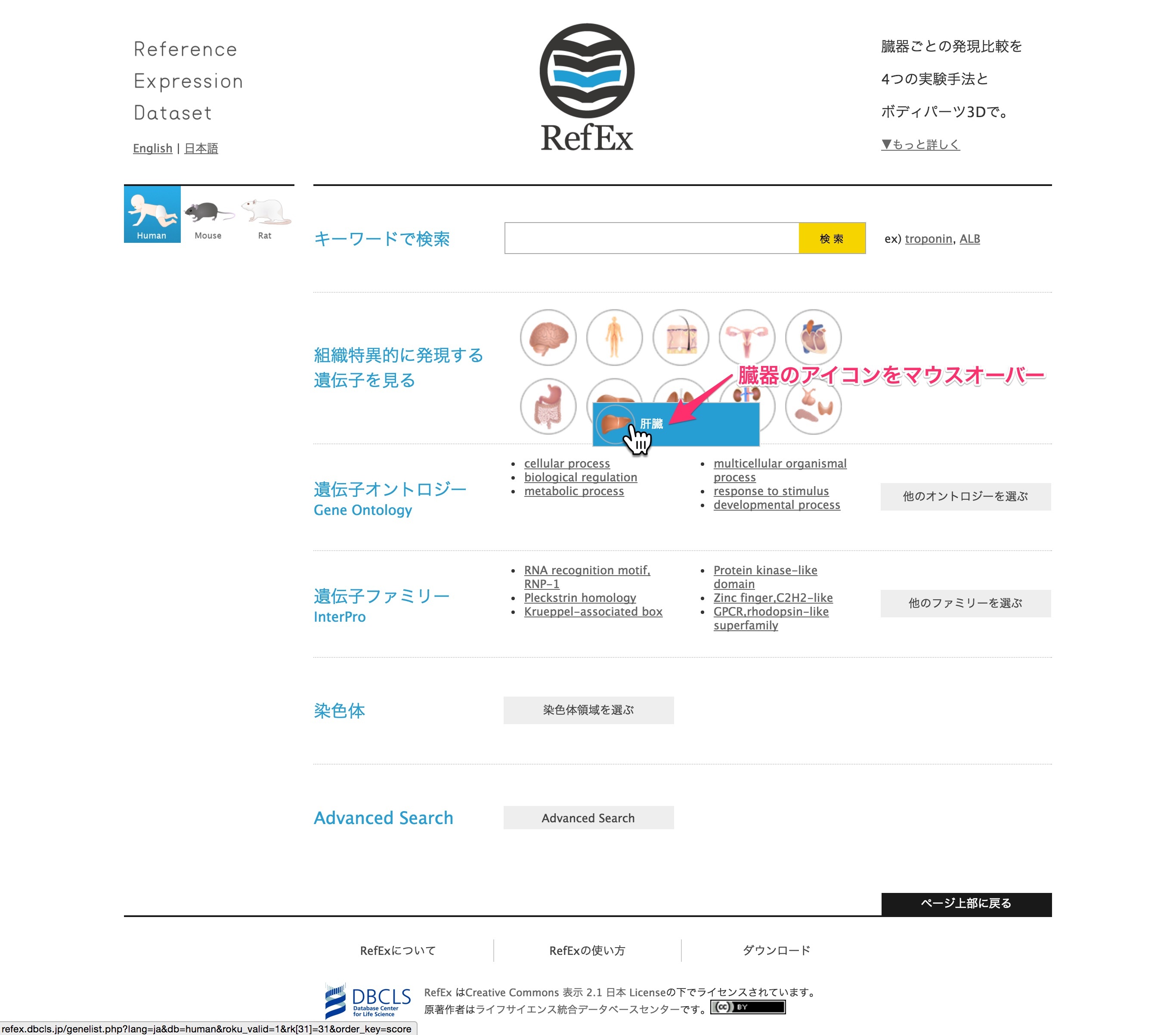
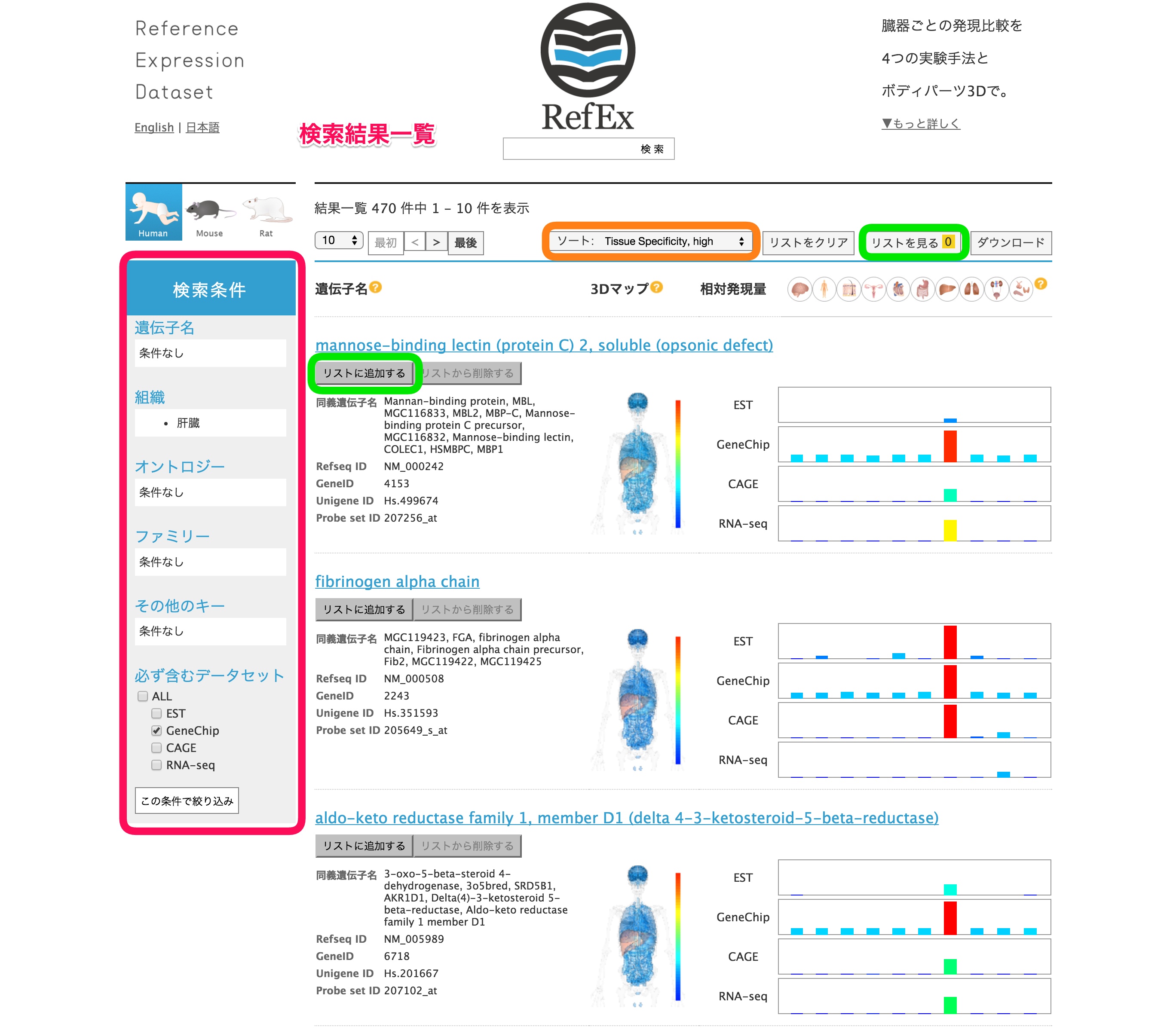
- 画面中央の「組織特異的に発現する遺伝子を見る」の臓器アイコンにカーソルを合わせると、更に詳細な部位のアイコンが出るので、調べたい臓器(例として肝臓)をクリックします。
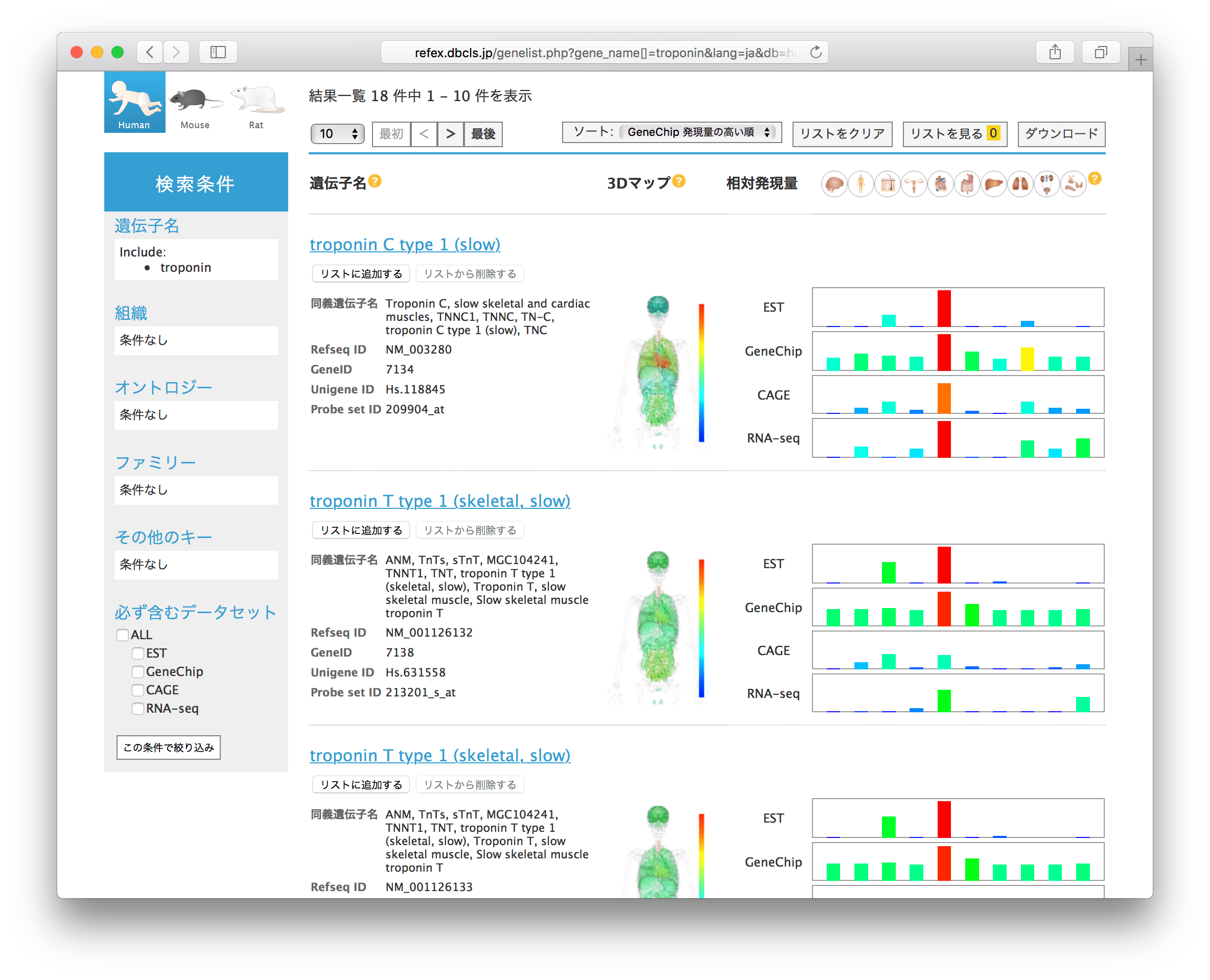
- 検索結果一覧が表示されます。検索結果一覧では、「ソート項目の切り替え」や「絞り込み検索」、「リストへの追加」ができます。(手順11以降で解説します。)
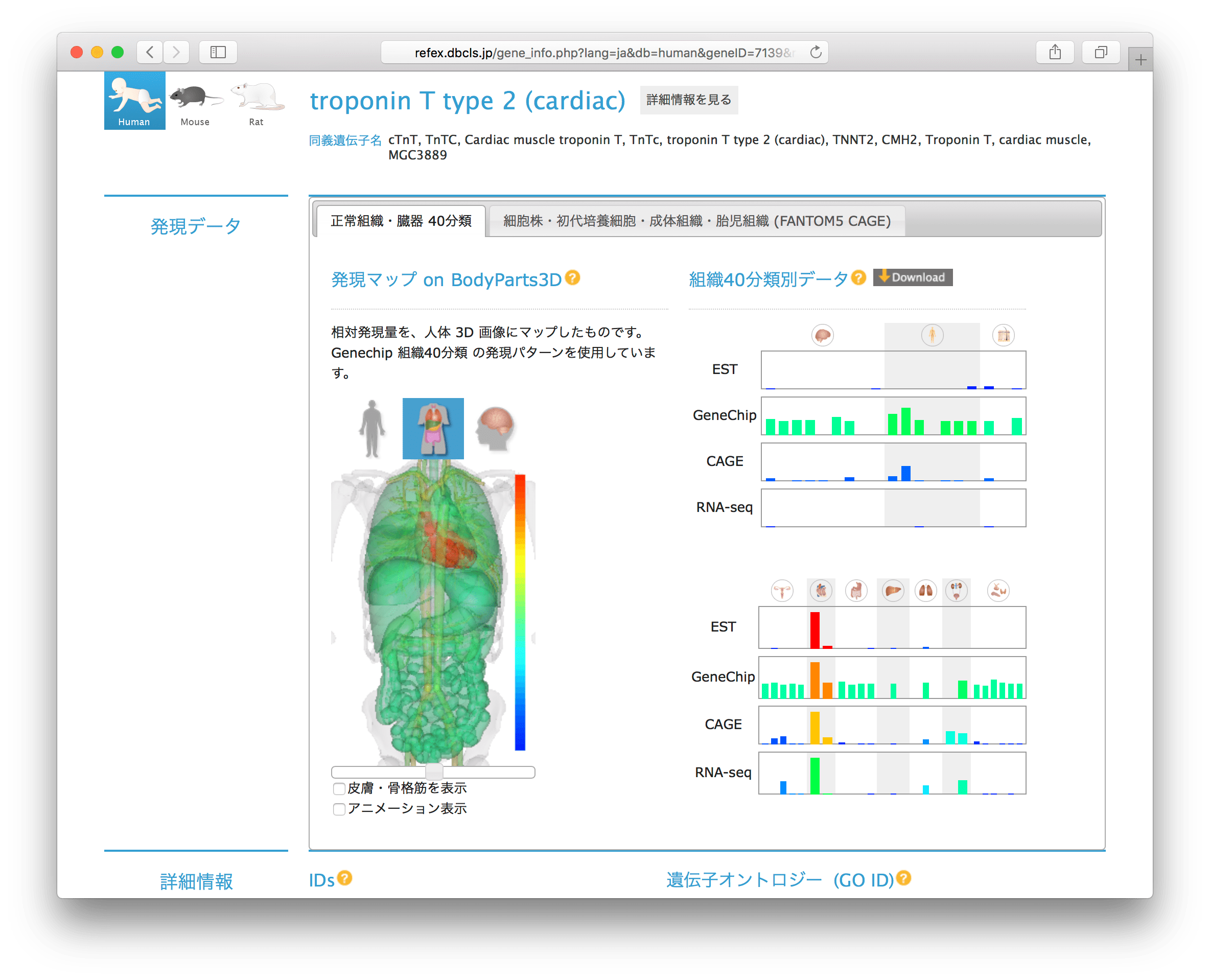
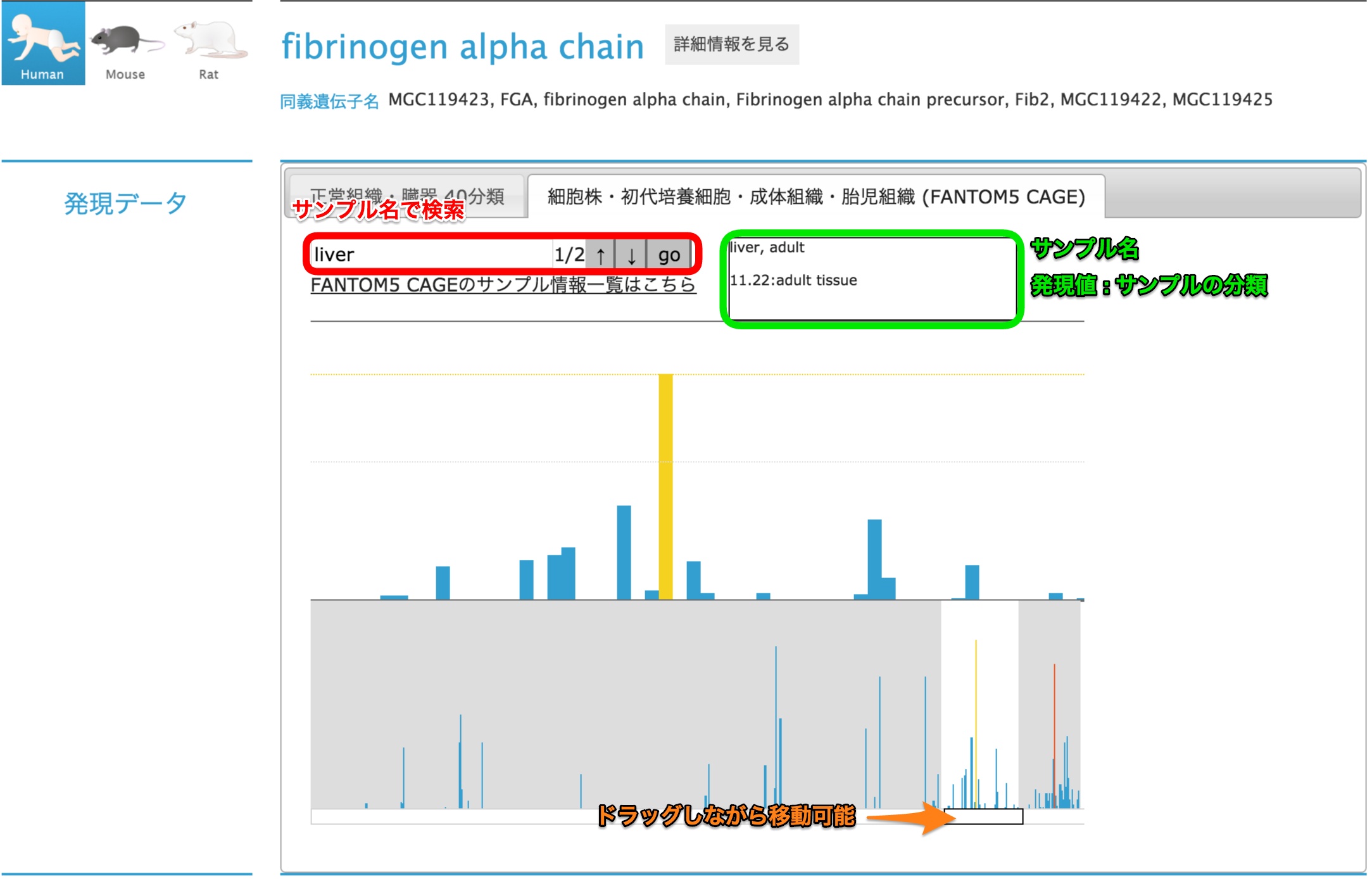
- 各遺伝子の青字の部分(例 fibrinogen alpha chain)をクリックすると詳細情報を閲覧できます。
- 「ヒートマップ on Bodyparts3D」では、表示する部位の切り替え(全身・体幹部・頭部)ができます。「皮膚・骨格筋を表示」もしくは「アニメーション表示」にチェックを入れるとどのように表示されるでしょうか。
- 「組織40分類別データ」では、バーの上にマウスオーバーすると測定部位と発現値が表示されます。
- 「Download」をクリックすると、表示中の遺伝子の組織40分類別の発現データがタブ区切り形式でダウンロードできます。
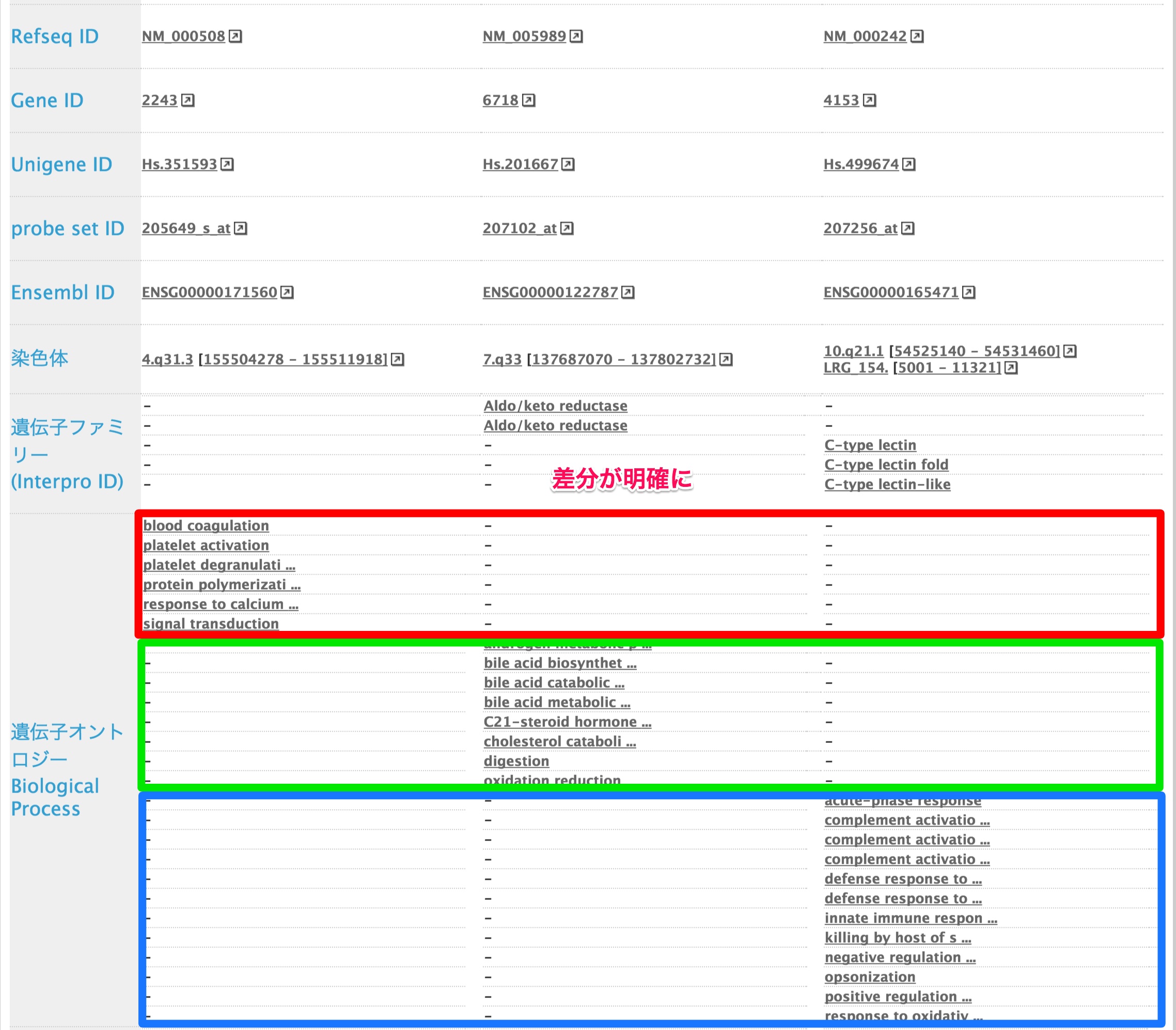
- 「Probe set ID」のリンク先をクリックすると、どういう情報が参照できるでしょうか。
- 遺伝子オントロジー(Gene Ontology:GO ID)をクリックすると、そのGO termを持つ他の遺伝子を一括で検索できます。
- 例として、GO:0007596 blood coagulation をクリックしてみましょう。
- 右側のFANTOM5 CAGEのタブをクリックすると、FANTOM5 CAGEデータのビューアに切り替わります。
- ビューアは上部が拡大図で、下部が全体表示になっています。
- 検索窓にキーワードを入れるとサンプル名を検索できます。ヒットしたサンプルはオレンジ色で強調されます。
- 右側に、サンプル名と発現値、サンプル分類が表示されます。
- RefEx用に整理したサンプル情報一覧も閲覧可能です。
- 検索結果一覧に戻ります。ソート項目を切り替えて、どのように結果が変わるでしょうか。
- 様々な条件で検索結果を絞り込むことができます。絞り込み検索は左のバーから行えます。
- 遺伝子名に「liver」を含むデータは何件あるでしょうか。
- 「遺伝子名」の下の「条件なし」をクリックして表示されるウインドウに「liver」と入力し、「Include」をクリックし、「この条件で絞り込み」を押します。
- 「遺伝子名」の項目で「Exclude」に「solute」を加えると、検索結果はどう変わるでしょうか。
- 「組織」の項目で、データ元をRNA-seqに変更したり、臓器の指定を追加すると検索結果はどう変わるでしょうか。
- 「必ず含むデータセット」の「ALL」にチェックを入れると、検索結果はどう変わるでしょうか。
- 個々の遺伝子の詳細情報は、リストに追加することで並列に比較することができます。
- 肝臓特異的遺伝子の検索結果一覧に移動して、3つの遺伝子を「リストに追加」してみましょう。
- 追加した件数は「リストを見る」の横に表示されます。
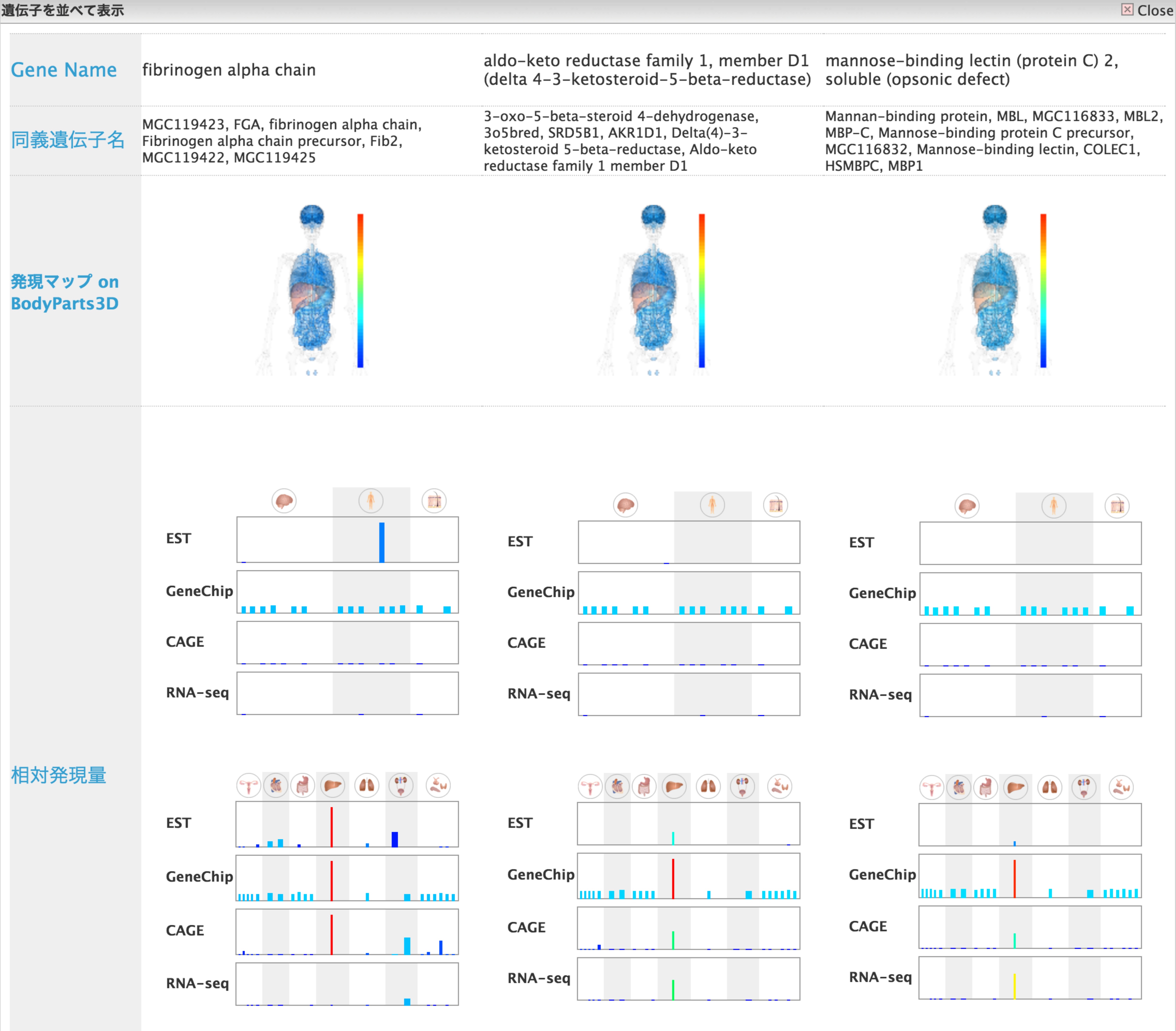
- 「リストを見る」をクリックするとリストに移動します。
- 『並べて表示する』にチェックを入れて、「遺伝子を並べて表示」をクリックします。
- 遺伝子発現データやGeneOntology情報を並列に比較することで見えてくる「違い」はなんでしょうか。その違いからどういうことが推測できるでしょうか。
- 自分の研究テーマに関連する、また興味のある遺伝子について検索してみましょう。
-
- Expression Atlas は、EMBL-EBI (欧州バイオインフォマティクス研究所) が運営する、様々な生物種における遺伝子発現情報を提供するウェブサイトである。40 の生物種について、3000 以上の実験に基づき、組織や細胞種、発生段階、疾患の有無などの条件別に、遺伝子の発現情報をまとめられています。
- 【統合TV】Expression Atlas で 様々な生物種の組織や疾患などにおける遺伝子発現の情報を調べる
-
- Bgee (a dataBase for Gene Expression Evolution: ビージーと発音します) は、SIB Swiss Institute of Bioinformatics/ローザンヌ大学の Robinson-Rechavi Group - Evolutionary Bioinformaticsが提供するデータベースで、これはRNA-seqやマイクロアレイなどから得られた遺伝子発現パターンについて、多くの生物種間で比較することができます。
- 【統合TV】Bgee を使って、複数の生物種の正常組織における遺伝子発現データを検索、比較、取得する
-
- AffymetrixのマイクロアレイであるGeneChipを用いたヒト、マウス、ラットを始め全9生物種におけるさまざまな組織や細胞(株)における遺伝子発現プロファイルのデータベースです。
- 【統合TV】遺伝子発現プロファイルデータベースBioGPSを使い倒す 2012
- マイクロアレイやNGS実験を行うと大量の発現変動遺伝子 (Differentially Expressed Genes: DEGs)が得られます。
- 一般的な遺伝子発現解析の第一歩は、実験条件によって得られた数十~数千のDEGsが生物学的にどういう意味を持つかを考えることです。
ChIP-Atlasは、論文などで報告された ChIP-seq データを閲覧し、利活用するためのウェブサービスです。データ処理の知識やスキルがない方でも簡単に利用できます。データソースは、公開 NGS データレポジトリ (NCBI, EMBL-EBI, DDBJ) に登録されたほぼ全ての ChIP-seq データです。ChIP-Atlas は、九州大学大学院医学研究院 発生再生学分野 (http://www.dev.med.kyushu-u.ac.jp) と DBCLS が共同で開発しています。
(https://chip-atlas.org/)
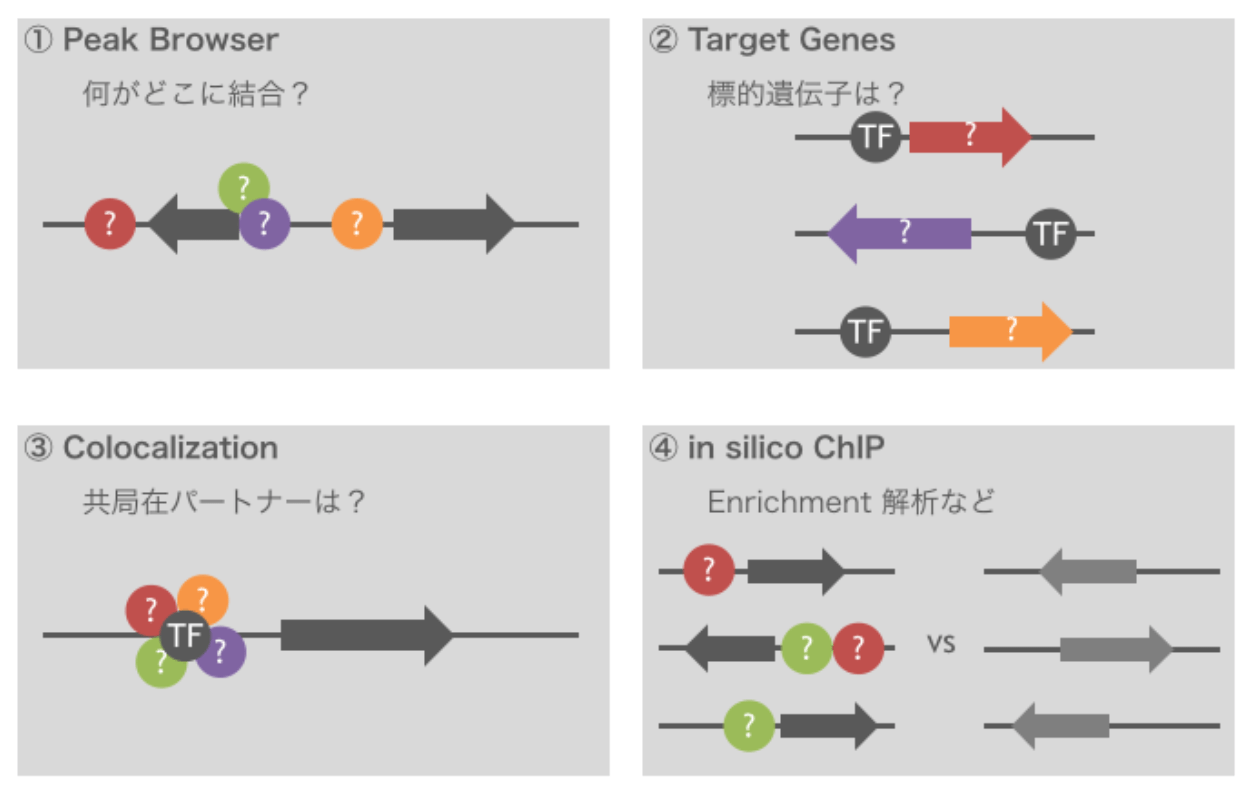
- 既報の ChIP-seq データをまとめて閲覧し、何がどこに結合しているかが一目でわかります。Integrative Genomics Viewer (IGV) によりスムーズなブラウジングが可能で、興味の遺伝子のシス調節領域を予測したり、それを制御する転写因子の予測ができます。
- 興味のある転写因子を選択し、その標的遺伝子候補を検索できます。
- 興味のある転写因子を選択し、それとゲノム上で共局在する転写因子候補を検索できます。
- ユーザデータを受け付け、既存データとの比較解析をおこないます。たとえば、興味のある遺伝子リストを submit すると、それらをまとめて制御する転写因子候補が返されます。ほかにも BED 形式のファイルや、シーケンスモチーフを submit すると、それらに enrichment する転写因子群が返されます。
- 【統合TV】ChIP-Atlasを使って興味ある遺伝子リストを制御する可能性の高い転写因子を調べる 〜Enrichment Analysisの使い方〜
- 論文として発表された ChIP-Seq データを閲覧したい
- 興味のあるゲノム領域における、転写因子や修飾ヒストンの分布を知りたい
- 興味のある転写因子の下流遺伝子や、複合体形成パートナーを知りたい
- 自身の研究データと公開 ChIP-seq データを用いて比較解析をおこないたい
- Source code and documentation
- Preprint
- Shinya Oki, Tazro Ohta, et al. Integrative analysis of transcription factor occupancy at enhancers and disease risk loci in noncoding genomic regions. bioRxiv 262899; doi: https://doi.org/10.1101/262899
- Paper
- Shinya Oki, Tazro Ohta, Go Shioi, Hideki Hatanaka, Osamu Ogasawara, Yoshihiro Okuda, Hideya Kawaji, Ryo Nakaki, Jun Sese, and Chikara Meno. ChIP-Atlas: a data-mining suite powered by full integration of public ChIP-seq data. EMBO Reports https://doi.org/10.15252/embr.201846255
- Database
- Oki, S; Ohta, T (2015): ChIP-Atlas. http://dx.doi.org/10.18908/lsdba.nbdc01558-000
- Publications citing ChIP-Atlas http://chip-atlas.org/publications
-
【統合TV】ChIP-Atlasを使って興味ある遺伝子リストを制御する可能性の高い転写因子を調べる 〜Enrichment Analysisの使い方〜
-
「発現差のあった遺伝子リスト」を持っている想定で、それらの遺伝子に結合しうる、あるいは上流でそれらの遺伝子の発現を制御する可能性がある転写因子を検索する
-
使用するデータ
- 190821_List_of_GeneSymbol_txt
- ある「興味ある遺伝子リスト」をGeneSymbolにID変換したデータ。
- これを使って、もともとどういう遺伝子リストだったかを考察します。
- ChIP-Atlas では、遺伝子IDとしてGeneSymbolのみを受け付けているので、それ以外のIDで遺伝子リストを持っている場合は、適宜変換が必要です。
- ID変換はいろいろなツールがありますが、今回はHGNC BioMartを利用します。
- HGNC(The HUGO Gene Nomenclature Committee)はヒトのGeneSymbolを認定・管理している機関。
- DAVID(Database for Annotation, Visualization and Integrated Discovery) のGene ID Conversion Toolも便利。(使い方動画)
- ID変換はいろいろなツールがありますが、今回はHGNC BioMartを利用します。
- 190821_List_of_GeneSymbol_txt
- ChIP-Atlas - Enrichment Analysisにアクセスします。
- 下図のようにオプションを設定します。
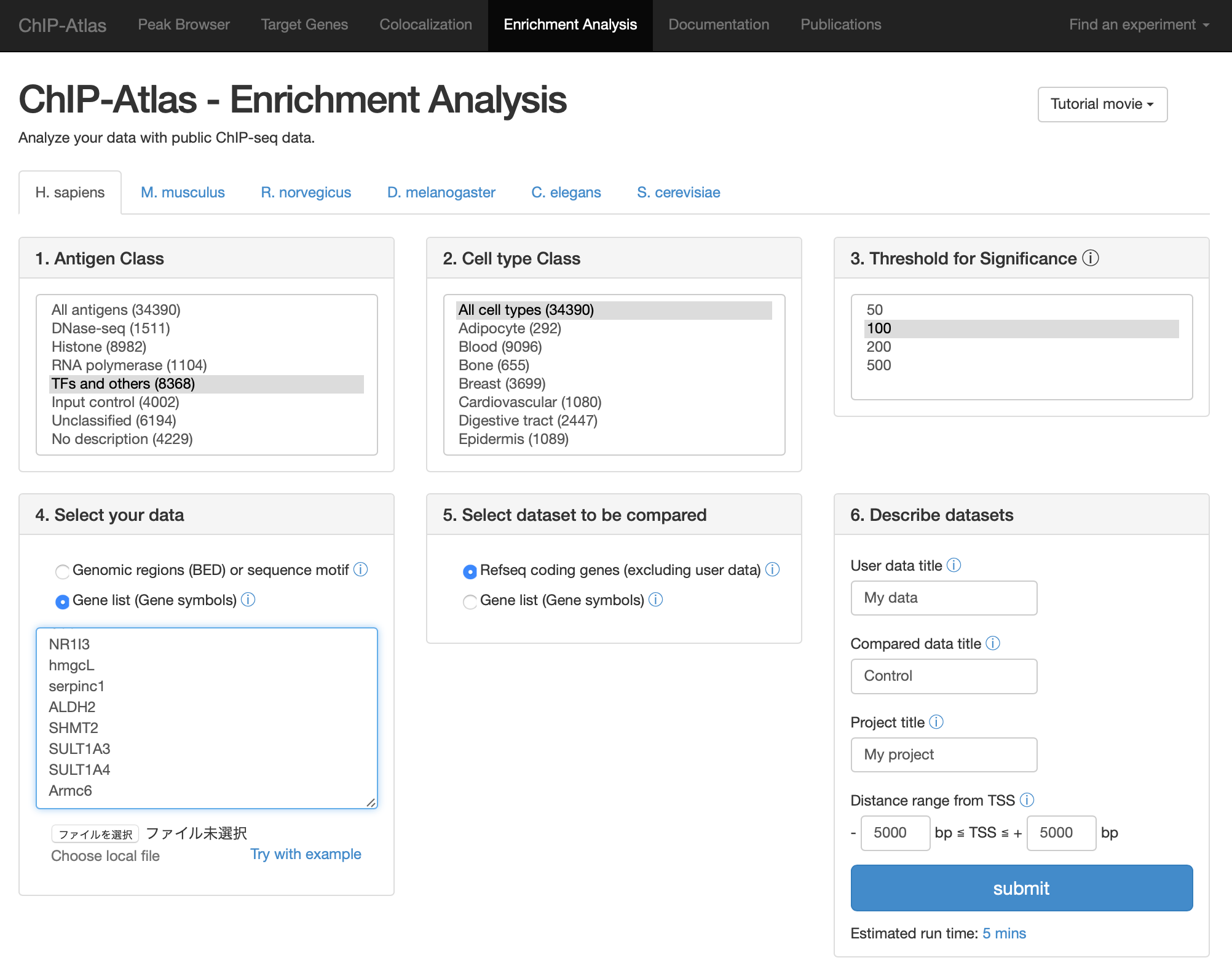
- submit すると遺伝研スパコンへクエリが飛びます。(ので、講義中は見てるだけにしてください)
- submit したあとの画面
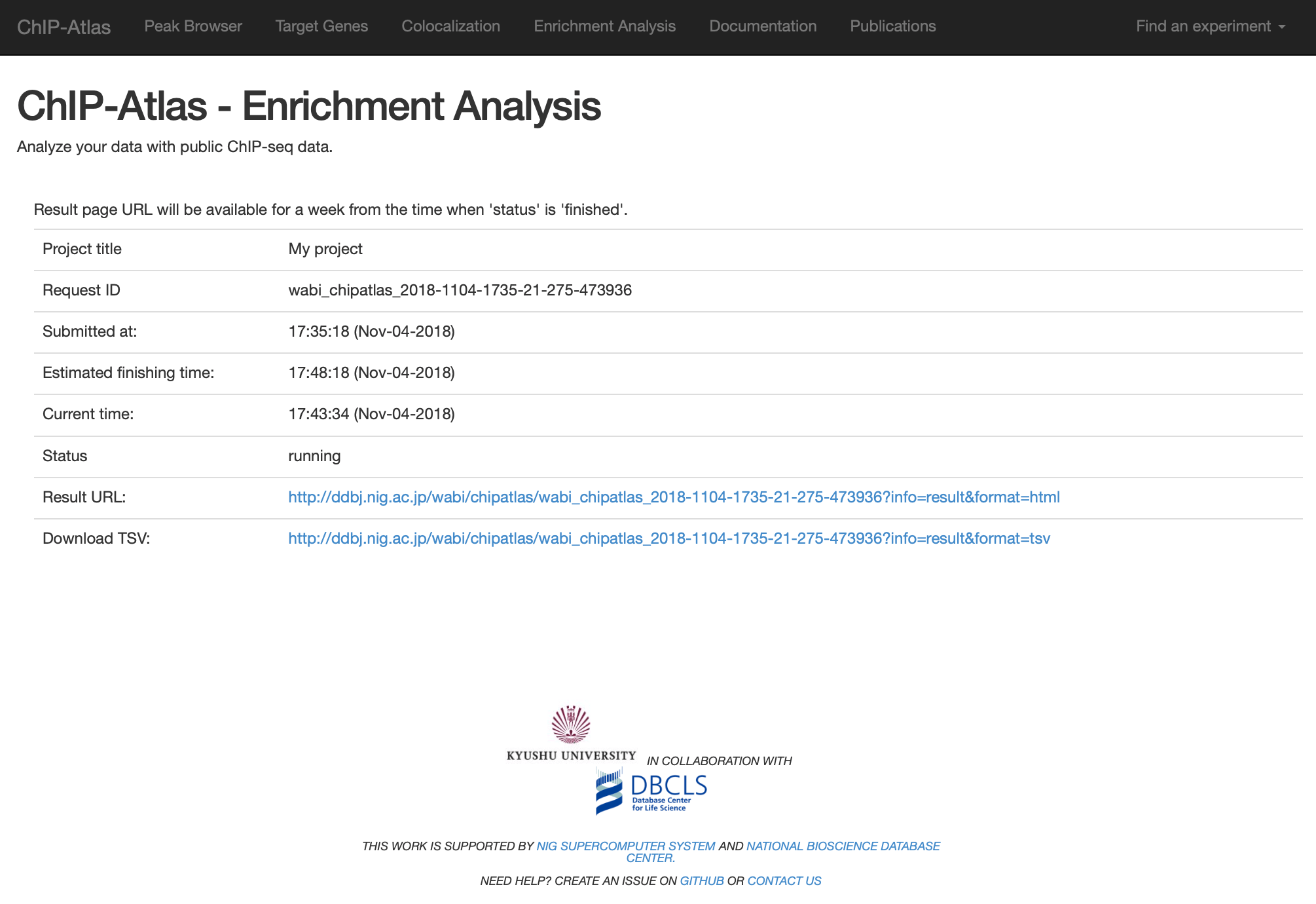
- 計算が終わるまで待ちます
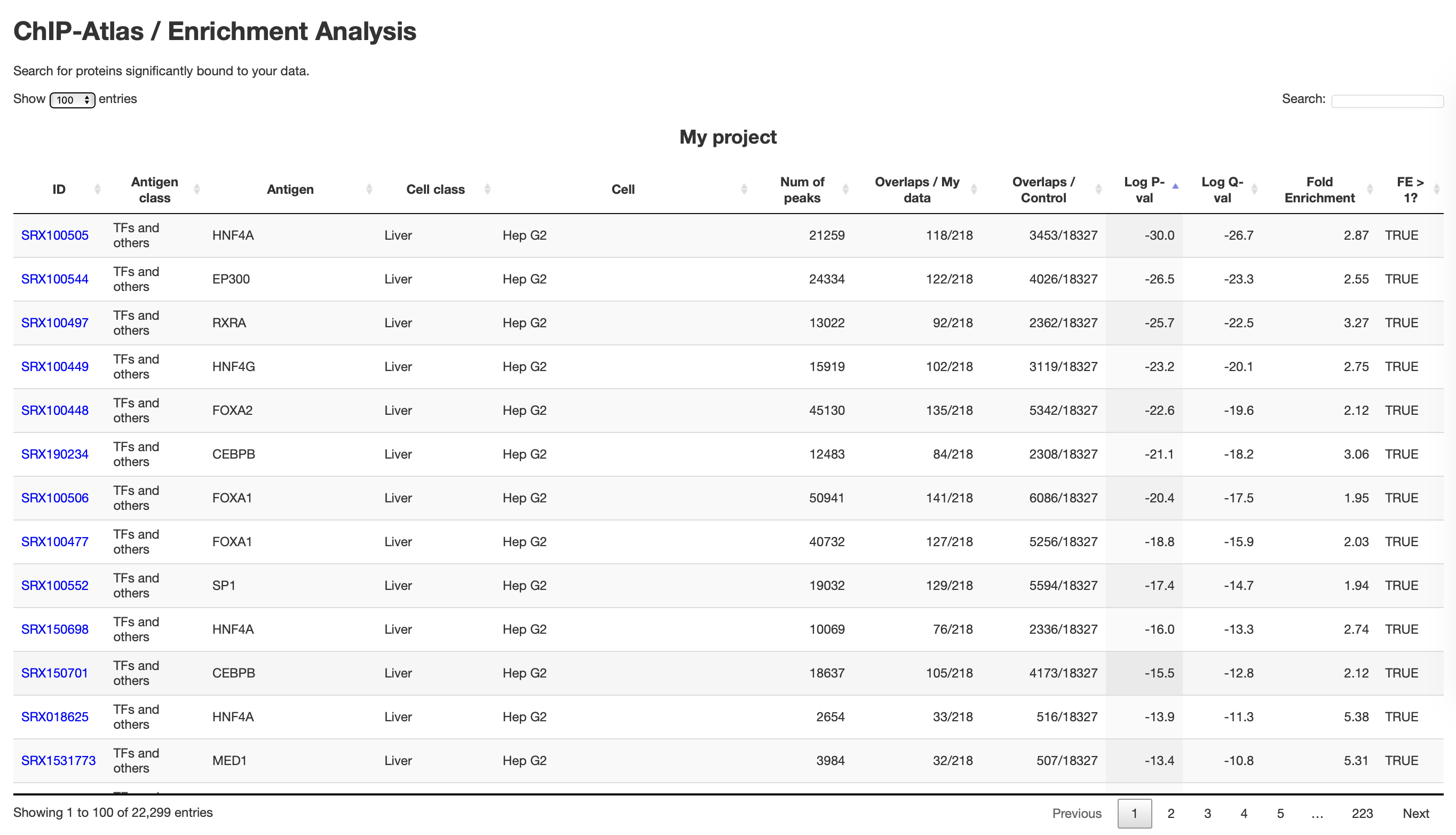
- 計算が終わると、「Result URL」が有効になります。
- 結果の解釈をします。
- 今回は、どういう「興味ある遺伝子リスト」を使ったか、について上記の結果から考察してみましょう。
- 「p-valueが低く、Overlaps/My dataが多く、Fold Enrichmentが高い」転写因子がたくさんヒットしてくる
- → 入力した遺伝子群をまとめて制御する、マスター転写因子を抽出できている可能性が高いと解釈できる。
-
- DAVIDはマイクロアレイ実験から得られたデータを解析するツールです。このツールを使うことで発現変動のあった遺伝子群の特徴を可視化し、直感的に分析することができます。DAVIDという名前はThe Database for Annotation, Visualization and Integrated Discoveryの頭文字に由来しています。
- 【統合TV】DAVIDを使ってマイクロアレイデータを解析する 2012
-
- Metascapeは、ウェブ上でヒト・マウス・ラットのエンリッチメント解析ができるツールです。リストに含まれる遺伝子にどのような機能を持つものが多いか、クラスタリングなどによって直感的に分析することができます。IDリストのほかタイムコースなどの複数リストデータも使用可能で、複数リスト間のアノテーションについて差分を表示することもできます。
- 【統合TV】Metascapeを使って、遺伝子リストの生物学的解釈をする
-
- 【統合TV】Dataset2Tools でオミックスデータとその解析事例、計算ツールを検索し、再現性の高い再解析を行う
- (6:00~) Enrichr による RNA-seq データのエンリッチメント解析
- 特定のモデル生物(ショウジョウバエ、酵母、線虫、ゼブラフィッシュ)に特化したEnrichrもある。
- https://togotv.dbcls.jp/
- 各動画はYouTubeに上がっており、おなじみの再生画面で快適にご覧いただけます。(環境に応じた解像度、倍速表示等)
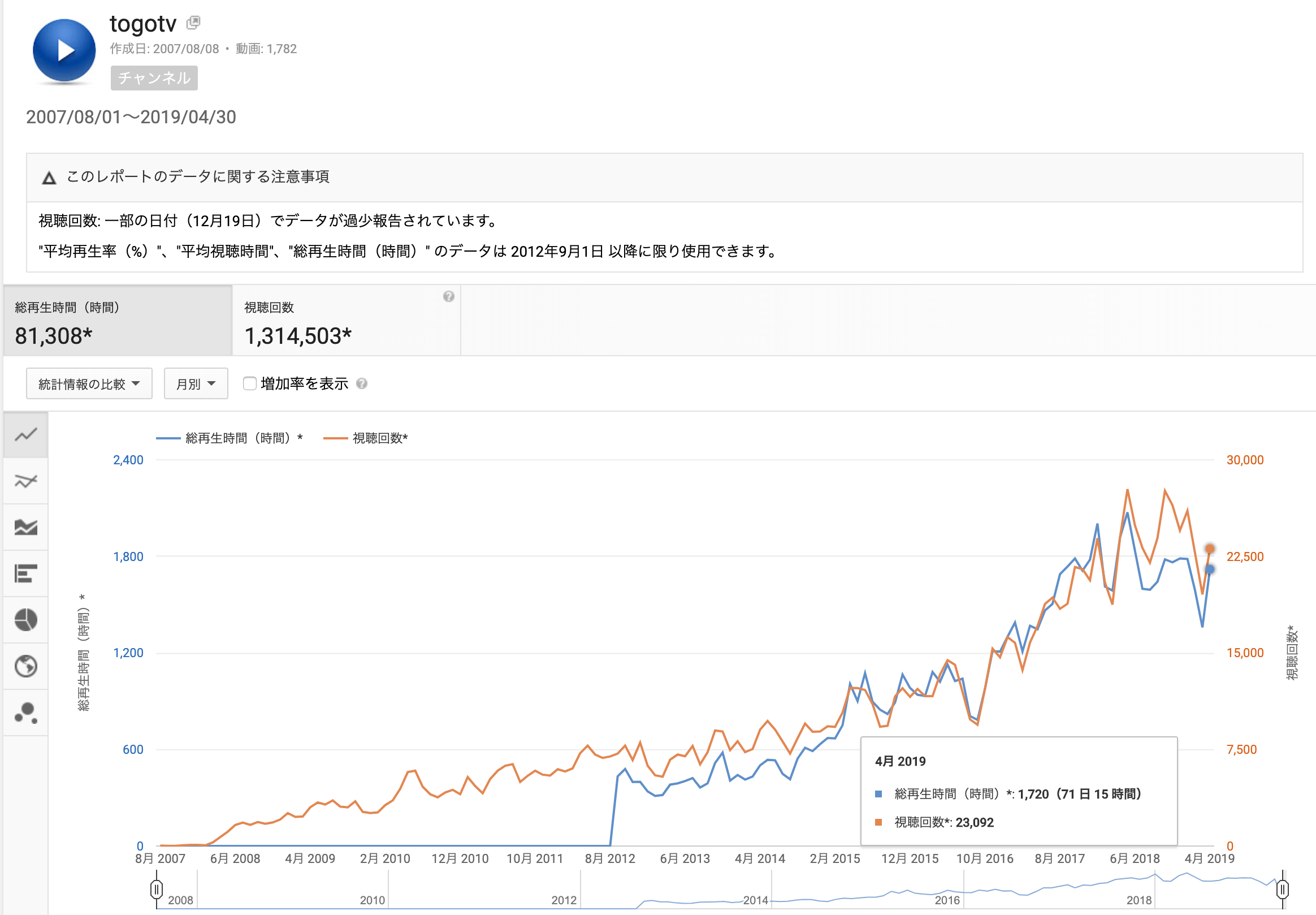
- 1600本を超える動画が公開されており、YouTube版だけで のべ 1,300,000回以上 再生されています。(2019年4月末現在)
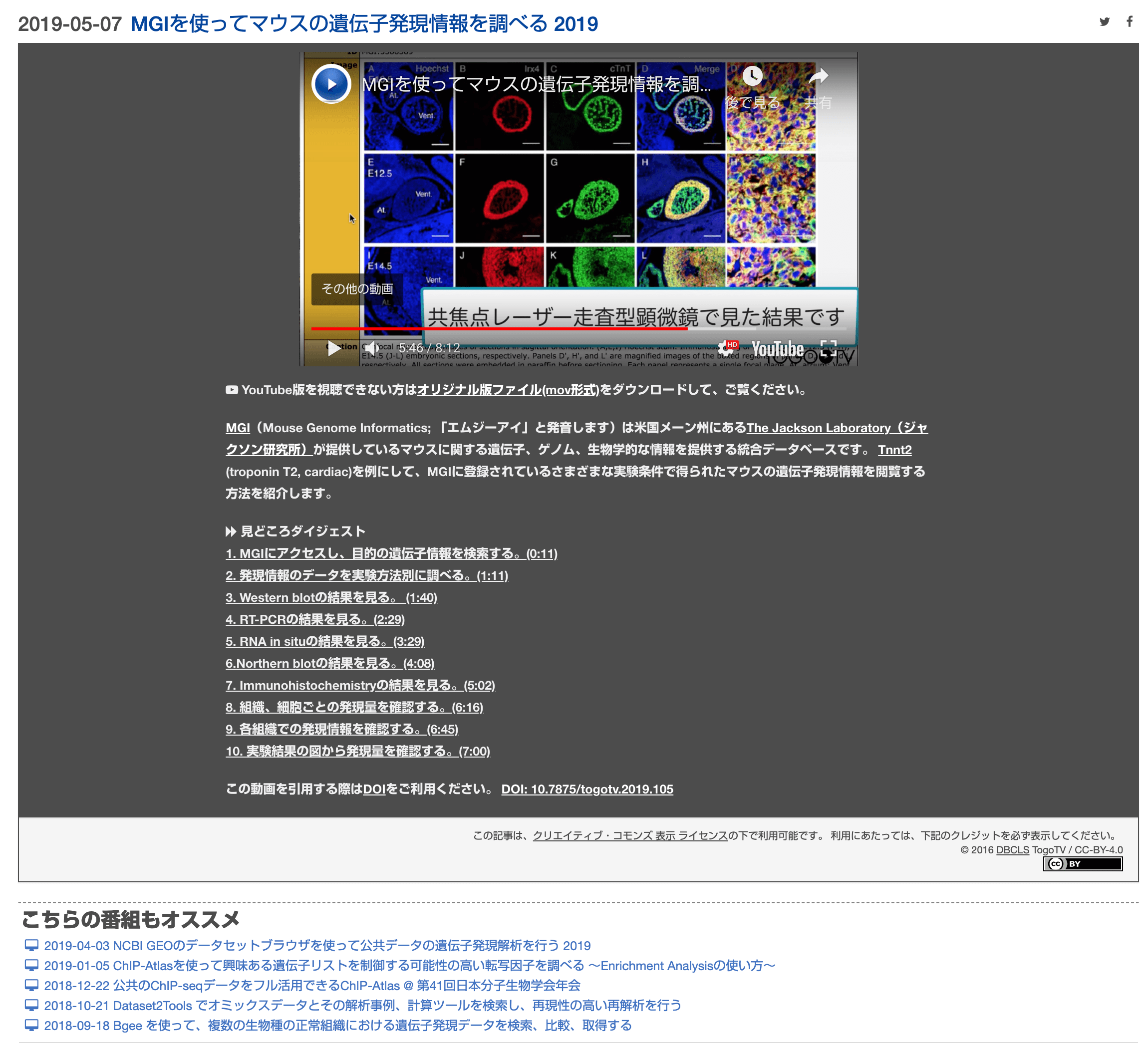
- ウェブサイトへのアクセスの仕方から結果の解釈まで、操作の一挙手一投足がわかります。
- 動画ファイルのみの一部および一括ダウンロード(NBDC生命科学系データベースアーカイブ)も可能で、オフライン視聴もできます。
- 動画の概要を示すダイジェスト見出しで視聴すべき箇所がわかりやすくなっています。
- 各動画には、恒久的な URL として利用されている DOI (Digital Object Identifier) が付与されています(引用可能)。
- 講義・講習などの参考資料や後輩指導の教材として利用できます。
- 本講義中も、本家サイトが繋がらない時は、統合TVを見ればおおよその内容がわかるようになっています。
- AJACS講習会資料
- 2014年8月以降に開催された過去の講習会の資料・テキストと動画が同時閲覧できます。
- 受講生の復習のみならず、初学者の学習教材として活用できます。
- Togo Picture Gallery(静止画)
- 誰でも自由に利用可能なライフサイエンス分野のイラストが、統合TVから閲覧、利用することができるようになりました。「自由に使える画像を探す」
- Togo picture galleryと生物アイコンの全画像500点以上を一覧できます。
- 研究発表のスライド作成や資料作成等に、ぜひお使いください。
- お探しの動画・静止画が見つからない場合は、統合TV番組リクエストフォームでお気軽にリクエストを。
- 統合TVでコンテンツを作ってみたい方も募集中です。
- オンラインで完結する作成環境を整備しており、遠隔地でもOKです。謝金あり。)
- 統合TVを効果的に利用するためのガイドブックが出版されました。(2018年12月)
-
生命科学データベース・ウェブツール - 図解と動画で使い方がわかる! 研究がはかどる定番18選 -
-
「定番」として何がよく使われているのかを知り、その使い方を学び、どう使うと便利なのかについて、体系的にまとめて俯瞰的に捉えられるように編集されています。
-
「この順で動画をみていくと、こういうスキルを獲得できる」というような体系的な教材です。
-
https://www.medsi.co.jp/books/products/detail.php?product_id=3665
-
-
DBCLSの提供する便利な各種サービスをレビュー
-
PubMed検索のプロによる文献検索のイロハを学ぶ
-
塩基配列解析に関わる基礎知識(遺伝子IDとそのデータベース)とゲノム編集について
-
次世代シーケンス(NGS)データ解析に必要な基礎知識とリテラシーを学ぶ
-
NGS解析について、さらにもっと基礎から応用までを深く学びたい方向け (それぞれ約50時間程度)
-
データ可視化・解析ツール
-
ビッグデータ時代の疾患ゲノム解析で使いこなしたいデータベース
- The Cancer Genome Atlas (TCGA) を使って各癌種の公開データを検索・ダウンロードする
- The Cancer Genome Atlas (TCGA) を使って各癌種の公開データを解析する
- COSMICでがん遺伝子の体細胞変異について調べる
- COSMIC-3Dを使って がん遺伝子のコードするタンパク質の立体構造を がんで見られる変異の情報と重ねて見る
- depmapを使ってがん細胞が依存する遺伝子の情報を調べる (がんの治療標的となる遺伝子を発見する)
- GWAS Catalog を使って、GWASで見つかった形質と多型の関連について検索する
- ClinVarで疾患に関連する変異を検索し、Variation Viewerで視覚的に変異を探す
- gnomADを使ってヒトのエキソームやゲノムのデータから変異を探す
- つまみ食い的ではありますが、ゲノムブラウザから始まり遺伝子発現・局在データに関する(ごく一部の)データベース・ウェブツールの使い方を紹介しました。
- データベースやウェブツールは、顕微鏡 や 実験試薬 などと同じ「道具(ツール)」なので使ってナンボです。
- 便利な「道具」を知って、その使い方が分かれば、あとは情報分析力と想像力の勝負です。
- 正面からしか見られなかったものが横や後ろやナナメから見ることができて初めて気づくことがあるかもしれません。
- 仮説構築から始まり、実験計画・検証、データ解析、そして論文執筆(以下ループ)という研究サイクルを加速化・効率化していきましょう。
- データベースやウェブツールで困ったら、「統合TV」でまず探して・見てみる
- 研究に役立ったら、ぜひ引用・クレジットを!
- NBDC/DBCLSの提供するサービス(あるいはそれ以外でも)が、あなたの研究に役立ったら、どんなに些細な事でもぜひ引用(論文、URL等)してください。NBDC/DBCLSの活動は、提供するサービスがどのくらい活用されたかについて主に引用数やアクセス数などで評価されており、利用者の方の積極的なサポートが必要不可欠です!!
- NBDC関連サービスの活用に関する情報提供フォーム