diff --git a/CONDUCT-es-419.md b/CONDUCT-es-419.md
new file mode 100644
index 0000000..37ef03e
--- /dev/null
+++ b/CONDUCT-es-419.md
@@ -0,0 +1,8 @@
+# Código de conducta
+
+Como proyecto entre 2i2c y MetaDocencia, el proyecto ScienceCore:climaterisk está sujeto a las políticas siguientes:
+
+- [Código de conducta de 2i2c](https://compass.2i2c.org/code-of-conduct)
+- [Pautas de Convivencia de MetaDocencia](https://www.metadocencia.org/pdc)
+
+Puede seguirse el procedimiento de notificación de cualquiera de las dos políticas.
diff --git a/CONTRIBUTING-es-419.md b/CONTRIBUTING-es-419.md
new file mode 100644
index 0000000..a627abf
--- /dev/null
+++ b/CONTRIBUTING-es-419.md
@@ -0,0 +1,13 @@
+# Contribuciones
+
+Este repositorio ha sido creado mediante una colaboración entre 2i2c y MetaDocencia. Para hacer un cambio en este repositorio, por favor genera un _pull request_.
+
+Si el cambio es menor(como un arreglo de formato o una corrección gramatical), se invita a quienes proporcionan mantenimiento a auto-mergear el cambio sin revisión (omitiendo las protecciones de la rama).
+
+Si el cambio no es trivial, solicita una revisión a otro miembro del equipo de ScienceCore:climaterisk.
+
+Una vez que el cambio haya sido aprobado, el PR podrá fusionarse. Si el PR proviene de un miembro del equipo de ScienceCore:climaterisk, normalmente se fusionará por sí mismo. Si el RP proviene de alguien externo al equipo ScienceCore:climaterisk, cualquier miembro de ese equipo puede hacer la fusión o merge.
+
+## Código de conducta
+
+Ten en cuenta que este tutorial ScienceCore: Determinación de riesgos con NASA Earthdata Cloud se publica con un [Código de conducta de contribuidores](CONDUCT.md). Al contribuir con este proyecto, te comprometes a cumplir sus términos.
diff --git a/book/es-419/01_Geospatial_Background/02_Working_with_Raster_Data.md b/book/es-419/01_Geospatial_Background/02_Working_with_Raster_Data.md
new file mode 100644
index 0000000..004f0be
--- /dev/null
+++ b/book/es-419/01_Geospatial_Background/02_Working_with_Raster_Data.md
@@ -0,0 +1,136 @@
+---
+jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Cómo trabajar con datos ráster
+
+De la [descripción de Wikipedia sobre las _imágenes ráster_](https://es.wikipedia.org/wiki/Imagen_de_mapa_de_bits):
+
+> En [computación gráfica](https://es.wikipedia.org/wiki/Computaci%C3%B3n_gr%C3%A1fica) y [fotografía digital](https://es.wikipedia.org/wiki/Fotograf%C3%Ada_digital), un gráfico rásterizado representa una imagen bidimensional como una matriz rectangular o rejilla de [píxeles](https://es.wikipedia.org/wiki/P%C3%Adxel), que puede visualizarse mediante una [pantalla de computadora](https://en.wikipedia.org/wiki/Computer_display), [_paper_](https://en.wikipedia.org/wiki/Paper), o cualquier otro medio de visualización.
+
+El término _"dato ráster"_ o _"ráster"_ proviene de los gráficos por computadora. En esencia, un ráster se refiere a una secuencia de valores numéricos dispuestos en una tabla rectangular (similar a una [matriz](https://es.wikipedia.org/wiki/Matriz_\(matem%C3%A1tica\)) de [álgebra lineal](https://es.wikipedia.org/wiki/%C3%81lgebra_lineal)).
+
+Los conjuntos de datos ráster son útiles para representar cantidades continuas, como presión, elevación, temperatura, clasificación de la cobertura terrestre, etc., muestreadas en una [teselación](https://es.wikipedia.org/wiki/Teselado), es decir, una cuadrícula discreta que divide una región bidimensional de extensión finita. En el contexto de los [Sistemas de Información Geográfica (GIS, por las siglas en inglés de, _Geographic Information System_)](https://en.wikipedia.org/wiki/Geographic_information_system), la región espacial es la proyección plana de una porción de la superficie terrestre.
+
+Los rásters se aproximan a las distribuciones numéricas continuas almacenando valores individuales dentro de cada celda de la _cuadrícula_ o _píxel_ (un término derivado del _"elemento de imagen"_ en los gráficos por computadora). Los rásters pueden representar datos recopilados en varios tipos de celdas de cuadrícula no rectangulares (por ejemplo, hexágonos). Para nuestros fines, restringiremos nuestra atención en las cuadrículas en las que todos los píxeles son rectángulos que tienen el mismo ancho y alto.
+
+
+
+En esta imagen se muestra un ejemplo de datos ráster. Fuente: *National Ecological Observatory Network* (NEON)(en español, Red Nacional de Observatorios Ecológicos).
+
+
+---
+
+## Cómo comprender los valores de los ráster
+
+Una diferencia sutil e importante entre una matriz numérica conocida del álgebra lineal y un ráster en el contexto de los SIG es que los conjuntos de datos reales a menudo están _incompletos_. Es decir, un ráster puede carecer de entradas o puede incluir valores de píxeles que se corrompieron durante el proceso de medición. Por eso, la mayoría de los datos ráster incluyen un esquema para representar los valores "Sin datos" de los píxeles en los que no es posible hacer una observación significativa. El esquema puede utilizar "No es un número" (["NaN" , por las siglas en inglés de _Not-a-Number_](https://es.wikipedia.org/wiki/NaN)) para los datos de punto flotante o un [_valor testigo_](https://en.wikipedia.org/wiki/Sentinel_value) para los datos enteros (por ejemplo, utilizando -1 para señalar los datos que faltan cuando los datos enteros significativos son estrictamente positivos).
+
+Otra propiedad importante de los datos ráster es que los valores de los píxeles se almacenan utilizando un _tipo de datos_ apropiado para el contexto. Por ejemplo, las funciones continuas, como la altitud o la temperatura, suelen almacenarse como datos ráster utilizando números de punto flotante. Por el contrario, las propiedades categóricas (como los tipos de cobertura del suelo) pueden almacenarse utilizando números enteros sin signo. Los conjuntos de datos ráster suelen ser grandes, así que elegir el tipo de datos siguiendo un criterio puede reducir significativamente el tamaño del archivo sin comprometer la calidad de la información. Veremos esto con ejemplos más adelante.
+
+---
+
+## Comprensión del Píxel vs. Coordenadas continuas
+
+Cada píxel de un ráster está indexado por su posición en la _fila y columna_ con respecto a la esquina superior izquierda de la _imagen_ o de las _coodenadas del píxel_. Estos valores representan el desplazamiento desde la esquina superior izquierda de la matriz, expresado convencionalmente utilizando una [_indexación desde cero_](https://en.wikipedia.org/wiki/Zero-based_numbering) (en inglés, _zero based numbering_). Por ejemplo, si se utiliza la indexación desde cero en la cuadrícula de píxeles $10\tveces10$ que se muestra a continuación, el píxel de la esquina superior izquierda tendrá el índice (0,0), el píxel de la esquina superior derecha tendrá el índice (0,9), el píxel de la esquina inferior izquierda tendrá el índice (9,0), y el píxel de la esquina inferior derecha tendrá el índice (9,9).
+
+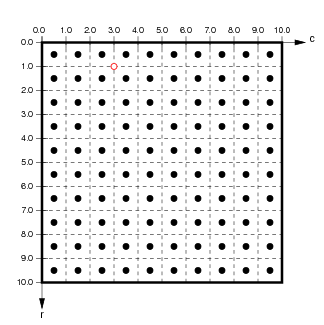
+
+(de `http://ioam.github.io/topographica`)
+
+La indexación desde cero no se observa universalmente (por ejemplo, MATLAB utiliza la indexación basada en matrices y arreglos), así que debemos ser conscientes de cuál convención se utiliza en cualquier herramienta utilizada. Sin importar si vamos a contar filas/columnas desde uno o desde cero, cuando se utilizan coordenadas de píxel/imagen/matriz, convencionalmente, el índice de la fila aumenta desde la fila superior y aumenta al desplazarse hacia abajo.
+
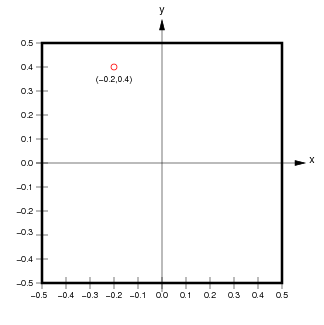
+Otra sutil distinción entre matrices y rásters es que los datos ráster suelen estar _georreferenciados_, esto significa que cada celda está asociada a un rectángulo geográfico concreto de la superficie terrestre con cierta área positiva. Esto a su vez significa que cada valor del píxel está asociado no solo con sus coordenadas de píxel/imagen, sino también con las coordenadas continuas de cada punto espacial dentro de ese rectángulo físico. Es decir, hay un _continuo_ de coordenadas espaciales asociadas a cada píxel en contraposición a un único par de coordenadas del píxel. Por ejemplo, en la cuadrícula de píxeles $5\times5$ que se muestra a continuación en el gráfico de la izquierda, el punto rojo asociado a las coordenadas del píxel $(0,1)$ se encuentra en el centro de ese píxel. Al mismo tiempo, el gráfico de la derecha muestra un punto rojo con coordenadas continuas $(-0,2,0,4)$.
+
+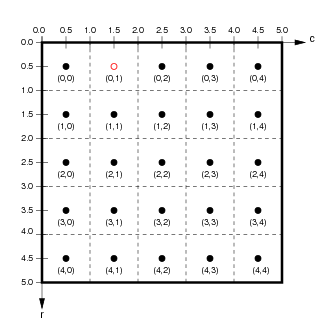
+
+
+(from `http://ioam.github.io/topographica`)
+
+Hay dos distinciones importantes que se deben observar:
+
+- Las coordenadas de imagen se expresan normalmente en orden inverso a las coordenadas continuas. Es decir, para las coordenadas de imagen $(f,c)$, la posición vertical, la fila $f$, es la primera ordenada, y la posición horizontal, la columna $c$, es la segunda ordenada. Por el contrario, cuando se expresa una posición en coordenadas continuas $(x,y)$, convencionalmente, la posición horizontal $x$ es la primera ordenada y la posición vertical $y$ es la segunda ordenada.
+- La orientación del eje vertical se invierte entre coordenadas de píxel y coordenadas continuas (aunque la orientación del eje horizontal es la misma). Es decir, el índice de fila $f$ aumenta _hacia abajo_ desde la esquina superior izquierda en coordenadas del píxel, mientras que la coordenada vertical continua $y$ aumenta hacia arriba desde la esquina inferior izquierda.
+
+Las convenciones contradictorias con la indexación basada en cero y el orden de las coordenadas son fuente de muchos problemas en programación. Por ejemplo, en la práctica, algunas herramientas SIG exigen que las coordenadas se proporcionen como `(longitud, latitud)` y otras como `(latitud, longitud)`. Con suerte, los usuarios de SIG pueden confiar en que las herramientas de _software_ manejen estas inconsistencias de forma transparente (consulta, por ejemplo, [esta discusión en la documentación de Holoviz](https://holoviews.org/user_guide/Continuous_Coordinates.html)). Cuando los resultados calculados no tienen sentido, lo importante es preguntar siempre:
+
+- qué convenciones se utilizan para representar los datos ráster que se recuperaron de una fuente determinada, y
+- qué convenciones requiere cualquier herramienta SIG se utiliza para manipular datos ráster.
+
+---
+
+## Conservación de los metadatos
+
+Los datos ráster usualmente van acompañados de una variedad de _metadatos_. Estos pueden incluir:
+
+- el _Sistema de Referencia de Coordenadas (SRC)(en inglés, _Coordinate Reference System_, CRS)_: las posibles representaciones incluyen el [_registro EPSG_](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset), [_WKT_](https://en.wikipedia.org/wiki/Well-known_text_representation_of_coordinate_reference_systems), etc.
+- el valor _NoData_: el/los valor(es) testigo/s que señalan datos ausentes/corruptos (por ejemplo, `-1` para datos enteros o `255` para datos enteros de 8 bits sin signo, etc.).
+- la _resolución espacial_: el área (en unidades adecuadas) de cada píxel.
+- los _límites_: es la extensión del rectángulo espacial georreferenciado por estos datos ráster.
+- la _fecha y hora de adquisición_: la fecha de adquisición de los datos, que a menudo se especifica mediante el [_Tiempo Universal Coordinada_](https://es.wikipedia.org/wiki/Tiempo_universal_coordinado) (UTC, por sus siglas en iglés de _Coordinated Universal Time_).
+
+Los distintos formatos de archivo utilizan diferentes estrategias para adjuntar metadatos a un determinado conjunto de datos ráster. Por ejemplo, los productos de datos NASA OPERA generalmente tienen nombres de archivo como:
+
+```
+OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif
+```
+
+Este nombre incluye una marca de tiempo (`20220815T185931Z`) y [una ubicación geográfica MGRS](https://en.wikipedia.org/wiki/Military_Grid_Reference_System) (`10TEM`). Este tipo de convenciones para la denominación de los archivos permite determinar los atributos de los datos ráster sin necesidad de recuperar todo el conjunto de datos, lo que puede reducir significativamente los costos de transferencia de los datos.
+
+---
+
+## Uso de GeoTIFF
+
+Hay numerosos formatos de archivo estándar que se utilizan para compartir muchos tipos de datos científicos (por ejemplo, [_HDF_](https://en.wikipedia.org/wiki/Hierarchical_Data_Format), [_Parquet_](https://parquet.apache.org/), [_CSV_](https://es.wikipedia.org/wiki/Valores_separados_por_comas), etc.). Además, hay [docenas de formatos de archivo especializados](https://www.spatialpost.com/list-common-gis-file-format/) para los [\*(_Sistemas de Información Geográfica (GIS)_](https://en.wikipedia.org/wiki/Geographic_information_system), por ejemplo, DRG, [_NetCDF_](https://docs.unidata.ucar.edu/nug/current/), USGS DEM, DXF, DWG, SVG, etc. En este tutorial, nos enfocaremos exclusivamente en el uso del formato de archivo _GeoTIFF_ para representar los datos ráster.
+
+[GeoTIFF](https://es.wikipedia.org/wiki/GeoTIFF) es un estándar de metadatos de dominio público diseñado para trabajar con archivos [Formato de archivo de imagen etiquetado](https://en.wikipedia.org/wiki/TIFF) ( (TIFF, por sus siglas en inglés de _Tagged Image File Format_))). El formato GeoTIFF permite incluir información de georreferenciación como metadatos geoespaciales dentro de los archivos de imagen. Las aplicaciones SIG utilizan GeoTIFF para fotografías aéreas, imágenes de satélite y mapas digitalizados. El formato de los datos GeoTIFF se describe detalladamente en el documento [estándar OGC GeoTIFF](https://www.ogc.org/standard/geotiff/).
+
+Un archivo GeoTIFF usualmente incluye metadatos geográficos como etiquetas embebidas. Estos pueden incluir metadatos de imágenes ráster tales como:
+
+- extensión espacial, es decir, el área que cubre el conjunto de datos,
+- el SRC qué se utiliza para almacenar los datos,
+- la resolución espacial, es decir, las dimensiones horizontal y vertical de los píxeles,
+- el número de valores del píxel en cada dirección,
+- el número de capas del archivo `.tif`,
+- elipsoides y geoides (es decir, modelos estimados de la forma de la Tierra), y
+- reglas matemáticas de proyección cartográfica para transformar los datos de un espacio tridimensional en una visualización bidimensional.
+
+A modo de ejemplo, carguemos los datos de un archivo GeoTIFF local utilizando el paquete `rioxarray` de Python.
+
+```{code-cell} python
+from pathlib import Path
+import rioxarray as rio
+
+LOCAL_PATH = Path.cwd().parent / 'assets' / 'OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif'
+```
+
+```{code-cell} python
+%%time
+da = rio.open_rasterio(LOCAL_PATH)
+```
+
+La función `rioxarray.open_rasterio` cargó los datos raster del archivo GeoTIFF local en un Xarray `DataArray` llamado `da`. Podemos analizar cómodamente el contenido de `da` en un cuaderno computacional Jupyter.
+
+```{code-cell} python
+da # examine contents
+```
+
+Este ráster es de alta resolución ($3600\times3600$ píxeles). Vamos a tomar una parte más pequeña (por ejemplo, un muestreo cada 200 píxeles) creando una instancia del objeto `slice` de Python `subset` y usando el método Xarray `DataArray.isel` para construir una matriz de menor resolución (que se renderizará más rápido). Entonces podemos hacer una visualización (renderizado por Matplotlib de manera predeterminada).
+
+```{code-cell} python
+subset = slice(0,None,200)
+view = da.isel(x=subset, y=subset)
+view.plot();
+```
+
+Observe que la visualización se etiqueta utilizando las coordenadas continuas (este, norte) asociadas a la extensión espacial de este ráster. Es decir, la sutil contabilidad necesaria para hacer un seguimiento de las coordenadas continuas y de píxel fue administrada de forma transparente por la API de la estructura de datos. ¡Eso es bueno!
+
+---
diff --git a/book/es-419/01_Geospatial_Background/03_Working_with_Vector_Data.md b/book/es-419/01_Geospatial_Background/03_Working_with_Vector_Data.md
new file mode 100644
index 0000000..bb7522d
--- /dev/null
+++ b/book/es-419/01_Geospatial_Background/03_Working_with_Vector_Data.md
@@ -0,0 +1,167 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Cómo trabajar con datos vectoriales
+
+Ciertas propiedades físicas de interés para los Sistemas de Informacion Geográfica (SIG) (en inglés, _Geographical Information System, GIS_), no se capturan convenientemente mediante datos ráster en cuadrículas discretas. Por ejemplo, las características geométricas de un paisaje, como carreteras, ríos y límites entre regiones, se describen mejor utilizando líneas o curvas en un sistema de coordenadas proyectado de manera adecuada. En el contexto de los GIS, los _datos vectoriales_ son representaciones geométricas de estos aspectos del paisaje.
+
+---
+
+## Cómo comprender de los Datos Vectoriales
+
+Los _datos vectoriales_ consisten en secuencias ordenadas de _vértices_, es decir, pares de números de la forma $(x,y)$. Las coordenadas continuas de cada vértice se asocian a una ubicación espacial física en algún Sistema de Referencia de Coordenadas (SRC) (en inglés, _Coordinate Reference System, CRS_) proyectado.
+
+- _Puntos_: Los vértices aislados representan funciones discretas de dimensión cero (por ejemplo, árboles, faroles, etc.).
+- _Líneas_ o _polilíneas_: Cualquier secuencia ordenada de por lo menos dos vértices constituye una _polilínea_ que puede visualizarse trazando líneas rectas entre vértices adyacentes. Las líneas o polilíneas son adecuadas para representar características unidimensionales como carreteras, caminos y ríos.
+- _Polígonos_: Cualquier secuencia ordenada de por lo menos tres vértices en la que el primero y el último coinciden constituye un _polígono_ (es decir, una forma cerrada). Los polígonos son adecuados para representar regiones bidimensionales como un lago o el límite de un bosque. Un uso común de los polígonos es representar las fronteras entre circunscripciones políticas (por ejemplo, países).
+
+Un conjunto único de datos vectoriales georreferenciados con un CRS en particular, generalmente contiene cualquier número de puntos, líneas o polígonos.
+
+
+
+Esta imagen muestra los tres tipos de datos vectoriales: puntos, líneas y polígonos. Fuente: *National Ecological Observatory Network* (NEON) (en español, Red Nacional de Observatorios Ecológicos).
+
+
+
+
+(Imágen de la izquierda) Vista aérea de un paisaje, (imágen de la derecha) representaciones vectoriales de puntos de características destacadas.
Fuente: *Sistemas de Información Geográfica* por Victor Olaya.
+
+
+
+
+(Imágen de la izquierda) Vista aérea de un río, (imágen de la derecha) representación vectorial de líneas del río.
Fuente: *Sistemas de Información Geográfica* por Victor Olaya.
+
+
+
+
+(Imagen de la izquierda) Vista aérea de un lago, (imagen de la derecha) representación vectorial poligonal del lago.
Fuente: *Sistemas de Información Geográfica* por Victor Olaya.
+
+
+Como ocurre con los datos ráster, generalmente las representaciones de datos vectoriales van acompañadas de metadatos para almacenar diversos atributos asociados al conjunto de datos. Los datos vectoriales usualmente se especifican con un nivel de precisión superior a la resolución que permiten la mayoría de las cuadrículas ráster. Además, las características geográficas representadas como datos vectoriales permiten efectuar cálculos que los datos ráster no permiten. Por ejemplo, es posible determinar diversas relaciones geométricas o topológicas:
+
+- ¿Un punto se encuentra dentro de los límites de una región geográfica?
+- ¿Dos carreteras se intersecan?
+- ¿Cuál es el punto más cercano de una carretera a otra región?
+
+Otras magnitudes geométricas, como la longitud de un río o la superficie de un lago, se obtienen aplicando técnicas de geometría computacional a representaciones de datos vectoriales.
+
+---
+
+## Uso de GeoJSON
+
+[GeoJSON](https://geojson.org/) es un subconjunto de [notación de objeto de JavaScript, (JSON, por las siglas en inglés de _JavaScript object notation_)](https://www.json.org). Fue desarrollado a principios de la década del 2000 para representar características geográficas simples junto con atributos no espaciales. El objetivo principal es ofrecer una especificación para la codificación de datos geoespaciales que sean decodificables por cualquier decodificador JSON.
+
+Los desarrolladores SIG responsables por GeoJSON lo diseñaron con la intención de que cualquier desarrollador web pudiera escribir un _parser_ GeoJSON personalizado, lo que permitiría muchas formas posibles de utilizar los datos geoespaciales más allá del software GIS estándar. Los detalles técnicos del formato GeoJSON se describen en el documento de estándares [RFC7946](https://datatracker.ietf.org/doc/html/rfc7946).
+
+Veamos cómo analizar y graficar archivos GeoJSON utilizando la librería [GeoPandas](https://geopandas.org/en/stable/). El archivo local `cables.geojson` almacena datos vectoriales de líneas que representan cables submarinos conectando diferentes masas de tierra.
+
+```{code-cell} python
+import geopandas as gpd
+from pathlib import Path
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+GEOJSON = Path(FILE_STEM, 'assets/cables.geojson')
+print(GEOJSON)
+```
+
+```{code-cell} python
+with open(GEOJSON) as f:
+ text = f.read()
+print(text[:1500])
+```
+
+Intentar leer la salida GeoJSON anterior es complicado pero predecible. Los archivos JSON están pensados para ser consumidos por máquinas y, por tanto, no son muy legibles. Los archivos JSON están pensados genéricamente para ser consumidos por máquinas y, por lo tanto, no son muy legibles.
+
+Utilicemos la función `geopandas.read_file` para cargar los datos vectoriales en un `GeoDataFrame`.
+
+```{code-cell} python
+gdf = gpd.read_file(GEOJSON)
+display(gdf.head())
+gdf.info()
+```
+
+Hay 530 filas, cada una de las cuales corresponde a datos de línea (una secuencia conectada de segmentos de línea). Podemos utilizar el atributo `color` de la columna `GeoDataFrame` como una opción dentro de la llamada a `.plot` para graficar estos cables.
+
+```{code-cell} python
+gdf.geometry.plot(color=gdf.color, alpha=0.25);
+```
+
+Usemos un archivo remoto para crear otro `GeoDataFrame`, esta vez que contenga datos de polígonos.
+
+```{code-cell} python
+URL = "http://d2ad6b4ur7yvpq.cloudfront.net/naturalearth-3.3.0/ne_110m_land.geojson"
+gdf = gpd.read_file(URL)
+
+gdf
+```
+
+Esta vez, el gráfico mostrará polígonos rellenos correspondientes a los países del mundo.
+
+```{code-cell} python
+gdf.plot(color='green', alpha=0.25) ;
+```
+
+El método `GeoDataFrame.loc` nos permite graficar subconjuntos concretos de países.
+
+```{code-cell} python
+gdf.loc[15:90].plot(color='green', alpha=0.25) ;
+```
+
+---
+
+## Uso de archivos Shapefiles
+
+El estándar [_shapefile_](https://es.wikipedia.org/wiki/Shapefile) es un formato digital para distribuir datos vectoriales geoespaciales y sus atributos asociados. El estándar, desarrollado por [ESRI](https://es.wikipedia.org/wiki/Esri) hace unos 30 años, es compatible con la mayoría de las herramientas de _software_ SIG modernas. El nombre "_shapefile_" es un poco engañoso porque este tipo de archivo normalmente consiste en un conjunto de varios archivos (algunos obligatorios, otros opcionales) almacenados en un directorio común con un nombre de archivo común.
+
+De [Wikipedia](https://es.wikipedia.org/wiki/Shapefile):
+
+> El formato shapefile almacena la geometría como formas geométricas primitivas como puntos, líneas y polígonos. Estas formas, junto con los atributos de datos vinculados a cada una de ellas, crean la representación de los datos geográficos. El término "shapefile" es bastante común, pero el formato consiste en una colección de archivos con un prefijo de nombre de archivo común, almacenada en el mismo directorio. Los tres archivos obligatorios tienen las extensiones de archivo .shp, .shx y .dbf. El _shapefile_ real se refiere específicamente al archivo .shp, pero por sí solo está incompleto para su distribución, ya que se requieren los demás archivos de apoyo. El _software_ SIG heredado espera que el prefijo del nombre de archivo se limite a ocho caracteres para ajustarse a la convención de nombres de archivo DOS 8.3, aunque las aplicaciones de _software_ modernas aceptan archivos con nombres más largos.
+
+Los _shapefiles_ utilizan el formato [_Binario Bien Conocido_ (WKB, por las siglas en inglés de, _Well-known Binary_)(https://libgeos.org/specifications/wkb/) para codificar las geometrías. Se trata de un formato tabular compacto, es decir, los números de la fila y la columna asumen un valor significativo. Algunas limitaciones menores de este formato incluyen la restricción de los nombres en los campos de los atributos a 10 caracteres o menos y la escasa compatibilidad con Unicode. Como resultado, el texto suele abreviarse y codificarse en ASCII.
+
+Puedes consultar el [Informe técnico sobre _Shapefile_ de ESRI](https://www.esri.com/content/dam/esrisites/sitecore-archive/Files/Pdfs/library/whitepapers/pdfs/shapefile.pdf) para obtener más información sobre los _shapefiles_.
+
+#### Archivos obligatorios
+
+- Archivo principal (`.shp`): formato shape, por ejemplo, los datos vectoriales espaciales (puntos, líneas y polígonos) que representan la geometría del objeto.
+- Archivo de índice (`.shx`): posiciones de índice de formas (para permitir la recuperación de la geometría de los atributos).
+- Archivo dBASE (`.dbf`): archivo de base de datos estándar que almacena el formato de atributos (atributos en columnas para cada tipo en formato dBase IV, normalmente legible por, por ejemplo, Microsoft Access o Excel).
+
+Los registros corresponden en secuencia en cada uno de estos archivos, es decir, los atributos del registro $k$ del archivo `dbf` describen la característica del registro $k$ del archivo `shp` asociado.
+
+#### Archivos opcionales
+
+- Archivo de proyección (`.prj`): descripción del sistema de referencia de coordenadas correspondiente utilizando una [Representación en texto bien conocido de los sistemas de referencia de coordenadas _Well-known text representation of coordinate reference systems_ (WKT o WKT-CRS) (en español, representación en texto conocido de los sistemas de referencia de coordenadas )](https://en.wikipedia.org/wiki/Well-known_text_representation_of_coordinate_reference_systems).
+- Archivo _Extensible Markup Language_ (en español, lenguaje de marcado extensible) (`.xml`): [metadatos geoespaciales](https://en.wikipedia.org/wiki/Geospatial_metadata) en formato [XML](https://es.wikipedia.org/wiki/Extensible_Markup_Language).
+- Archivo de página de códigos (`.cpg`): archivos de texto sin formato para describir la codificación aplicada para crear el _shapefile_. Si tu _shapefile_ no tiene un archivo `.cpg`, entonces utiliza la codificación predeterminada del sistema.
+- Archivos de índice espacial (`.sbn` y `.sbx`): archivos de índice codificados para acelerar los tiempos de carga.
+
+Hay muchos archivos opcionales más. Consulta el [libro blanco](https://www.esri.com/content/dam/esrisites/sitecore-archive/Files/Pdfs/library/whitepapers/pdfs/shapefile.pdf) (en inglés, _whitepaper_).
+
+Al igual que los archivos GeoJSON, _los shapefiles_ se pueden leer directamente utilizando `geopandas.read_file` para cargar el archivo `.shp`. Lo haremos ahora utilizando un _shapefile_ de ejemplo que muestra la delimitación del área de un incendio forestal.
+
+```{code-cell} python
+SHAPEFILE = Path(FILE_STEM, 'assets/shapefiles/mckinney/McKinney_NIFC.shp')
+gdf = gpd.read_file(SHAPEFILE)
+gdf.info()
+gdf.head()
+```
+
+```{code-cell} python
+gdf.plot(color='red', alpha=0.5);
+```
+
+Volveremos a utilizar este _shapefile_ en cuadernos computacioales posteriores y explicaremos con más detalle lo que representa.
+
+---
diff --git a/book/es-419/02_Software_Tools_Techniques/01_Loading_Raster_Data_from_GeoTIFF_Files.md b/book/es-419/02_Software_Tools_Techniques/01_Loading_Raster_Data_from_GeoTIFF_Files.md
new file mode 100644
index 0000000..31ecea2
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/01_Loading_Raster_Data_from_GeoTIFF_Files.md
@@ -0,0 +1,343 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Carga de datos ráster desde archivos GeoTIFF
+
+
+
+Dado que la mayoría de los datos geoespaciales con los que trabajaremos en este tutorial están almacenados en archivos GeoTIFF, debemos saber cómo trabajar con esos archivos. La solución más sencilla es utilizar [rioxarray](https://corteva.github.io/rioxarray/html/index.html). Esta solución se encarga de muchos detalles complicados de forma transparente. También podemos utilizar [Rasterio](https://rasterio.readthedocs.io/en/stable) como herramienta para leer datos o metadatos de archivos GeoTIFF. Un uso adecuado de Rasterio puede marcar una gran diferencia a la hora de trabajar con archivos remotos en la nube.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import numpy as np
+import rasterio
+import rioxarray as rio
+from pathlib import Path
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+***
+
+## [rioxarray](https://corteva.github.io/rioxarray/html/index.html)
+
+
+
+`rioxarray` es un paquete que _extiende_ el paquete Xarray (hablaremos al respecto más adelante). Las principales funciones de `rioxarray` que utilizaremos en este tutorial son:
+
+- `rioxarray.open_rasterio` para cargar archivos GeoTIFF directamente en estructuras Xarray `DataArray`, y
+- `xarray.DataArray.rio` para proporcionar usos útiles (por ejemplo, para especificar información CRS).
+
+Para acostumbrarnos a trabajar con archivos GeoTIFF, utilizaremos algunos ejemplos específicos en este cuaderno computacional y en otros posteriores. Más adelante explicaremos qué tipo de datos contiene el archivo, por el momento, solo queremos acostumbrarnos a cargar datos.
+
+
+
+### Carga de archivos en un DataArray
+
+
+
+Observa en primer lugar que `open_rasterio` funciona con direcciones de archivos locales y URL remotas.
+
+- Como era de esperarse, el acceso local es más rápido que el remoto.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+REMOTE_URL ='https://opera-provisional-products.s3.us-west-2.amazonaws.com/DIST/DIST_HLS/WG/DIST-ALERT/McKinney_Wildfire/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif'
+data_remote = rio.open_rasterio(REMOTE_URL)
+```
+
+
+
+La siguiente operación compara elementos de un Xarray `DataArray` elemento a elemento (el uso del método `.all` es similar a lo que haríamos para comparar arrays NumPy). Por lo general, esta no es una forma recomendable de comparar matrices, series, dataframes u otras estructuras de datos grandes que contengan datos de punto flotante. Sin embargo, en este caso concreto, como las dos estructuras de datos se leyeron del mismo archivo almacenado en dos ubicaciones diferentes, la comparación elemento a elemento tiene sentido. Confirma que los datos cargados en la memoria desde dos fuentes distintas son idénticos en cada bit.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+(data_remote == data).all() # Verify that the data is identical from both sources
+```
+
+***
+
+## [rasterio](https://rasterio.readthedocs.io/en/stable)
+
+
+
+Esta sección puede omitirse si `rioxarray` funciona adecuadamente para nuestros análisis, es decir, si la carga de datos en la memoria no es prohibitiva. Cuando _no_ sea el caso, `rasterio` proporciona estrategias alternativas para explorar los archivos GeoTIFF. Es decir, el paquete `rasterio` ofrece formas de bajo nivel para cargar datos que `rioxarray` cuando sea necesario.
+
+De la [documentación de Rasterio](https://rasterio.readthedocs.io/en/stable):
+
+> Antes de Rasterio había una opción en Python para acceder a los diferentes tipos de archivos de datos ráster utilizados en el campo de los SIG: los enlaces de Python distribuidos con la [Biblioteca de Abstracción de Datos Geoespaciales](http://gdal.org/) (GDAL, por sus siglas en inglés de _Geospatial Data Abstraction Library_). Estos enlaces extienden Python, pero proporcionan poca abstracción para la Interface de programación de aplicaciones C (API C, por sus siglas en inglés de _Application Programming Interface_) de GDAL. Esto significa que los programas Python que los utilizan tienden a leerse y ejecutarse como programas de C. Por ejemplo, los enlaces a Python de GDAL obligan a los usuarios a tener cuidado con los punteros de C incorrectos, que pueden bloquear los programas. Esto es malo: entre otras consideraciones hemos elegido Python en vez de C para evitar problemas con los punteros.
+>
+> ¿Cómo sería tener una abstracción de datos geoespaciales en la biblioteca estándar de Python? ¿Una que utilizara características y modismos modernos del lenguaje Python? ¿Una que liberara a los usuarios de la preocupación por los punteros incorrectos y otras trampas de la programación en C? El objetivo de Rasterio es ser este tipo de biblioteca de datos ráster, que exprese el modelo de datos de GDAL utilizando menos clases de extensión no idiomáticas y tipos y protocolos de Python más idiomáticos, a la vez que funciona tan rápido como los enlaces de Python de GDAL.
+>
+> Alto rendimiento, menor carga cognitiva, código más limpio y transparente. Eso es Rasterio.
+
+
+
+***
+
+### Abrir archivos con rasterio.open
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Show rasterio.open works using context manager
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+print(LOCAL_PATH)
+```
+
+
+
+Dada una fuente de datos (por ejemplo, un archivo GeoTIFF en el contexto actual), podemos abrir un objeto `DatasetReader` asociado utilizando `rasterio.open`. Técnicamente, debemos recordar cerrar el objeto después. Es decir, nuestro código quedaría así:
+
+```{code-cell} python
+ds = rasterio.open(LOCAL_PATH)
+# ..
+# do some computation
+# ...
+ds.close()
+```
+
+Al igual que con el manejo de archivos en Python, podemos utilizar un _administrador de contexto_ (es decir, una cláusula `with`) en su lugar.
+
+```python
+with rasterio.open(LOCAL_PATH) as ds:
+ # ...
+ # do some computation
+ # ...
+
+# more code outside the scope of the with block.
+```
+
+El conjunto de datos se cerrará automáticamente fuera del bloque `with`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{type(ds)=}')
+ assert not ds.closed
+
+# outside the scope of the with block
+assert ds.closed
+```
+
+
+
+La principal ventaja al utilizar `rasterio.open` en vez de `rioxarray.open_rasterio` para abrir un archivo es que este último método abre el archivo y carga inmediatamente su contenido en un `DataDarray` en la memoria.
+
+Por el contrario, al utilizar `rasterio.open` se abre el archivo en su lugar y su contenido _no_ se carga inmediatamente en la memoria. Los datos del archivo _pueden_ leerse, pero esto debe hacerse explícitamente. Esto representa una gran diferencia cuando se trabaja con datos remotos. Transferir todo el contenido a través de una red de datos implica ciertos costos. Por ejemplo, si examinamos los metadatos, que suelen ser mucho más pequeños y pueden transferirse rápidamente, podemos descubrir, por ejemplo, que no está justificado mover todo un _array_ de datos a través de la red.
+
+
+
+***
+
+### Análisis de los atributos DatasetReader
+
+
+
+Cuando se abre un archivo utilizando `rasterio.open`, el objeto instanciado es de la clase `DatasetReader`. Esta clase tiene una serie de atributos y métodos de interés para nosotros:
+
+| | | |
+| --------- | ----------- | -------- |
+| `profile` | `height` | `width` |
+| `shape` | `count` | `nodata` |
+| `crs` | `transform` | `bounds` |
+| `xy` | `index` | `read` |
+
+En primer lugar, dado un `DatasetReader` `ds` asociado a una fuente de datos, el análisis de `ds.profile` devuelve cierta información de diagnóstico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{ds.profile=}')
+```
+
+
+
+Los atributos `ds.height`, `ds.width`, `ds.shape`, `ds.count`, `ds.nodata` y `ds.transform` se incluyen en la salida de `ds.profile`, pero también son accesibles individualmente.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{ds.height=}')
+ print(f'{ds.width=}')
+ print(f'{ds.shape=}')
+ print(f'{ds.count=}')
+ print(f'{ds.nodata=}')
+ print(f'{ds.crs=}')
+ print(f'{ds.transform=}')
+```
+
+***
+
+### Lectura de datos en la memoria
+
+
+
+El método `ds.read` carga un _array_ del archivo de datos en la memoria. Ten en cuenta que esto se puede hacer en archivos locales o remotos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+with rasterio.open(LOCAL_PATH) as ds:
+ array = ds.read()
+ print(f'{array.shape=}')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+with rasterio.open(REMOTE_URL) as ds:
+ array = ds.read()
+ print(f'{array.shape=}')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f'{type(array)=}')
+```
+
+
+
+El _array_ cargado en la memoria con `ds.read` es una matriz NumPy. Este puede ser encapsulado por un Xarray `DataArray` si proporcionamos código adicional para especificar las etiquetas de las coordenadas y demás.
+
+
+
+***
+
+### Mapeo de coordenadas
+
+
+
+Anteriormente, cargamos los datos de un archivo local en un `DataArray` llamado `da` utilizando `rioxarray.open_rasterio`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+da = rio.open_rasterio(LOCAL_PATH)
+da
+```
+
+
+
+De este modo se simplificó la carga de datos ráster de un archivo GeoTIFF en un Xarray `DataArray` a la vez que cargaban los metadatos automáticamente. En particular, las coordenadas asociadas a los píxeles se almacenaron en `da.coords` (los ejes de coordenadas predeterminados son `band`, `x` y `y` para este _array_ tridimensional).
+
+Si ignoramos la dimensión extra de `band`, los píxeles de los datos ráster se asocian con coordenadas de píxel (enteros) y coordenadas espaciales (valores reales, típicamente un punto en el centro de cada píxel).
+
+
+
+(de `http://ioam.github.io/topographica`)
+
+Los accesores `da.isel` y `da.sel` nos permiten extraer porciones del _array_ utilizando coordenadas de píxel o coordenadas espaciales, respectivamente.
+
+
+
+
+
+Si utilizamos `rasterio.open` para abrir un archivo, el atributo `transform` de `DatasetReader` proporciona los detalles sobre cómo realizar la conversión entre coordenadas de píxel y espaciales. Utilizaremos esta propiedad en algunos de los casos prácticos más adelante.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{ds.transform=}')
+ print(f'{np.abs(ds.transform[0])=}')
+ print(f'{np.abs(ds.transform[4])=}')
+```
+
+
+
+El atributo `ds.transform` es un objeto que describe una [_transformación afín_](https://es.wikipedia.org/wiki/Transformaci%C3%B3n_af%C3%ADn) (representada anteriormente como una matriz $2\times3$). Observa que los valores absolutos de las entradas diagonales de la matriz `ds.transform` dan las dimensiones espaciales de los píxeles ($30\mathrm{m}\times30\mathrm{m}$ en este caso).
+
+También podemos utilizar este objeto para convertir las coordenadas de los píxeles en las coordenadas espaciales correspondientes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{ds.transform * (0,0)=}') # top-left pixel
+ print(f'{ds.transform * (0,3660)=}') # bottom-left pixel
+ print(f'{ds.transform * (3660,0)=}') # top-right pixel
+ print(f'{ds.transform * (3660,3660)=}') # bottom-right pixel
+```
+
+
+
+El atributo `ds.bounds` muestra los límites de la región espacial (izquierda, abajo, derecha, arriba).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'coordinate bounds: {ds.bounds=}')
+```
+
+
+
+El método `ds.xy` también convierte coordenadas de índice entero en coordenadas continuas. Observa que `ds.xy` asigna enteros al centro de los píxeles. Los bucles siguientes imprimen la primera esquina superior izquierda de las coordenadas en coordenadas de píxel y, después, las coordenadas espaciales correspondientes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ for k in range(3):
+ for l in range(4):
+ print(f'({k:2d},{l:2d})','\t', end='')
+ print()
+ print()
+ for k in range(3):
+ for l in range(4):
+ e,n = ds.xy(k,l)
+ print(f'({e},{n})','\t', end='')
+ print()
+ print()
+```
+
+
+
+`ds.index` hace lo contrario: dadas las coordenadas espaciales `(x,y)`, devuelve los índices enteros del píxel que contiene ese punto.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(ds.index(500000, 4700015))
+```
+
+
+
+Estas conversiones pueden ser complicadas, sobre todo porque las coordenadas de píxel corresponden a los centros de los píxeles y también porque la segunda coordenada espacial `y` _disminuye_ a medida que la segunda coordenada de píxel _aumenta_. Hacer un seguimiento de detalles tediosos como este es en parte la razón por la que resulta útil cargar desde `rioxarray`, es decir, que nosotros lo hagamos. Pero vale la pena saber que podemos reconstruir este mapeo si es necesario a partir de los metadatos en el archivo GeoTIFF (utilizaremos este hecho más adelante).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(ds.bounds)
+ print(ds.transform * (0.5,0.5)) # Maps to centre of top left pixel
+ print(ds.xy(0,0)) # Same as above
+ print(ds.transform * (0,0)) # Maps to top left corner of top left pixel
+ print(ds.xy(-0.5,-0.5)) # Same as above
+ print(ds.transform[0], ds.transform[4])
+```
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/02_Array_Manipulation_with_Xarray.md b/book/es-419/02_Software_Tools_Techniques/02_Array_Manipulation_with_Xarray.md
new file mode 100644
index 0000000..d99c045
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/02_Array_Manipulation_with_Xarray.md
@@ -0,0 +1,317 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Manipulación de arreglos con [Xarray](https://docs.xarray.dev/en/stable/index.html)
+
+
+
+Hay numerosas formas de trabajar con datos geoespaciales, así que elegir una herramienta puede ser difícil. La principal librería que utilizaremos es [_Xarray_](https://docs.xarray.dev/en/stable/index.html) por sus estructuras de datos `DataArray` y `Dataset`, y sus utilidades asociadas, así como [NumPy](https://numpy.org) y [Pandas](https://pandas.pydata.org) para manipular arreglos numéricos homogéneos y datos tabulares, respectivamente.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+from pathlib import Path
+import numpy as np, pandas as pd, xarray as xr
+import rioxarray as rio
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+***
+
+
+
+ +
+La principal estructura de datos de Xarray es [`DataArray`](https://docs.xarray.dev/en/stable/user-guide/data-structures.html), que ofrece soporte para arreglos multidimensionales etiquetados. El [Projecto Pythia](https://foundations.projectpythia.org/core/xarray.html) proporciona una amplia introducción a este paquete. Nos enfocaremos principalmente en las partes específicas del API Xarray que utilizaremos para nuestros análisis geoespaciales particulares.
+
+Vamos a cargar una estructura de datos `xarray.DataArray` de ejemplo desde un archivo cuya ubicación viene determinada por `LOCAL_PATH`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+```
+
+***
+
+## Análisis de la `repr` enriquecida de `DataArray`
+
+
+
+Cuando se utiliza un cuaderno computacional de Jupyter, los datos Xarray `DataArray` `data` se pueden analizar de forma interactiva.
+
+- La celda de salida contiene un cuaderno computacional Jupyter `repr` enriquecido para la clase `DataArray`.
+- Los triángulos situados junto a los encabezados "Coordinates", "Indexes" y "Attributes" pueden pulsarse con el mouse para mostrar una vista ampliada.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f'{type(data)=}\n')
+data
+```
+
+***
+
+## Análisis de los atributos de `DataArray` mediante programación
+
+
+
+Por supuesto, aunque esta vista gráfica es práctica, también es posible acceder a varios atributos de `DataArray` mediante programación. Esto nos permite escribir una lógica programatica para manipular los `DataArray` condicionalmente según sea necesario. Por ejemplo:
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords)
+```
+
+
+
+Las dimensiones `data.dims` son las cadenas/etiquetas asociadas a los ejes del `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.dims
+```
+
+
+
+Podemos extraer las coordenadas como arreglos NumPy unidimensionales (homogéneas) utilizando los atributos `coords` y `.values`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords['x'].values)
+```
+
+
+
+`data.attrs` es un diccionario que contiene otros metadatos analizados a partir de las etiquetas GeoTIFF (los "Atributos" en la vista gráfica). Una vez más, esta es la razón por la que `rioxarray` es útil. Es posible escribir código que cargue datos de varios formatos de archivo en Xarray `DataArray`, pero este paquete encapsula mucho del código desordenado que, por ejemplo, rellenaría `data.attrs`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.attrs
+```
+
+***
+
+## Uso del método de acceso `rio` de `DataArray`
+
+
+
+Tal como se mencionó, `rioxarray` extiende la clase `xarray.DataArray` con un método de acceso llamado `rio`. El método de acceso `rio` agrega efectivamente un espacio de nombres con una variedad de atributos. Podemos usar una lista de comprensión de Python para mostrar los que no empiezan con guión bajo (los llamados métodos/atributos "private" y "dunder").
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+[name for name in dir(data.rio) if not name.startswith('_')]
+```
+
+
+
+El atributo `data.rio.crs` es importante para nuestros propósitos. Proporciona acceso al sistema de referencia de coordenadas asociado a este conjunto de datos ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(type(data.rio.crs))
+print(data.rio.crs)
+```
+
+
+
+El atributo `.rio.crs` es una estructura de datos de la clase `CRS` del proyecto [pyproj](https://pyproj4.github.io/pyproj/stable/index.html). La `repr` de Python para esta clase devuelve una cadena como `EPSG:32610`. Este número se refiere al [conjunto de datos de parámetros geodésicos _European Petroleum Survey Group_ (EPGS)](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset) (en español, Grupo Europeo de Estudio sobre el Petróleo).
+
+De [Wikipedia](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset):
+
+> El [EPSG Geodetic Parameter Dataset (también conocido como registro EPSG)](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset) es un registro público de [datums geodésicos](https://es.wikipedia.org/wiki/Sistema_de_referencia_geod%C3%A9sico), [sistemas de referencia espacial](https://es.wikipedia.org/wiki/Sistema_de_referencia_espacial), [elipsoides terrestres](https://es.wikipedia.org/wiki/Elipsoide_de_referencia), transformaciones de coordenadas y [unidades de medida](https://es.wikipedia.org/wiki/Unidad_de_medida) relacionadas, originados por un miembro del [EPGS](https://en.wikipedia.org/wiki/European_Petroleum_Survey_Group) en 1985. A cada entidad se le asigna un código EPSG comprendido entre 1024 y 32767, junto con una representación estándar de [texto conocido (WKT)](https://en.wikipedia.org/wiki/Well-known_text_representation_of_coordinate_reference_systems) legible por máquina. El mantenimiento del conjunto de datos corre a cargo del Comité de Geomática [IOGP](https://en.wikipedia.org/wiki/International_Association_of_Oil_%26_Gas_Producers).
+
+
+
+***
+
+## Manipulación de los datos en un `DataArray`
+
+
+
+Estos datos se almacenan utilizando un CRS [ sistema de coordenadas universal transversal de Mercator (UTM)](https://en.wikipedia.org/wiki/Universal_Transverse_Mercator_coordinate_system) (por sus siglas en inglés de _Mercator transversal universal_) particular. Las etiquetas de las coordenadas serían convencionalmente _este_ y _norte_. Sin embargo, a la hora de hacer el trazo, será conveniente utilizar _longitud_ y _latitud_ en su lugar. Reetiquetaremos las coordenadas para reflejar esto, es decir, la coordenada llamada `x` se reetiquetará como `longitude` y la coordenada llamada `y` se reetiquetará como `latitude`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = data.rename({'x':'longitude', 'y':'latitude'})
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords)
+```
+
+
+
+Una vez más, aunque los valores numéricos almacenados en los arreglos de coordenadas no tienen sentido estrictamente como valores (longitud, latitud), aplicaremos estas etiquetas ahora para simplificar el trazado más adelante.
+
+Los objetos Xarray `DataArray` permiten estraer subconjuntos de forma muy similar a las listas de Python. Las dos celdas siguientes extraen ambas el mismo subarreglo mediante dos llamadas a métodos diferentes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.isel(longitude=slice(0,2))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.sel(longitude=[499_995, 500_025])
+```
+
+
+
+En vez de utilizar paréntesis para cortar secciones de arreglos (como en NumPy), para `DataArray`, podemos utilizar los métodos `sel` o `isel` para seleccionar subconjuntos por valores de coordenadas continuas o por posiciones enteras (es decir, coordenadas de "píxel") respectivamente. Esto es similar al uso de `.loc` and `.iloc` en Pandas para extraer entradas de una Pandas `Series` o `DataFrame`.
+
+Si tomamos un subconjunto en 2D de los `DataArray` `data` 3D, podemos graficarlo usando el método de acceso `.plot` (hablaremos al respecto más adelante).
+
+
+
+```python jupyter={"source_hidden": false}
+data.isel(band=0).plot();
+```
+
+
+
+Este gráfico tarda un poco en procesarse porque el arreglo representado tiene $3,600\times3,600$ píxeles. Podemos utilizar la función `slice` de Python para extraer, por ejemplo, cada 100 píxeles en cualquier dirección para trazar una imagen de menor resolución mucho más rápido.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 100
+subset = slice(0,None,steps)
+view = data.isel(longitude=subset, latitude=subset, band=0)
+view.plot();
+```
+
+
+
+El gráfico producido es bastante oscuro (lo que refleja que la mayoría de las entradas son cero según la leyenda). Observa que los ejes se etiquetan automáticamente utilizando las `coords` que renombramos antes.
+
+
+
+***
+
+## Extracción de datos `DataArray` a NumPy, Pandas
+
+
+
+Observa que un `DataArray` encapsula de un arreglo NumPy. Ese arreglo NumPy se puede recuperar usando el atributo `.values`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+array = data.values
+print(f'{type(array)=}')
+print(f'{array.shape=}')
+print(f'{array.dtype=}')
+print(f'{array.nbytes=}')
+```
+
+
+
+Estos datos ráster se almacenan como datos enteros sin signo de 8 bits, es decir, un byte por cada píxel. Un entero de 8 bits sin signo puede representar valores enteros entre 0 y 255. En un arreglo con algo más de trece millones de elementos, eso significa que hay muchos valores repetidos. Podemos verlo poniendo los valores de los píxeles en una Pandas `Series` y usando el método `.value_counts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+s_flat = pd.Series(array.flatten()).value_counts()
+s_flat.sort_index()
+```
+
+
+
+La mayoría de las entradas de este arreglo ráster son cero. Los valores numéricos varían entre 0 y 100 con la excepción de unos 1,700 píxeles con el valor 255. Esto tendrá más sentido cuando hablemos de la especificación del producto de datos DIST.
+
+
+
+***
+
+## Acumulación y concatenación de una secuencia de `DataArrays`
+
+
+
+A menudo es conveniente apilar múltiples arreglos bidimensionales de datos ráster en un único arreglo tridimensional. En NumPy, esto se hace típicamente con la función [`numpy.concatenate`](https://numpy.org/doc/stable/reference/generated/numpy.concatenate.html). Hay una funcionalidad similar en Xarray—[`xarray.concat`](https://docs.xarray.dev/en/stable/generated/xarray.concat.html) (que es similar en diseño a la función [`pandas.concat`](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.concat.html)). La principal diferencia entre `numpy.concatenate` y `xarray.concat` es que esta última función debe tener en cuenta las _coordenadas etiquetadas_, mientras que la primera no. Esto es importante cuando, por ejemplo, los ejes de coordenadas de dos rásters se superponen pero no están perfectamente alineados.
+
+Para ver cómo funciona el apilamiento de rásteres, empezaremos haciendo una lista de tres archivos GeoTIFF (almacenados localmente), inicializando una lista de `stack` vacía, y después construyendo una lista de `DataArrays` en un bucle.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+RASTER_FILES = list((Path(FILE_STEM, 'assets').glob('OPERA*VEG*.tif')))
+
+stack = []
+for path in RASTER_FILES:
+ print(f"Stacking {path.name}..")
+ data = rio.open_rasterio(path).rename(dict(x='longitude', y='latitude'))
+ band_name = path.stem.split('_')[-1]
+ data.coords.update({'band': [band_name]})
+ data.attrs = dict(description=f"OPERA DIST product", units=None)
+ stack.append(data)
+```
+
+
+
+He aquí algunas observaciones importantes sobre el bucle de código anterior:
+
+- El uso de `rioxarray.open_rasterio` para cargar un Xarray `DataArray` en memoria hace mucho trabajo por nosotros. En particular, se asegura de que las coordenadas continuas están alineadas con las coordenadas de píxeles subyacentes.
+- De manera predeterminada, `data.coords` tiene las claves `x` y `y` que elegimos reetiquetar como `longitude` y `latitude` respectivamente. Técnicamente, los valores de las coordenadas continuas que se cargaron desde este archivo GeoTIFF en particular se expresan en coordenadas UTM (es decir, este y norte), pero, posteriormente, al trazar, las etiquetas `longitude` y `latitude` serán más convenientes.
+- `data.coords['band']`, tal como se cargó desde el archivo, tiene el valor `1`. Elegimos sobrescribir ese valor con el nombre de la banda (que extraemos del nombre del archivo como `band_name`).
+- De manera predeterminada, `rioxarray.open_rasterio` completa `data.attrs` con pares clave-valor extraídos de las etiquetas TIFF. Para diferentes bandas/capas, estos diccionarios de atributos podrían tener claves o valores conflictivos. Puede ser aconsejable conservar estos metadatos en algunas circunstancias. Simplemente elegimos descartarlos en este contexto para evitar posibles conflictos. El diccionario mínimo de atributos de la estructura de datos final tendrá como únicas claves `description` y `units`.
+
+Dado que construimos una lista de `DataArray` en la lista `stack`, podemos ensamblar un `DataArray` tridimensional utilizando `xarray.concat`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = xr.concat(stack, dim='band')
+```
+
+
+
+La función `xarray.concat` acepta una secuencia de objetos `xarray.DataArray` con dimensiones conformes y los _concatena_ a lo largo de una dimensión especificada. Para este ejemplo, apilamos rásteres bidimensionales que corresponden a diferentes bandas o capas. Por eso utilizamos la opción `dim='band'` en la llamada a `xarray.concat`. Más adelante, en cambio, apilaremos rásteres bidimensionales a lo largo de un eje _temporal_ (esto implica un código ligeramente diferente para garantizar el etiquetado y la alineación correctos).
+
+Examinemos `stack` mediante su `repr`en este cuaderno computacional Jupyter.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+
+
+Observa que `stack` tiene un CRS asociado que fue analizado por `rioxarray.open_rasterio`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack.rio.crs
+```
+
+
+
+Este proceso es muy útil para el análisis (suponiendo que haya suficiente memoria disponible para almacenar toda la colección de rásteres). Más adelante, utilizaremos este enfoque varias veces para manipular colecciones de rásteres de dimensiones conformes. El apilamiento se puede utilizar para producir una visualización dinámica con un control deslizante o, alternativamente, para producir un gráfico estático.
+
+
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md b/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md
new file mode 100644
index 0000000..e156d8b
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md
@@ -0,0 +1,486 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Visualización de datos con GeoViews y HvPlot
+
+
+
+Las principales herramientas que utilizaremos para la visualización de datos provienen de la familia [Holoviz](https://holoviz.org/) de librerías Python, principalmente [GeoViews](https://geoviews.org/) y [hvPlot](https://hvplot.holoviz.org/). Estas están construidas en gran parte sobre [HoloViews](https://holoviews.org/) y soportan múltiples _backends_ para la representación de gráficos ([Bokeh](http://bokeh.pydata.org/) para visualización interactiva y [Matplotlib](http://matplotlib.org/) para gráficos estáticos con calidad de publicación.
+
+
+
+***
+
+## [GeoViews](https://geoviews.org/)
+
+
+
+
+
+La principal estructura de datos de Xarray es [`DataArray`](https://docs.xarray.dev/en/stable/user-guide/data-structures.html), que ofrece soporte para arreglos multidimensionales etiquetados. El [Projecto Pythia](https://foundations.projectpythia.org/core/xarray.html) proporciona una amplia introducción a este paquete. Nos enfocaremos principalmente en las partes específicas del API Xarray que utilizaremos para nuestros análisis geoespaciales particulares.
+
+Vamos a cargar una estructura de datos `xarray.DataArray` de ejemplo desde un archivo cuya ubicación viene determinada por `LOCAL_PATH`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+```
+
+***
+
+## Análisis de la `repr` enriquecida de `DataArray`
+
+
+
+Cuando se utiliza un cuaderno computacional de Jupyter, los datos Xarray `DataArray` `data` se pueden analizar de forma interactiva.
+
+- La celda de salida contiene un cuaderno computacional Jupyter `repr` enriquecido para la clase `DataArray`.
+- Los triángulos situados junto a los encabezados "Coordinates", "Indexes" y "Attributes" pueden pulsarse con el mouse para mostrar una vista ampliada.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f'{type(data)=}\n')
+data
+```
+
+***
+
+## Análisis de los atributos de `DataArray` mediante programación
+
+
+
+Por supuesto, aunque esta vista gráfica es práctica, también es posible acceder a varios atributos de `DataArray` mediante programación. Esto nos permite escribir una lógica programatica para manipular los `DataArray` condicionalmente según sea necesario. Por ejemplo:
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords)
+```
+
+
+
+Las dimensiones `data.dims` son las cadenas/etiquetas asociadas a los ejes del `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.dims
+```
+
+
+
+Podemos extraer las coordenadas como arreglos NumPy unidimensionales (homogéneas) utilizando los atributos `coords` y `.values`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords['x'].values)
+```
+
+
+
+`data.attrs` es un diccionario que contiene otros metadatos analizados a partir de las etiquetas GeoTIFF (los "Atributos" en la vista gráfica). Una vez más, esta es la razón por la que `rioxarray` es útil. Es posible escribir código que cargue datos de varios formatos de archivo en Xarray `DataArray`, pero este paquete encapsula mucho del código desordenado que, por ejemplo, rellenaría `data.attrs`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.attrs
+```
+
+***
+
+## Uso del método de acceso `rio` de `DataArray`
+
+
+
+Tal como se mencionó, `rioxarray` extiende la clase `xarray.DataArray` con un método de acceso llamado `rio`. El método de acceso `rio` agrega efectivamente un espacio de nombres con una variedad de atributos. Podemos usar una lista de comprensión de Python para mostrar los que no empiezan con guión bajo (los llamados métodos/atributos "private" y "dunder").
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+[name for name in dir(data.rio) if not name.startswith('_')]
+```
+
+
+
+El atributo `data.rio.crs` es importante para nuestros propósitos. Proporciona acceso al sistema de referencia de coordenadas asociado a este conjunto de datos ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(type(data.rio.crs))
+print(data.rio.crs)
+```
+
+
+
+El atributo `.rio.crs` es una estructura de datos de la clase `CRS` del proyecto [pyproj](https://pyproj4.github.io/pyproj/stable/index.html). La `repr` de Python para esta clase devuelve una cadena como `EPSG:32610`. Este número se refiere al [conjunto de datos de parámetros geodésicos _European Petroleum Survey Group_ (EPGS)](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset) (en español, Grupo Europeo de Estudio sobre el Petróleo).
+
+De [Wikipedia](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset):
+
+> El [EPSG Geodetic Parameter Dataset (también conocido como registro EPSG)](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset) es un registro público de [datums geodésicos](https://es.wikipedia.org/wiki/Sistema_de_referencia_geod%C3%A9sico), [sistemas de referencia espacial](https://es.wikipedia.org/wiki/Sistema_de_referencia_espacial), [elipsoides terrestres](https://es.wikipedia.org/wiki/Elipsoide_de_referencia), transformaciones de coordenadas y [unidades de medida](https://es.wikipedia.org/wiki/Unidad_de_medida) relacionadas, originados por un miembro del [EPGS](https://en.wikipedia.org/wiki/European_Petroleum_Survey_Group) en 1985. A cada entidad se le asigna un código EPSG comprendido entre 1024 y 32767, junto con una representación estándar de [texto conocido (WKT)](https://en.wikipedia.org/wiki/Well-known_text_representation_of_coordinate_reference_systems) legible por máquina. El mantenimiento del conjunto de datos corre a cargo del Comité de Geomática [IOGP](https://en.wikipedia.org/wiki/International_Association_of_Oil_%26_Gas_Producers).
+
+
+
+***
+
+## Manipulación de los datos en un `DataArray`
+
+
+
+Estos datos se almacenan utilizando un CRS [ sistema de coordenadas universal transversal de Mercator (UTM)](https://en.wikipedia.org/wiki/Universal_Transverse_Mercator_coordinate_system) (por sus siglas en inglés de _Mercator transversal universal_) particular. Las etiquetas de las coordenadas serían convencionalmente _este_ y _norte_. Sin embargo, a la hora de hacer el trazo, será conveniente utilizar _longitud_ y _latitud_ en su lugar. Reetiquetaremos las coordenadas para reflejar esto, es decir, la coordenada llamada `x` se reetiquetará como `longitude` y la coordenada llamada `y` se reetiquetará como `latitude`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = data.rename({'x':'longitude', 'y':'latitude'})
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords)
+```
+
+
+
+Una vez más, aunque los valores numéricos almacenados en los arreglos de coordenadas no tienen sentido estrictamente como valores (longitud, latitud), aplicaremos estas etiquetas ahora para simplificar el trazado más adelante.
+
+Los objetos Xarray `DataArray` permiten estraer subconjuntos de forma muy similar a las listas de Python. Las dos celdas siguientes extraen ambas el mismo subarreglo mediante dos llamadas a métodos diferentes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.isel(longitude=slice(0,2))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.sel(longitude=[499_995, 500_025])
+```
+
+
+
+En vez de utilizar paréntesis para cortar secciones de arreglos (como en NumPy), para `DataArray`, podemos utilizar los métodos `sel` o `isel` para seleccionar subconjuntos por valores de coordenadas continuas o por posiciones enteras (es decir, coordenadas de "píxel") respectivamente. Esto es similar al uso de `.loc` and `.iloc` en Pandas para extraer entradas de una Pandas `Series` o `DataFrame`.
+
+Si tomamos un subconjunto en 2D de los `DataArray` `data` 3D, podemos graficarlo usando el método de acceso `.plot` (hablaremos al respecto más adelante).
+
+
+
+```python jupyter={"source_hidden": false}
+data.isel(band=0).plot();
+```
+
+
+
+Este gráfico tarda un poco en procesarse porque el arreglo representado tiene $3,600\times3,600$ píxeles. Podemos utilizar la función `slice` de Python para extraer, por ejemplo, cada 100 píxeles en cualquier dirección para trazar una imagen de menor resolución mucho más rápido.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 100
+subset = slice(0,None,steps)
+view = data.isel(longitude=subset, latitude=subset, band=0)
+view.plot();
+```
+
+
+
+El gráfico producido es bastante oscuro (lo que refleja que la mayoría de las entradas son cero según la leyenda). Observa que los ejes se etiquetan automáticamente utilizando las `coords` que renombramos antes.
+
+
+
+***
+
+## Extracción de datos `DataArray` a NumPy, Pandas
+
+
+
+Observa que un `DataArray` encapsula de un arreglo NumPy. Ese arreglo NumPy se puede recuperar usando el atributo `.values`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+array = data.values
+print(f'{type(array)=}')
+print(f'{array.shape=}')
+print(f'{array.dtype=}')
+print(f'{array.nbytes=}')
+```
+
+
+
+Estos datos ráster se almacenan como datos enteros sin signo de 8 bits, es decir, un byte por cada píxel. Un entero de 8 bits sin signo puede representar valores enteros entre 0 y 255. En un arreglo con algo más de trece millones de elementos, eso significa que hay muchos valores repetidos. Podemos verlo poniendo los valores de los píxeles en una Pandas `Series` y usando el método `.value_counts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+s_flat = pd.Series(array.flatten()).value_counts()
+s_flat.sort_index()
+```
+
+
+
+La mayoría de las entradas de este arreglo ráster son cero. Los valores numéricos varían entre 0 y 100 con la excepción de unos 1,700 píxeles con el valor 255. Esto tendrá más sentido cuando hablemos de la especificación del producto de datos DIST.
+
+
+
+***
+
+## Acumulación y concatenación de una secuencia de `DataArrays`
+
+
+
+A menudo es conveniente apilar múltiples arreglos bidimensionales de datos ráster en un único arreglo tridimensional. En NumPy, esto se hace típicamente con la función [`numpy.concatenate`](https://numpy.org/doc/stable/reference/generated/numpy.concatenate.html). Hay una funcionalidad similar en Xarray—[`xarray.concat`](https://docs.xarray.dev/en/stable/generated/xarray.concat.html) (que es similar en diseño a la función [`pandas.concat`](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.concat.html)). La principal diferencia entre `numpy.concatenate` y `xarray.concat` es que esta última función debe tener en cuenta las _coordenadas etiquetadas_, mientras que la primera no. Esto es importante cuando, por ejemplo, los ejes de coordenadas de dos rásters se superponen pero no están perfectamente alineados.
+
+Para ver cómo funciona el apilamiento de rásteres, empezaremos haciendo una lista de tres archivos GeoTIFF (almacenados localmente), inicializando una lista de `stack` vacía, y después construyendo una lista de `DataArrays` en un bucle.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+RASTER_FILES = list((Path(FILE_STEM, 'assets').glob('OPERA*VEG*.tif')))
+
+stack = []
+for path in RASTER_FILES:
+ print(f"Stacking {path.name}..")
+ data = rio.open_rasterio(path).rename(dict(x='longitude', y='latitude'))
+ band_name = path.stem.split('_')[-1]
+ data.coords.update({'band': [band_name]})
+ data.attrs = dict(description=f"OPERA DIST product", units=None)
+ stack.append(data)
+```
+
+
+
+He aquí algunas observaciones importantes sobre el bucle de código anterior:
+
+- El uso de `rioxarray.open_rasterio` para cargar un Xarray `DataArray` en memoria hace mucho trabajo por nosotros. En particular, se asegura de que las coordenadas continuas están alineadas con las coordenadas de píxeles subyacentes.
+- De manera predeterminada, `data.coords` tiene las claves `x` y `y` que elegimos reetiquetar como `longitude` y `latitude` respectivamente. Técnicamente, los valores de las coordenadas continuas que se cargaron desde este archivo GeoTIFF en particular se expresan en coordenadas UTM (es decir, este y norte), pero, posteriormente, al trazar, las etiquetas `longitude` y `latitude` serán más convenientes.
+- `data.coords['band']`, tal como se cargó desde el archivo, tiene el valor `1`. Elegimos sobrescribir ese valor con el nombre de la banda (que extraemos del nombre del archivo como `band_name`).
+- De manera predeterminada, `rioxarray.open_rasterio` completa `data.attrs` con pares clave-valor extraídos de las etiquetas TIFF. Para diferentes bandas/capas, estos diccionarios de atributos podrían tener claves o valores conflictivos. Puede ser aconsejable conservar estos metadatos en algunas circunstancias. Simplemente elegimos descartarlos en este contexto para evitar posibles conflictos. El diccionario mínimo de atributos de la estructura de datos final tendrá como únicas claves `description` y `units`.
+
+Dado que construimos una lista de `DataArray` en la lista `stack`, podemos ensamblar un `DataArray` tridimensional utilizando `xarray.concat`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = xr.concat(stack, dim='band')
+```
+
+
+
+La función `xarray.concat` acepta una secuencia de objetos `xarray.DataArray` con dimensiones conformes y los _concatena_ a lo largo de una dimensión especificada. Para este ejemplo, apilamos rásteres bidimensionales que corresponden a diferentes bandas o capas. Por eso utilizamos la opción `dim='band'` en la llamada a `xarray.concat`. Más adelante, en cambio, apilaremos rásteres bidimensionales a lo largo de un eje _temporal_ (esto implica un código ligeramente diferente para garantizar el etiquetado y la alineación correctos).
+
+Examinemos `stack` mediante su `repr`en este cuaderno computacional Jupyter.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+
+
+Observa que `stack` tiene un CRS asociado que fue analizado por `rioxarray.open_rasterio`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack.rio.crs
+```
+
+
+
+Este proceso es muy útil para el análisis (suponiendo que haya suficiente memoria disponible para almacenar toda la colección de rásteres). Más adelante, utilizaremos este enfoque varias veces para manipular colecciones de rásteres de dimensiones conformes. El apilamiento se puede utilizar para producir una visualización dinámica con un control deslizante o, alternativamente, para producir un gráfico estático.
+
+
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md b/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md
new file mode 100644
index 0000000..e156d8b
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md
@@ -0,0 +1,486 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Visualización de datos con GeoViews y HvPlot
+
+
+
+Las principales herramientas que utilizaremos para la visualización de datos provienen de la familia [Holoviz](https://holoviz.org/) de librerías Python, principalmente [GeoViews](https://geoviews.org/) y [hvPlot](https://hvplot.holoviz.org/). Estas están construidas en gran parte sobre [HoloViews](https://holoviews.org/) y soportan múltiples _backends_ para la representación de gráficos ([Bokeh](http://bokeh.pydata.org/) para visualización interactiva y [Matplotlib](http://matplotlib.org/) para gráficos estáticos con calidad de publicación.
+
+
+
+***
+
+## [GeoViews](https://geoviews.org/)
+
+
+
+ +
+De la [documentación de GeoViews](https://geoviews.org/index.html):
+
+> GeoViews es una librería de [Python](http://python.org/) que facilita la exploración y visualización de conjuntos de datos geográficos, meteorológicos y oceanográficos, como los que se utilizan en la investigación meteorológica, climática y de teledetección.
+>
+> GeoViews se basa en la biblioteca [HoloViews](http://holoviews.org/) y permite crear visualizaciones flexibles de datos multidimensionales. GeoViews agrega una familia de tipos de gráficos geográficos basados en la librería [Cartopy](http://scitools.org.uk/cartopy), trazados con los paquetes [Matplotlib](http://matplotlib.org/) o [Bokeh](http://bokeh.pydata.org/). Con GeoViews, puedes trabajar de forma fácil y natural con grandes conjuntos de datos geográficos multidimensionales, visualizando al instante cualquier subconjunto o combinación de ellos. Al mismo tiempo, podrás acceder siempre a los datos crudos subyacentes a cualquier gráfico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import warnings
+warnings.filterwarnings('ignore')
+from pathlib import Path
+from pprint import pprint
+
+import geoviews as gv
+gv.extension('bokeh')
+from geoviews import opts
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+***
+
+### Visualización de un mapa base
+
+
+
+Un _mapa base_ o _capa de mosaico_ es útil cuando se muestran datos vectoriales o ráster porque nos permite superponer los datos geoespaciales relevantes sobre un mapa geográfico conocido como fondo. La principal funcionalidad que utilizaremos es `gv.tile_sources`. Podemos utilizar el método `opts` para especificar parámetros de configuración adicionales. A continuación, utilizaremos el servicio de mapas web _Open Street Map (OSM)_ (en español, Mapas de Calles Abiertos) para crear el objeto `basemap`. Cuando mostramos la representación de este objeto en la celda del cuaderno computacional, el menú de Bokeh que está a la derecha permite la exploración interactiva.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.OSM.opts(width=600, height=400)
+basemap # When displayed, this basemap can be zoomed & panned using the menu at the right
+```
+
+***
+
+### Gráficos de puntos
+
+
+
+Para empezar, vamos a definir una tupla regular en Python para las coordenadas de longitud y latitud de Tokio, Japón.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+tokyo_lonlat = (139.692222, 35.689722)
+print(tokyo_lonlat)
+```
+
+
+
+La clase `geoviews.Points` acepta una lista de tuplas (cada una de la forma `(x, y)`) y construye un objeto `Points` que puede ser visualizado. Podemos superponer el punto creado en los mosaicos OpenStreetMap de `basemap` utilizando el operador `*` en Holoviews. También podemos utilizar `geoviews.opts` para establecer varias preferencias de visualización para estos puntos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+tokyo_point = gv.Points([tokyo_lonlat])
+point_opts = opts.Points(
+ size=48,
+ alpha=0.5,
+ color='red'
+ )
+print(type(tokyo_point))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Use Holoviews * operator to overlay plot on basemap
+# Note: zoom out to see basemap (starts zoomed "all the way in")
+(basemap * tokyo_point).opts(point_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# to avoid starting zoomed all the way in, this zooms "all the way out"
+(basemap * tokyo_point).opts(point_opts, opts.Overlay(global_extent=True))
+```
+
+***
+
+### Gráficos de rectángulos
+
+
+
+- Forma estándar de representar un rectángulo (también llamado caja delimitadora) con vértices $$(x_{\mathrm{min}},y_{\mathrm{min}}), (x_{\mathrm{min}},y_{\mathrm{max}}), (x_{\mathrm{max}},y_{\mathrm{min}}), (x_{\mathrm{max}},y_{\mathrm{max}})$$
+ (suponiendo que $x_{\mathrm{max}}>x_{\mathrm{min}}$ & $y_{\mathrm{max}}>y_{\mathrm{min}}$) es como única cuadrupla
+ $$(x_{\mathrm{min}},y_{\mathrm{min}},x_{\mathrm{max}},y_{\mathrm{max}}),$$
+ es decir, las coordenadas de la esquina inferior izquierda seguidas de las coordenadas de la esquina superior derecha.
+
+ Vamos a crear una función sencilla para generar un rectángulo de un ancho y altura dados, según la coordenada central.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle centered at pt of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+
+
+Podemos probar la función anterior utilizando las coordenadas de longitud y latitud de Marruecos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Verify that the function bounds works as intended
+marrakesh_lonlat = (-7.93, 31.67)
+dlon, dlat = 0.5, 0.25
+marrakesh_bbox = make_bbox(marrakesh_lonlat, dlon, dlat)
+print(marrakesh_bbox)
+```
+
+
+
+La función `geoviews.Rectangles` acepta una lista de cajas delimitadoras (cada uno descrito por una tupla de la forma `(x_min, y_min, x_max, y_max)`) para el trazado. También podemos utilizar `geoviews.opts` para adaptar el rectángulo a nuestras necesidades.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+rectangle = gv.Rectangles([marrakesh_bbox])
+rect_opts = opts.Rectangles(
+ line_width=0,
+ alpha=0.1,
+ color='red'
+ )
+```
+
+
+
+Podemos graficar un punto para Marruecos al igual que antes utilizando `geoviews.Points` (personalizado utilizando `geoviews.opts`).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+marrakesh_point = gv.Points([marrakesh_lonlat])
+point_opts = opts.Points(
+ size=48,
+ alpha=0.25,
+ color='blue'
+ )
+```
+
+
+
+Por último, podemos superponer todas estas características en el mapa base con las opciones aplicadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+(basemap * rectangle * marrakesh_point).opts( rect_opts, point_opts )
+```
+
+
+
+Utilizaremos el método anterior para visualizar _(AOIs)_ al construir consultas de búsqueda para los productos EarthData de la NASA. En particular, la convención de representar una caja delimitadora por ordenadas (izquierda, inferior, derecha, superior) también se utiliza en la API [PySTAC](https://pystac.readthedocs.io/en/stable/).
+
+
+
+***
+
+## [hvPlot](https://hvplot.holoviz.org/)
+
+
+
+
+
+De la [documentación de GeoViews](https://geoviews.org/index.html):
+
+> GeoViews es una librería de [Python](http://python.org/) que facilita la exploración y visualización de conjuntos de datos geográficos, meteorológicos y oceanográficos, como los que se utilizan en la investigación meteorológica, climática y de teledetección.
+>
+> GeoViews se basa en la biblioteca [HoloViews](http://holoviews.org/) y permite crear visualizaciones flexibles de datos multidimensionales. GeoViews agrega una familia de tipos de gráficos geográficos basados en la librería [Cartopy](http://scitools.org.uk/cartopy), trazados con los paquetes [Matplotlib](http://matplotlib.org/) o [Bokeh](http://bokeh.pydata.org/). Con GeoViews, puedes trabajar de forma fácil y natural con grandes conjuntos de datos geográficos multidimensionales, visualizando al instante cualquier subconjunto o combinación de ellos. Al mismo tiempo, podrás acceder siempre a los datos crudos subyacentes a cualquier gráfico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import warnings
+warnings.filterwarnings('ignore')
+from pathlib import Path
+from pprint import pprint
+
+import geoviews as gv
+gv.extension('bokeh')
+from geoviews import opts
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+***
+
+### Visualización de un mapa base
+
+
+
+Un _mapa base_ o _capa de mosaico_ es útil cuando se muestran datos vectoriales o ráster porque nos permite superponer los datos geoespaciales relevantes sobre un mapa geográfico conocido como fondo. La principal funcionalidad que utilizaremos es `gv.tile_sources`. Podemos utilizar el método `opts` para especificar parámetros de configuración adicionales. A continuación, utilizaremos el servicio de mapas web _Open Street Map (OSM)_ (en español, Mapas de Calles Abiertos) para crear el objeto `basemap`. Cuando mostramos la representación de este objeto en la celda del cuaderno computacional, el menú de Bokeh que está a la derecha permite la exploración interactiva.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.OSM.opts(width=600, height=400)
+basemap # When displayed, this basemap can be zoomed & panned using the menu at the right
+```
+
+***
+
+### Gráficos de puntos
+
+
+
+Para empezar, vamos a definir una tupla regular en Python para las coordenadas de longitud y latitud de Tokio, Japón.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+tokyo_lonlat = (139.692222, 35.689722)
+print(tokyo_lonlat)
+```
+
+
+
+La clase `geoviews.Points` acepta una lista de tuplas (cada una de la forma `(x, y)`) y construye un objeto `Points` que puede ser visualizado. Podemos superponer el punto creado en los mosaicos OpenStreetMap de `basemap` utilizando el operador `*` en Holoviews. También podemos utilizar `geoviews.opts` para establecer varias preferencias de visualización para estos puntos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+tokyo_point = gv.Points([tokyo_lonlat])
+point_opts = opts.Points(
+ size=48,
+ alpha=0.5,
+ color='red'
+ )
+print(type(tokyo_point))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Use Holoviews * operator to overlay plot on basemap
+# Note: zoom out to see basemap (starts zoomed "all the way in")
+(basemap * tokyo_point).opts(point_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# to avoid starting zoomed all the way in, this zooms "all the way out"
+(basemap * tokyo_point).opts(point_opts, opts.Overlay(global_extent=True))
+```
+
+***
+
+### Gráficos de rectángulos
+
+
+
+- Forma estándar de representar un rectángulo (también llamado caja delimitadora) con vértices $$(x_{\mathrm{min}},y_{\mathrm{min}}), (x_{\mathrm{min}},y_{\mathrm{max}}), (x_{\mathrm{max}},y_{\mathrm{min}}), (x_{\mathrm{max}},y_{\mathrm{max}})$$
+ (suponiendo que $x_{\mathrm{max}}>x_{\mathrm{min}}$ & $y_{\mathrm{max}}>y_{\mathrm{min}}$) es como única cuadrupla
+ $$(x_{\mathrm{min}},y_{\mathrm{min}},x_{\mathrm{max}},y_{\mathrm{max}}),$$
+ es decir, las coordenadas de la esquina inferior izquierda seguidas de las coordenadas de la esquina superior derecha.
+
+ Vamos a crear una función sencilla para generar un rectángulo de un ancho y altura dados, según la coordenada central.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle centered at pt of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+
+
+Podemos probar la función anterior utilizando las coordenadas de longitud y latitud de Marruecos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Verify that the function bounds works as intended
+marrakesh_lonlat = (-7.93, 31.67)
+dlon, dlat = 0.5, 0.25
+marrakesh_bbox = make_bbox(marrakesh_lonlat, dlon, dlat)
+print(marrakesh_bbox)
+```
+
+
+
+La función `geoviews.Rectangles` acepta una lista de cajas delimitadoras (cada uno descrito por una tupla de la forma `(x_min, y_min, x_max, y_max)`) para el trazado. También podemos utilizar `geoviews.opts` para adaptar el rectángulo a nuestras necesidades.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+rectangle = gv.Rectangles([marrakesh_bbox])
+rect_opts = opts.Rectangles(
+ line_width=0,
+ alpha=0.1,
+ color='red'
+ )
+```
+
+
+
+Podemos graficar un punto para Marruecos al igual que antes utilizando `geoviews.Points` (personalizado utilizando `geoviews.opts`).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+marrakesh_point = gv.Points([marrakesh_lonlat])
+point_opts = opts.Points(
+ size=48,
+ alpha=0.25,
+ color='blue'
+ )
+```
+
+
+
+Por último, podemos superponer todas estas características en el mapa base con las opciones aplicadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+(basemap * rectangle * marrakesh_point).opts( rect_opts, point_opts )
+```
+
+
+
+Utilizaremos el método anterior para visualizar _(AOIs)_ al construir consultas de búsqueda para los productos EarthData de la NASA. En particular, la convención de representar una caja delimitadora por ordenadas (izquierda, inferior, derecha, superior) también se utiliza en la API [PySTAC](https://pystac.readthedocs.io/en/stable/).
+
+
+
+***
+
+## [hvPlot](https://hvplot.holoviz.org/)
+
+
+
+ +
+- [hvPlot](https://hvplot.holoviz.org/) está diseñado para extender la API `.plot` de `DataFrames` de Pandas.
+- Funciona para `DataFrames` de Pandas y `DataArrays`/`Datasets` de Xarray.
+
+
+
+***
+
+### Graficar desde un DataFrame con hvplot.pandas
+
+
+
+El código siguiente carga un `DataFrame` de Pandas con datos de temperatura.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import pandas as pd, numpy as np
+from pathlib import Path
+LOCAL_PATH = Path(FILE_STEM, 'assets/temperature.csv')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = pd.read_csv(LOCAL_PATH, index_col=0, parse_dates=[0])
+df.head()
+```
+
+***
+
+#### Revisando la API de `DataFrame.plot` de Pandas
+
+
+
+Vamos a extraer un subconjunto de columnas de este `DataFrame` y generar un gráfico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast = df[['Vancouver', 'Portland', 'San Francisco', 'Seattle', 'Los Angeles']]
+west_coast.head()
+```
+
+
+
+La API de `.plot` de `DataFrame` de Pandas proporciona acceso a varios métodos de visualización. Aquí usaremos `.plot.line`, pero hay otras opciones disponibles (por ejemplo, `.plot.area`, `.plot.bar`, `.plot.nb`, `.plot.scatter`, etc.). Esta API se ha repetido en varias librerías debido a su conveniencia.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast.plot.line(); # This produces a static Matplotlib plot
+```
+
+***
+
+#### Usando la API de hvPlot `DataFrame.hvplot`
+
+
+
+Importando `hvplot.pandas`, se puede generar un gráfico interactivo similar. La API para `.hvplot` imita esto para `.plot`. Por ejemplo, podemos generar la gráfica de línea anterior usando `.hvplot.line`. En este caso, el _backend_ para los gráficos por defecto es Bokeh, así que el gráfico es _interactivo_.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import hvplot.pandas
+west_coast.hvplot.line() # This produces an interactive Bokeh plot
+```
+
+
+
+La API `.plot` de DataFrame de Pandas proporciona acceso a una serie de métodos de graficación.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast.hvplot.line(width=600, height=300, grid=True)
+```
+
+
+
+La API `hvplot` también funciona cuando está enlazada junto con otras llamadas del método `DataFrame`. Por ejemplo, podemos muestrear los datos de temperatura y calcular la media para suavizarlos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+smoothed = west_coast.resample('2d').mean()
+smoothed.hvplot.line(width=600, height=300, grid=True)
+```
+
+***
+
+### Graficar desde un `DataArray` con `hvplot.xarray`
+
+
+
+La API `.plot` de Pandas también se extendió a Xarray, es decir, para `DataArray`. de Xarray
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import xarray as xr
+import hvplot.xarray
+import rioxarray as rio
+```
+
+
+
+Para empezar, carga un archivo GeoTIFF local usando `rioxarray` en una estructura Zarray de `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = rio.open_rasterio(LOCAL_PATH)
+data
+```
+
+
+
+Hacemos algunos cambios menores al `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = data.squeeze() # to reduce 3D array with singleton dimension to 2D array
+data = data.rename({'x':'easting', 'y':'northing'})
+data
+```
+
+***
+
+#### Revisando la API `DataFrame.plot` de Pandas
+
+
+
+La API `DataArray.plot` por defecto usa el `pcolormesh` de Matplotlib para mostrar un arreglo de 2D almacenado dentro de un `DataArray`. La renderización de esta imagen moderadamente de alta resolución lleva un poco de tiempo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.plot(); # by default, uses pcolormesh
+```
+
+***
+
+#### Usando la API de hvPlot `DataFrame.hvplot`
+
+
+
+De nuevo, la API `DataArray.hvplot` imita la API `DataArray.plot`; de forma predeterminada, utiliza una subclase derivada de `holoviews.element.raster.Image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.hvplot() # by default uses Image class
+print(f'{type(plot)=}')
+plot
+```
+
+
+
+El resultado anterior es una visualización interactiva, procesada usando Bokeh. Esto es un poco lento, pero podemos añadir algunas opciones para acelerar la renderización. También se requiere una manipulación de la misma; por ejemplo, la imagen no es cuadrada, el mapa de colores no resalta características útiles, los ejes son transpuestos, etc.
+
+
+
+***
+
+#### Creando opciones para mejorar los gráficos de manera incremental
+
+
+
+Añadamos opciones para mejorar la imagen. Para hacer esto, iniciaremos un diccionario de Python `image_opts` para usar dentro de la llamada al método `image`. Creando opciones para mejorar los gráficos de manera incremental.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(rasterize=True, dynamic=True)
+pprint(image_opts)
+```
+
+
+
+Para empezar, hagamos la llamada explícita a `hvplot.image` y especifiquemos la secuencia de ejes. Y apliquemos las opciones del diccionario `image_opts`. Utilizaremos la operación `dict-unpacking` `**image_opts` cada vez que invoquemos a `data.hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+A continuación, vamos a corregir el ratio y las dimensiones de la imagen.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(frame_width=500, frame_height=500, aspect='equal')
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+A continuación, vamos a corregir el ratio y las dimensiones de la imagen.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update( cmap='hot_r', clim=(0,100), alpha=0.8 )
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+Antes de añadir un mapa de base, tenemos que tener en cuenta el sistema de coordenadas. Esto se almacena en el archivo GeoTIFF y, cuando se lee usando `rioxarray.open_rasterio`, se disponibilizada mediante el atributo `data.rio.crs`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+crs = data.rio.crs
+crs
+```
+
+
+
+Podemos usar el CRS recuperado arriba como un argumento opcional para `hvplot.image`. Ten en cuenta que las coordenadas han cambiado en los ejes, pero las etiquetas no son las correctas. Podemos arreglarlo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(crs=crs)
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+Ahora vamos a corregir las etiquetas. Utilizaremos el sistema Holoviews/GeoViews `opts` para especificar estas opciones.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+label_opts = dict(title='VEG_ANOM_MAX', xlabel='Longitude (degrees)', ylabel='Latitude (degrees)')
+pprint(image_opts)
+pprint(label_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts).opts(**label_opts)
+plot
+```
+
+
+
+Vamos a superponer la imagen en un mapa base para que podamos ver el terreno debajo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+base = gv.tile_sources.ESRI
+base * plot
+```
+
+
+
+Finalmente, como los píxeles blancos distraen vamos a filtrarlos utilizando el método `DataArray` `where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.where(data>0).hvplot.image(x='easting', y='northing', **image_opts).opts(**label_opts)
+plot * base
+```
+
+
+
+En este cuaderno computacional aplicamos algunas estrategias comunes para generar gráficos. Los usaremos extensamente en el resto del tutorial.
+
+
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md b/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md
new file mode 100644
index 0000000..0506822
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md
@@ -0,0 +1,417 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Construyendo visualizaciones avanzadas
+
+
+
+Vamos a aplicar algunas de las herramientas que hemos visto hasta ahora para obtener algunas visualizaciones más sofisticadas. Estas incluirán el uso de datos vectoriales de un `GeoDataFrame` de _GeoPandas_, se construirán gráficos estáticos y dinámicos a partir de un arreglo 3D y se combinarán datos vectoriales y datos ráster.
+
+Como contexto, los archivos que examinaremos se basan en [el incendio McKinney que ocurrió en el 2022](https://en.wikipedia.org/wiki/McKinney_Fire), en el Bosque Nacional Klamath (al oeste del condado de Siskiyou, California). Los datos vectoriales representan una instantánea del límite de un incendio forestal. Los datos ráster corresponden a la alteración que se observó en la superficie de la vegetación (esto se explicará con mayor detalle más adelante).
+
+
+
+## Importación preliminar y direcciones de los archivos
+
+
+
+Para empezar, se necesitan algunas importaciones típicas de paquetes. También definiremos algunas direcciones a archivos locales que contienen datos geoespaciales relevantes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+from pathlib import Path
+import numpy as np, pandas as pd, xarray as xr
+import geopandas as gpd
+import rioxarray as rio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+ASSET_PATH = Path(FILE_STEM, 'assets')
+SHAPE_FILE = ASSET_PATH / 'shapefiles' / 'mckinney' / 'McKinney_NIFC.shp'
+RASTER_FILES = list(ASSET_PATH.glob('OPERA*VEG*.tif'))
+RASTER_FILE = RASTER_FILES[0]
+```
+
+***
+
+## Graficar datos vectoriales desde un `GeoDataFrame`
+
+
+
+
+
+- [hvPlot](https://hvplot.holoviz.org/) está diseñado para extender la API `.plot` de `DataFrames` de Pandas.
+- Funciona para `DataFrames` de Pandas y `DataArrays`/`Datasets` de Xarray.
+
+
+
+***
+
+### Graficar desde un DataFrame con hvplot.pandas
+
+
+
+El código siguiente carga un `DataFrame` de Pandas con datos de temperatura.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import pandas as pd, numpy as np
+from pathlib import Path
+LOCAL_PATH = Path(FILE_STEM, 'assets/temperature.csv')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = pd.read_csv(LOCAL_PATH, index_col=0, parse_dates=[0])
+df.head()
+```
+
+***
+
+#### Revisando la API de `DataFrame.plot` de Pandas
+
+
+
+Vamos a extraer un subconjunto de columnas de este `DataFrame` y generar un gráfico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast = df[['Vancouver', 'Portland', 'San Francisco', 'Seattle', 'Los Angeles']]
+west_coast.head()
+```
+
+
+
+La API de `.plot` de `DataFrame` de Pandas proporciona acceso a varios métodos de visualización. Aquí usaremos `.plot.line`, pero hay otras opciones disponibles (por ejemplo, `.plot.area`, `.plot.bar`, `.plot.nb`, `.plot.scatter`, etc.). Esta API se ha repetido en varias librerías debido a su conveniencia.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast.plot.line(); # This produces a static Matplotlib plot
+```
+
+***
+
+#### Usando la API de hvPlot `DataFrame.hvplot`
+
+
+
+Importando `hvplot.pandas`, se puede generar un gráfico interactivo similar. La API para `.hvplot` imita esto para `.plot`. Por ejemplo, podemos generar la gráfica de línea anterior usando `.hvplot.line`. En este caso, el _backend_ para los gráficos por defecto es Bokeh, así que el gráfico es _interactivo_.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import hvplot.pandas
+west_coast.hvplot.line() # This produces an interactive Bokeh plot
+```
+
+
+
+La API `.plot` de DataFrame de Pandas proporciona acceso a una serie de métodos de graficación.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast.hvplot.line(width=600, height=300, grid=True)
+```
+
+
+
+La API `hvplot` también funciona cuando está enlazada junto con otras llamadas del método `DataFrame`. Por ejemplo, podemos muestrear los datos de temperatura y calcular la media para suavizarlos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+smoothed = west_coast.resample('2d').mean()
+smoothed.hvplot.line(width=600, height=300, grid=True)
+```
+
+***
+
+### Graficar desde un `DataArray` con `hvplot.xarray`
+
+
+
+La API `.plot` de Pandas también se extendió a Xarray, es decir, para `DataArray`. de Xarray
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import xarray as xr
+import hvplot.xarray
+import rioxarray as rio
+```
+
+
+
+Para empezar, carga un archivo GeoTIFF local usando `rioxarray` en una estructura Zarray de `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = rio.open_rasterio(LOCAL_PATH)
+data
+```
+
+
+
+Hacemos algunos cambios menores al `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = data.squeeze() # to reduce 3D array with singleton dimension to 2D array
+data = data.rename({'x':'easting', 'y':'northing'})
+data
+```
+
+***
+
+#### Revisando la API `DataFrame.plot` de Pandas
+
+
+
+La API `DataArray.plot` por defecto usa el `pcolormesh` de Matplotlib para mostrar un arreglo de 2D almacenado dentro de un `DataArray`. La renderización de esta imagen moderadamente de alta resolución lleva un poco de tiempo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.plot(); # by default, uses pcolormesh
+```
+
+***
+
+#### Usando la API de hvPlot `DataFrame.hvplot`
+
+
+
+De nuevo, la API `DataArray.hvplot` imita la API `DataArray.plot`; de forma predeterminada, utiliza una subclase derivada de `holoviews.element.raster.Image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.hvplot() # by default uses Image class
+print(f'{type(plot)=}')
+plot
+```
+
+
+
+El resultado anterior es una visualización interactiva, procesada usando Bokeh. Esto es un poco lento, pero podemos añadir algunas opciones para acelerar la renderización. También se requiere una manipulación de la misma; por ejemplo, la imagen no es cuadrada, el mapa de colores no resalta características útiles, los ejes son transpuestos, etc.
+
+
+
+***
+
+#### Creando opciones para mejorar los gráficos de manera incremental
+
+
+
+Añadamos opciones para mejorar la imagen. Para hacer esto, iniciaremos un diccionario de Python `image_opts` para usar dentro de la llamada al método `image`. Creando opciones para mejorar los gráficos de manera incremental.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(rasterize=True, dynamic=True)
+pprint(image_opts)
+```
+
+
+
+Para empezar, hagamos la llamada explícita a `hvplot.image` y especifiquemos la secuencia de ejes. Y apliquemos las opciones del diccionario `image_opts`. Utilizaremos la operación `dict-unpacking` `**image_opts` cada vez que invoquemos a `data.hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+A continuación, vamos a corregir el ratio y las dimensiones de la imagen.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(frame_width=500, frame_height=500, aspect='equal')
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+A continuación, vamos a corregir el ratio y las dimensiones de la imagen.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update( cmap='hot_r', clim=(0,100), alpha=0.8 )
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+Antes de añadir un mapa de base, tenemos que tener en cuenta el sistema de coordenadas. Esto se almacena en el archivo GeoTIFF y, cuando se lee usando `rioxarray.open_rasterio`, se disponibilizada mediante el atributo `data.rio.crs`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+crs = data.rio.crs
+crs
+```
+
+
+
+Podemos usar el CRS recuperado arriba como un argumento opcional para `hvplot.image`. Ten en cuenta que las coordenadas han cambiado en los ejes, pero las etiquetas no son las correctas. Podemos arreglarlo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(crs=crs)
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+Ahora vamos a corregir las etiquetas. Utilizaremos el sistema Holoviews/GeoViews `opts` para especificar estas opciones.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+label_opts = dict(title='VEG_ANOM_MAX', xlabel='Longitude (degrees)', ylabel='Latitude (degrees)')
+pprint(image_opts)
+pprint(label_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts).opts(**label_opts)
+plot
+```
+
+
+
+Vamos a superponer la imagen en un mapa base para que podamos ver el terreno debajo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+base = gv.tile_sources.ESRI
+base * plot
+```
+
+
+
+Finalmente, como los píxeles blancos distraen vamos a filtrarlos utilizando el método `DataArray` `where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.where(data>0).hvplot.image(x='easting', y='northing', **image_opts).opts(**label_opts)
+plot * base
+```
+
+
+
+En este cuaderno computacional aplicamos algunas estrategias comunes para generar gráficos. Los usaremos extensamente en el resto del tutorial.
+
+
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md b/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md
new file mode 100644
index 0000000..0506822
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md
@@ -0,0 +1,417 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Construyendo visualizaciones avanzadas
+
+
+
+Vamos a aplicar algunas de las herramientas que hemos visto hasta ahora para obtener algunas visualizaciones más sofisticadas. Estas incluirán el uso de datos vectoriales de un `GeoDataFrame` de _GeoPandas_, se construirán gráficos estáticos y dinámicos a partir de un arreglo 3D y se combinarán datos vectoriales y datos ráster.
+
+Como contexto, los archivos que examinaremos se basan en [el incendio McKinney que ocurrió en el 2022](https://en.wikipedia.org/wiki/McKinney_Fire), en el Bosque Nacional Klamath (al oeste del condado de Siskiyou, California). Los datos vectoriales representan una instantánea del límite de un incendio forestal. Los datos ráster corresponden a la alteración que se observó en la superficie de la vegetación (esto se explicará con mayor detalle más adelante).
+
+
+
+## Importación preliminar y direcciones de los archivos
+
+
+
+Para empezar, se necesitan algunas importaciones típicas de paquetes. También definiremos algunas direcciones a archivos locales que contienen datos geoespaciales relevantes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+from pathlib import Path
+import numpy as np, pandas as pd, xarray as xr
+import geopandas as gpd
+import rioxarray as rio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+ASSET_PATH = Path(FILE_STEM, 'assets')
+SHAPE_FILE = ASSET_PATH / 'shapefiles' / 'mckinney' / 'McKinney_NIFC.shp'
+RASTER_FILES = list(ASSET_PATH.glob('OPERA*VEG*.tif'))
+RASTER_FILE = RASTER_FILES[0]
+```
+
+***
+
+## Graficar datos vectoriales desde un `GeoDataFrame`
+
+
+
+ +
+El paquete [GeoPandas](https://geopandas.org/en/stable/index.html) contiene muchas herramientas útiles para trabajar con datos geoespaciales vectoriales. En particular, el [`GeoDataFrame` de GeoPandas](https://geopandas.org/en/stable/docs/reference/api/geopandas.GeoDataFrame.html) es una subclase del `DataFrame` de Pandas que está específicamente diseñado, por ejemplo, para datos vectoriales almacenados en _shapefiles_.
+
+Como ejemplo, vamos a cargar algunos datos vectoriales desde la ruta local `SHAPEFILE` (tal como se definió anteriormente).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df = gpd.read_file(SHAPE_FILE)
+shape_df
+```
+
+
+
+El objeto `shape_df` es un [`GeoDataFrame`](https://geopandas.org/en/stable/docs/reference/api/geopandas.GeoDataFrame.html) que tiene más de dos docenas de columnas de metadatos en una sola fila. La columna principal que nos interesa es la de `geometry`. Esta columna almacena las coordenadas de los vértices de un `MULTIPOLYGON`, es decir, un conjunto de polígonos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df.geometry
+```
+
+
+
+Los vértices de los polígonos parecen expresarse como pares de `(longitude, latitude)`. Podemos determinar qué sistema de coordenadas específico se utiliza examinando el atributo `crs` del `GeoDataFrame`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(shape_df.crs)
+```
+
+
+
+Utilicemos `hvplot` para generar un gráfico de este conjunto de datos vectoriales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df.hvplot()
+```
+
+
+
+La proyección en este gráfico es un poco extraña. La documentación de HoloViz incluye una [discusión sobre las consideraciones cuando se visualizan datos geográficos](https://hvplot.holoviz.org/user_guide/Geographic_Data.html). El punto importante a recordar en este contexto es que la opción `geo=True` es útil.
+
+Vamos a crear dos diccionarios de Python, `shapeplot_opts` y `layout_opts`, y construiremos una visualización.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts= dict(geo=True)
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+print(f"{shapeplot_opts=}")
+print(f"{layout_opts=}")
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+La documentación de HoloViz incluye una [discusión sobre las consideraciones cuando se grafican datos geográficos](https://hvplot.holoviz.org/user_guide/Geographic_Data.html). El punto importante que se debe recordar en el contexto inmediato es que la opción `geo=True` es útil.
+
+- Definir `color=None` significa que los polígonos no se rellenarán.
+- Al especificar `line_width` y `line_color` se modifica la apariencia de los límites de los polígonos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update( color=None ,
+ line_width=2,
+ line_color='red')
+print(shapeplot_opts)
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+Rellenemos los polígonos con color naranja utilizando la opción `color=orange` y hagamos que el área rellenada sea parcialmente transparente especificando un valor para `alpha` entre cero y uno.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update(color='orange', alpha=0.25)
+print(shapeplot_opts)
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+### Agregado de un mapa base
+
+
+
+A continuación, proporcionemos un mapa base y superpongamos los polígonos trazados utilizando el operador `*`. Observa el uso de paréntesis para aplicar el método `opts` a la salida del operador `*`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.ESRI(height=500, width=500, padding=0.1)
+
+(shape_df.hvplot(**shapeplot_opts) * basemap).opts(**layout_opts)
+```
+
+
+
+El mapa base no necesita ser superpuesto como un objeto separado, puede especificarse utilizando la palabra clave `tiles`. Por ejemplo, al establecer `tiles='OSM'` se utilizan los mosaicos de [OpenStreetMap](ttps://www.openstreetmap.org). Observa que las dimensiones de la imagen renderizada probablemente no sean las mismas que las de la imagen anterior con los mosaicos [ESRI](https://www.esri.com), ya que antes especificamos explícitamente la altura de 500, con la opción `height=500`, y el ancho de 500, con la opción `width=500`, en la definición del `basemap`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update(tiles='OSM')
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+Vamos a eliminar la opción `tiles` de `shapeplot_opts` y a vincular el objeto del gráfico resultante al identificador `shapeplot`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+del shapeplot_opts['tiles']
+print(shapeplot_opts)
+
+shapeplot = shape_df.hvplot(**shapeplot_opts)
+shapeplot
+```
+
+### Combinación de datos vectoriales con datos ráster en un gráfico estático
+
+
+
+Combinemos estos datos vectoriales con algunos datos ráster. Cargaremos los datos raster desde un archivo local utilizando la función `rioxarray.open_rasterio`. Por practicidad, usaremos el encadenamiento de métodos para reetiquetar las coordenadas del `DataArray` cargado y usaremos el método `squeeze` para convertir el arreglo tridimensional que se cargó en uno bidimensional.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = (
+ rio.open_rasterio(RASTER_FILE)
+ .rename(dict(x='longitude', y='latitude'))
+ .squeeze()
+ )
+raster
+```
+
+
+
+Utilizaremos un diccionario de Python `image_opts` para almacenar argumentos útiles y pasarlos a `hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ cmap='hot_r',
+ clim=(0, 100),
+ alpha=0.8,
+ project=True,
+ )
+
+raster.hvplot.image(**image_opts)
+```
+
+
+
+Podemos superponer `shapeplot` con los datos ráster graficados utilizando el operador `*`. Podemos utilizar las herramientas `Pan`, `Wheel Zoom` y `Box Zoom` en la barra de herramientas de Bokeh a la derecha del gráfico para hacer _zoom_ y comprobar que los datos vectoriales fueron trazados encima de los datos ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster.hvplot.image(**image_opts) * shapeplot
+```
+
+
+
+Además, podemos superponer los datos vectoriales y ráster en mosaicos ESRI utilizando `basemap`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster.hvplot.image(**image_opts) * shapeplot * basemap
+```
+
+
+
+Observa que muchos de los píxeles blancos oscurecen el gráfico. Resulta que estos píxeles corresponden a ceros en los datos ráster, los cuales pueden ser ignorados sin problema. Podemos filtrarlos utilizando el método `where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = raster.where((raster!=0))
+layout_opts.update(title="McKinney 2022 Wildfires")
+
+(raster.hvplot.image(**image_opts) * shapeplot * basemap).opts(**layout_opts)
+```
+
+***
+
+## Construcción de gráficos estáticos a partir de un arreglo 3D
+
+
+
+Vamos a cargar una secuencia de archivos ráster en un arreglo tridimensional y generaremos algunos gráficos. Para empezar, construiremos un bucle para leer `DataArrays` de los archivos `RASTER_FILES` y utilizaremos `xarray.concat` para generar un único arreglo de rásters tridimensional (es decir, tres rásters de $3,600\times3,600$ apilados verticalmente). Aprenderemos las interpretaciones específicas asociadas con el conjunto de datos ráster en un cuaderno computacional posterior. Por el momento, los trataremos como datos sin procesar con los que experimentaremos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = []
+for path in RASTER_FILES:
+ data = rio.open_rasterio(path).rename(dict(x='longitude', y='latitude'))
+ band_name = path.stem.split('_')[-1]
+ data.coords.update({'band': [band_name]})
+ data.attrs = dict(description=f"OPERA DIST product", units=None)
+ stack.append(data)
+
+stack = xr.concat(stack, dim='band')
+stack = stack.where(stack!=0)
+```
+
+
+
+Renombramos los ejes `longitude` y `latitude` y filtramos los píxeles con valor cero para simplificar la visualización posterior.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+
+
+Una vez que el `stack` de `DataArray` esté construido, podemos enfocarnos en la visualización.
+
+Si queremos generar un gráfico estático con varias imágenes, podemos utilizar `hvplot.image` junto con el método `.layout`. Para ver cómo funciona, empecemos por redefinir los diccionarios `image_opts` y `layout_opts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict( x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ cmap='hot_r',
+ crs=stack.rio.crs,
+ alpha=0.8,
+ project=True,
+ aspect='equal',
+ shared_axes=False,
+ colorbar=True,
+ tiles='ESRI',
+ padding=0.1)
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+```
+
+
+
+Para acelerar el renderizado, construiremos inicialmente un objeto `view` que seleccione subconjuntos de píxeles. Definimos inicialmente el parámetro `steps=200` para restringir la vista a cada 200 píxeles en cualquier dirección. Si reducimos los `steps`, se tarda más en renderizar. Establecer `steps=1` o `steps=None` equivale a seleccionar todos los píxeles.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+layout_opts.update(frame_width=250, frame_height=250)
+
+
+view = stack.isel(latitude=subset, longitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts).layout()
+```
+
+
+
+El método `layout` traza de manera predeterminada cada uno de los tres rásteres seleccionados a lo largo del eje `band` horizontalmente.
+
+
+
+***
+
+## Construcción de una vista dinámica a partir de un arreglo 3D
+
+
+
+Otra forma de visualizar un arreglo tridimensional es asociar un _widget_ de selección a uno de los ejes. Si llamamos a `hvplot.image` sin agregar el método `.layout` el resultado es un _mapa dinámico_. En este caso, el _widget_ de selección nos permite elegir cortes a lo largo del eje `band`.
+
+Una vez más, aumentar el valor del parámetro `steps` reduce el tiempo de renderizado. Reducirlo a `1` o a `None` renderiza a resolución completa.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+layout_opts.update(frame_height=400, frame_width=400)
+
+view = stack.isel(latitude=subset, longitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts)
+```
+
+
+
+Más adelante apilaremos muchos rásters con distintas marcas temporales a lo largo de un eje `time`. Cuando haya muchos cortes, el _widget_ de selección se renderizará como un deslizador en vez de como un menú desplegable. Podemos controlar la ubicación del _widget_ utilizando una opción de palabra clave `widget_location`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+view.hvplot.image(widget_location='bottom_left', **image_opts, **layout_opts)
+```
+
+
+
+Observa que agregar la opción `widget_location` modifica ligeramente la secuencia en la que se especifican las opciones. Es decir, si invocamos algo como
+
+```python
+view.hvplot.image(widget_location='top_left', **image_opts).opts(**layout_opts)
+```
+
+se genera una excepción (de ahí la invocación en la celda de código anterior). Hay algunas dificultades sutiles en la elaboración de la secuencia correcta para aplicar opciones al personalizar objetos HoloViz/Hvplot (principalmente debido a las formas en que los espacios de los nombres se superponen con Bokeh u otros _backends_ de renderizado). La mejor estrategia es empezar con el menor número de opciones posible y experimentar añadiendo opciones de forma incremental hasta que obtengamos la visualización final que deseamos.
+
+
+
+***
+
+### Combinación de datos vectoriales con datos ráster en una vista dinámica.
+
+
+
+Por último, vamos a superponer nuestros datos vectoriales anteriores, los límites del incendio forestal, con la vista dinámica de este arreglo tridimensional de rásteres. Podemos utilizar el operador `*` para combinar la salida de `hvplot.image` con `shapeplot`, la vista renderizada de los datos vectoriales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+view = stack.isel(latitude=subset, longitude=subset)
+
+image_opts.update(colorbar=False)
+layout_opts.update(frame_height=500, frame_width=500)
+(view.hvplot.image(**image_opts) * shapeplot).opts(**layout_opts)
+```
+
+
+
+Una vez más, la correcta especificación de las opciones puede requerir un poco de experimentación. Las ideas importantes que se deben tomar en cuenta aquí son:
+
+- cómo cargar conjuntos de datos relevantes con `geopandas` and `rioxarray`, y
+- cómo utilizar `hvplot` de forma interactiva e incremental para generar visualizaciones atractivas.
+
+
+
+***
diff --git a/book/es-419/03_Using_NASA_EarthData/01_Using_OPERA_DIST_Products.md b/book/es-419/03_Using_NASA_EarthData/01_Using_OPERA_DIST_Products.md
new file mode 100644
index 0000000..81569dc
--- /dev/null
+++ b/book/es-419/03_Using_NASA_EarthData/01_Using_OPERA_DIST_Products.md
@@ -0,0 +1,343 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.4
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Utilización de los productos OPERA DIST
+
+
+
+## El proyecto OPERA
+
+
+
+
+
+
+
+
+El paquete [GeoPandas](https://geopandas.org/en/stable/index.html) contiene muchas herramientas útiles para trabajar con datos geoespaciales vectoriales. En particular, el [`GeoDataFrame` de GeoPandas](https://geopandas.org/en/stable/docs/reference/api/geopandas.GeoDataFrame.html) es una subclase del `DataFrame` de Pandas que está específicamente diseñado, por ejemplo, para datos vectoriales almacenados en _shapefiles_.
+
+Como ejemplo, vamos a cargar algunos datos vectoriales desde la ruta local `SHAPEFILE` (tal como se definió anteriormente).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df = gpd.read_file(SHAPE_FILE)
+shape_df
+```
+
+
+
+El objeto `shape_df` es un [`GeoDataFrame`](https://geopandas.org/en/stable/docs/reference/api/geopandas.GeoDataFrame.html) que tiene más de dos docenas de columnas de metadatos en una sola fila. La columna principal que nos interesa es la de `geometry`. Esta columna almacena las coordenadas de los vértices de un `MULTIPOLYGON`, es decir, un conjunto de polígonos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df.geometry
+```
+
+
+
+Los vértices de los polígonos parecen expresarse como pares de `(longitude, latitude)`. Podemos determinar qué sistema de coordenadas específico se utiliza examinando el atributo `crs` del `GeoDataFrame`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(shape_df.crs)
+```
+
+
+
+Utilicemos `hvplot` para generar un gráfico de este conjunto de datos vectoriales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df.hvplot()
+```
+
+
+
+La proyección en este gráfico es un poco extraña. La documentación de HoloViz incluye una [discusión sobre las consideraciones cuando se visualizan datos geográficos](https://hvplot.holoviz.org/user_guide/Geographic_Data.html). El punto importante a recordar en este contexto es que la opción `geo=True` es útil.
+
+Vamos a crear dos diccionarios de Python, `shapeplot_opts` y `layout_opts`, y construiremos una visualización.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts= dict(geo=True)
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+print(f"{shapeplot_opts=}")
+print(f"{layout_opts=}")
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+La documentación de HoloViz incluye una [discusión sobre las consideraciones cuando se grafican datos geográficos](https://hvplot.holoviz.org/user_guide/Geographic_Data.html). El punto importante que se debe recordar en el contexto inmediato es que la opción `geo=True` es útil.
+
+- Definir `color=None` significa que los polígonos no se rellenarán.
+- Al especificar `line_width` y `line_color` se modifica la apariencia de los límites de los polígonos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update( color=None ,
+ line_width=2,
+ line_color='red')
+print(shapeplot_opts)
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+Rellenemos los polígonos con color naranja utilizando la opción `color=orange` y hagamos que el área rellenada sea parcialmente transparente especificando un valor para `alpha` entre cero y uno.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update(color='orange', alpha=0.25)
+print(shapeplot_opts)
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+### Agregado de un mapa base
+
+
+
+A continuación, proporcionemos un mapa base y superpongamos los polígonos trazados utilizando el operador `*`. Observa el uso de paréntesis para aplicar el método `opts` a la salida del operador `*`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.ESRI(height=500, width=500, padding=0.1)
+
+(shape_df.hvplot(**shapeplot_opts) * basemap).opts(**layout_opts)
+```
+
+
+
+El mapa base no necesita ser superpuesto como un objeto separado, puede especificarse utilizando la palabra clave `tiles`. Por ejemplo, al establecer `tiles='OSM'` se utilizan los mosaicos de [OpenStreetMap](ttps://www.openstreetmap.org). Observa que las dimensiones de la imagen renderizada probablemente no sean las mismas que las de la imagen anterior con los mosaicos [ESRI](https://www.esri.com), ya que antes especificamos explícitamente la altura de 500, con la opción `height=500`, y el ancho de 500, con la opción `width=500`, en la definición del `basemap`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update(tiles='OSM')
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+Vamos a eliminar la opción `tiles` de `shapeplot_opts` y a vincular el objeto del gráfico resultante al identificador `shapeplot`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+del shapeplot_opts['tiles']
+print(shapeplot_opts)
+
+shapeplot = shape_df.hvplot(**shapeplot_opts)
+shapeplot
+```
+
+### Combinación de datos vectoriales con datos ráster en un gráfico estático
+
+
+
+Combinemos estos datos vectoriales con algunos datos ráster. Cargaremos los datos raster desde un archivo local utilizando la función `rioxarray.open_rasterio`. Por practicidad, usaremos el encadenamiento de métodos para reetiquetar las coordenadas del `DataArray` cargado y usaremos el método `squeeze` para convertir el arreglo tridimensional que se cargó en uno bidimensional.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = (
+ rio.open_rasterio(RASTER_FILE)
+ .rename(dict(x='longitude', y='latitude'))
+ .squeeze()
+ )
+raster
+```
+
+
+
+Utilizaremos un diccionario de Python `image_opts` para almacenar argumentos útiles y pasarlos a `hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ cmap='hot_r',
+ clim=(0, 100),
+ alpha=0.8,
+ project=True,
+ )
+
+raster.hvplot.image(**image_opts)
+```
+
+
+
+Podemos superponer `shapeplot` con los datos ráster graficados utilizando el operador `*`. Podemos utilizar las herramientas `Pan`, `Wheel Zoom` y `Box Zoom` en la barra de herramientas de Bokeh a la derecha del gráfico para hacer _zoom_ y comprobar que los datos vectoriales fueron trazados encima de los datos ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster.hvplot.image(**image_opts) * shapeplot
+```
+
+
+
+Además, podemos superponer los datos vectoriales y ráster en mosaicos ESRI utilizando `basemap`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster.hvplot.image(**image_opts) * shapeplot * basemap
+```
+
+
+
+Observa que muchos de los píxeles blancos oscurecen el gráfico. Resulta que estos píxeles corresponden a ceros en los datos ráster, los cuales pueden ser ignorados sin problema. Podemos filtrarlos utilizando el método `where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = raster.where((raster!=0))
+layout_opts.update(title="McKinney 2022 Wildfires")
+
+(raster.hvplot.image(**image_opts) * shapeplot * basemap).opts(**layout_opts)
+```
+
+***
+
+## Construcción de gráficos estáticos a partir de un arreglo 3D
+
+
+
+Vamos a cargar una secuencia de archivos ráster en un arreglo tridimensional y generaremos algunos gráficos. Para empezar, construiremos un bucle para leer `DataArrays` de los archivos `RASTER_FILES` y utilizaremos `xarray.concat` para generar un único arreglo de rásters tridimensional (es decir, tres rásters de $3,600\times3,600$ apilados verticalmente). Aprenderemos las interpretaciones específicas asociadas con el conjunto de datos ráster en un cuaderno computacional posterior. Por el momento, los trataremos como datos sin procesar con los que experimentaremos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = []
+for path in RASTER_FILES:
+ data = rio.open_rasterio(path).rename(dict(x='longitude', y='latitude'))
+ band_name = path.stem.split('_')[-1]
+ data.coords.update({'band': [band_name]})
+ data.attrs = dict(description=f"OPERA DIST product", units=None)
+ stack.append(data)
+
+stack = xr.concat(stack, dim='band')
+stack = stack.where(stack!=0)
+```
+
+
+
+Renombramos los ejes `longitude` y `latitude` y filtramos los píxeles con valor cero para simplificar la visualización posterior.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+
+
+Una vez que el `stack` de `DataArray` esté construido, podemos enfocarnos en la visualización.
+
+Si queremos generar un gráfico estático con varias imágenes, podemos utilizar `hvplot.image` junto con el método `.layout`. Para ver cómo funciona, empecemos por redefinir los diccionarios `image_opts` y `layout_opts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict( x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ cmap='hot_r',
+ crs=stack.rio.crs,
+ alpha=0.8,
+ project=True,
+ aspect='equal',
+ shared_axes=False,
+ colorbar=True,
+ tiles='ESRI',
+ padding=0.1)
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+```
+
+
+
+Para acelerar el renderizado, construiremos inicialmente un objeto `view` que seleccione subconjuntos de píxeles. Definimos inicialmente el parámetro `steps=200` para restringir la vista a cada 200 píxeles en cualquier dirección. Si reducimos los `steps`, se tarda más en renderizar. Establecer `steps=1` o `steps=None` equivale a seleccionar todos los píxeles.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+layout_opts.update(frame_width=250, frame_height=250)
+
+
+view = stack.isel(latitude=subset, longitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts).layout()
+```
+
+
+
+El método `layout` traza de manera predeterminada cada uno de los tres rásteres seleccionados a lo largo del eje `band` horizontalmente.
+
+
+
+***
+
+## Construcción de una vista dinámica a partir de un arreglo 3D
+
+
+
+Otra forma de visualizar un arreglo tridimensional es asociar un _widget_ de selección a uno de los ejes. Si llamamos a `hvplot.image` sin agregar el método `.layout` el resultado es un _mapa dinámico_. En este caso, el _widget_ de selección nos permite elegir cortes a lo largo del eje `band`.
+
+Una vez más, aumentar el valor del parámetro `steps` reduce el tiempo de renderizado. Reducirlo a `1` o a `None` renderiza a resolución completa.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+layout_opts.update(frame_height=400, frame_width=400)
+
+view = stack.isel(latitude=subset, longitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts)
+```
+
+
+
+Más adelante apilaremos muchos rásters con distintas marcas temporales a lo largo de un eje `time`. Cuando haya muchos cortes, el _widget_ de selección se renderizará como un deslizador en vez de como un menú desplegable. Podemos controlar la ubicación del _widget_ utilizando una opción de palabra clave `widget_location`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+view.hvplot.image(widget_location='bottom_left', **image_opts, **layout_opts)
+```
+
+
+
+Observa que agregar la opción `widget_location` modifica ligeramente la secuencia en la que se especifican las opciones. Es decir, si invocamos algo como
+
+```python
+view.hvplot.image(widget_location='top_left', **image_opts).opts(**layout_opts)
+```
+
+se genera una excepción (de ahí la invocación en la celda de código anterior). Hay algunas dificultades sutiles en la elaboración de la secuencia correcta para aplicar opciones al personalizar objetos HoloViz/Hvplot (principalmente debido a las formas en que los espacios de los nombres se superponen con Bokeh u otros _backends_ de renderizado). La mejor estrategia es empezar con el menor número de opciones posible y experimentar añadiendo opciones de forma incremental hasta que obtengamos la visualización final que deseamos.
+
+
+
+***
+
+### Combinación de datos vectoriales con datos ráster en una vista dinámica.
+
+
+
+Por último, vamos a superponer nuestros datos vectoriales anteriores, los límites del incendio forestal, con la vista dinámica de este arreglo tridimensional de rásteres. Podemos utilizar el operador `*` para combinar la salida de `hvplot.image` con `shapeplot`, la vista renderizada de los datos vectoriales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+view = stack.isel(latitude=subset, longitude=subset)
+
+image_opts.update(colorbar=False)
+layout_opts.update(frame_height=500, frame_width=500)
+(view.hvplot.image(**image_opts) * shapeplot).opts(**layout_opts)
+```
+
+
+
+Una vez más, la correcta especificación de las opciones puede requerir un poco de experimentación. Las ideas importantes que se deben tomar en cuenta aquí son:
+
+- cómo cargar conjuntos de datos relevantes con `geopandas` and `rioxarray`, y
+- cómo utilizar `hvplot` de forma interactiva e incremental para generar visualizaciones atractivas.
+
+
+
+***
diff --git a/book/es-419/03_Using_NASA_EarthData/01_Using_OPERA_DIST_Products.md b/book/es-419/03_Using_NASA_EarthData/01_Using_OPERA_DIST_Products.md
new file mode 100644
index 0000000..81569dc
--- /dev/null
+++ b/book/es-419/03_Using_NASA_EarthData/01_Using_OPERA_DIST_Products.md
@@ -0,0 +1,343 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.4
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Utilización de los productos OPERA DIST
+
+
+
+## El proyecto OPERA
+
+
+
+
+
+
+  +
+
+Del proyecto [Observational Products for End-Users from Remote Sensing Analysis (OPERA)](https://www.jpl.nasa.gov/go/opera) (en español, Productos de Observación para Usuarios Finales a partir del Analisis por Teledetección):
+
+> Iniciado en abril del 2021, el proyecto OPERA del _Jet Propulsion Laboratory_ (JPL) (en español, Laboratorio de Propulsión a Chorro) recopila datos satelitales ópticos y de radar para generar seis conjuntos de productos:
+>
+> - un conjunto de productos sobre la extensión de las aguas de la superficie terrestre a escala casi mundial
+> - un conjunto de productos de Alteraciones de la Superficie terrestre a escala casi mundial
+> - un producto con Corrección Radiométrica del Terreno a escala casi mundial
+> - un conjunto de productos _Coregistered Single Look Complex_ para Norteamérica
+> - un conjunto de productos de Desplazamiento para Norteamérica
+> - un conjunto de productos de Movimiento Vertical del Terreno en Norteamérica
+
+Es decir, OPERA es una iniciativa de la National Aeronautics and Space Administration (NASA, en español, Administración Nacional de Aeronáutica y del Espacio) que toma, por ejemplo, datos de teledetección óptica o radar recopilados desde satélites, y genera una variedad de conjuntos de datos preprocesados para uso público. Los productos de OPERA no son imágenes de satélite sin procesar, sino el resultado de una clasificación algorítmica para determinar, por ejemplo, qué regiones terrestres contienen agua o dónde se ha modificado la vegetación. Las imágenes de satélite sin procesar se recopilan a partir de mediciones realizadas por los instrumentos a bordo de las misiones de los satélites Sentinel-1 A/B, Sentinel-2 A/B y Landsat-8/9 (de ahí el término _Harmonized Landsat-Sentinel_" (HLS) (en español, Landsat-Sentinel Armonizadas) para en numerosas descripciones de productos).
+
+
+
+***
+
+## El producto OPERA _Land Surface Disturbance_ (DIST) (en español, Perturbación de la superficie terrestre)
+
+
+
+Uno de estos productos de datos de OPERA es el producto DIST (descrito con más detalle en la especificación del producto [OPERA DIST HLS](https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf)).
+Los productos DIST mapean la _perturbación de la vegetación_ (en concreto, la pérdida de cubierta vegetal por píxel HLS siempre que haya una disminución indicada) a partir de escenas armonizadas Landsat-8 y Sentinel-2 A/B (HLS). Una de las aplicaciones de estos datos es cuantificar los daños causados por los _incendios forestales_. El producto DIST_ALERT se publica a intervalos regulares (al igual que las imágenes HLS, aproximadamente cada 12 días en un determinado mosaico/región). El producto DIST_ANN resume las mediciones de las alteraciones a lo largo de un año.
+
+Los productos DIST cuantifican los datos de reflectancia de la superficie (RS) (en inglés, Surface Reflectance, SR) adquiridos a partir de imágenes terrestres operacionales _Operational Land Imager_ (OLI) (en español, Generador de Imágenes Terrestres Operacional) a bordo del satélite de teledetección Landsat-8 y del _Multi-Spectral Instrument_ (MSI) (en español, Instrumento Multiespectral) a bordo del satélite de teledetección Sentinel-2 A/B. Los productos de datos HLS DIST son archivos de tipo ráster, cada uno de ellos asociado a mosaicos de la superficie terrestre. Cada mosaico se representa mediante coordenadas cartográficas proyectadas alineadas con el [Sistema de Referencia de Cuadrículas Militares (MGRS, por sus siglas en inglés de _Military Grid Reference System_)](https://en.wikipedia.org/wiki/Military_Grid_Reference_System). Cada mosaico se divide en 3,660 filas y 3,660 columnas con un espaciado de píxeles de 30 metros (así que un mosaico es de $109.8\,\mathrm{km}$ largo en cada lado). Los mosaicos vecinos se solapan 4.900 metros en cada dirección (los detalles se describen detalladamente en la [especificación de producto DIST](https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf)).
+
+Los productos OPERA DIST se distribuyen como [GeoTIFFs optimizados para la nube](https://www.cogeo.org/); en la práctica, esto significa que las diferentes bandas se almacenan en archivos de formato TIFFs (TIFF, por sus siglas en inglés, _Tagged Image File Format_) distintos. La especificación TIFF permite el almacenamiento de matrices multidimensionales en un único archivo. El almacenamiento de bandas distintas en diferentes archivos TIFF permite que estos se descarguen de forma independiente.
+
+
+
+***
+
+
+
+## Banda 1: Valor máximo de la anomalía de pérdida de vegetación (VEG_ANOM_MAX)
+
+
+
+
+
+Examina un archivo local con un ejemplo de datos DIST-ALERT. El archivo contiene la primera banda de datos de alteración: la _anomalía de pérdida máxima de vegetación_. Para cada píxel, se trata de un valor entre 0% y 100% que representa la diferencia porcentual entre la cobertura vegetal que se observa actualmente y un valor de referencia histórico. Es decir, un valor de 100 corresponde a una pérdida total de vegetación en un píxel y un valor de 0 corresponde a que no hubo pérdida de vegetación. Los valores de los píxeles se almacenan como enteros sin signo de 8 bits (UInt8) porque los valores de los píxeles solo deben oscilar entre 0 y 100. Un valor del píxel de 255 indica que faltan datos, es decir, que los datos HLS no pudieron determinar un valor máximo de anomalía en la vegetación para ese píxel. Por supuesto, el uso de datos enteros sin signo de 8 bits es mucho más eficiente para el almacenamiento y para la transmisión de datos a través de una red (en comparación con, por ejemplo, datos de punto flotante de 32 o 64 bits).
+
+Empieza importando las librerías necesarias. Observa que también estamos importando la clase `FixedTicker` de la librería Bokeh para hacer que los gráficos interactivos sean un poco más atractivos.
+
+
+
+```{code-cell} python editable=true jupyter={"source_hidden": false} slideshow={"slide_type": ""}
+# Notebook dependencies
+import warnings
+warnings.filterwarnings('ignore')
+from pathlib import Path
+import rioxarray as rio
+import geoviews as gv
+gv.extension('bokeh')
+import hvplot.xarray
+from bokeh.models import FixedTicker
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+
+
+Lee los datos de un archivo local `'OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif'`. Antes de cargarlo, analiza los metadatos incluídos en el nombre del archivo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+filename = LOCAL_PATH.name
+print(filename)
+```
+
+
+
+Este nombre de archivo bastante largo incluye varios campos separados por caracteres de guión bajo (`_`). Podemos utilizar el método `str.split` de Python para ver más fácilmente los distintos campos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+filename.split('_') # Use the Python str.split method to view the distinct fields more easily.
+```
+
+
+
+Los archivos de los productos OPERA tienen un esquema de nombres particular (como se describe en la [especificación de producto DIST](https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf)). En la salida anterior, puedes extraer ciertos metadatos para este ejemplo:
+
+1. _Product_: `OPERA`;
+2. _Level_: `L3` ;
+3. _ProductType_: `DIST-ALERT-HLS` ;
+4. _TileID_: `T10TEM` (cadena de caracteres que hace referencia a un mosaico del [MGRS](https://en.wikipedia.org/wiki/Military_Grid_Reference_System));
+5. _AcquisitionDateTime_: `20220815T185931Z` (cadena que representa una marca de tiempo GMT para la adquisición de los datos);
+6. _ProductionDateTime_ : `20220817T153514Z` (cadena que representa una marca de tiempo GMT para cuando se generó el producto de los datos);
+7. _Sensor_: `S2A` (identificador del satélite que adquirió los datos sin procesar: `L8` (Landsat-8), `S2A` (Sentinel-2 A) o `S2B` (Sentinel-2 B);
+8. _Resolution_: `30` (por ejemplo, píxeles de longitud lateral $30\mathrm{m}$);
+9. _ProductVersion_: `v0.1` (versión del producto); y
+10. _LayerName_: `VEG-ANOM-MAX`
+
+Ten en cuenta que la NASA utiliza nomenclaturas convencionales como [Earthdata Search](https://search.earthdata.nasa.gov) para extraer datos significativos de los [_SpatioTemporal Asset Catalogs_ (STACs)](https://stacspec.org/) (en español, Catálogos de Activos Espaciales y Temporales). Más adelante se utilizarán estos campo— en particular _TileID_ y _LayerName_; para filtrar los resultados de la búsqueda antes de recuperar los datos remotos.
+
+
+
+
+
+Sube los datos de este archivo local en un `DataArray`, que es un tipo de dato de Xarray, utilizando `rioxarray.open_rasterio`. Reetiqueta las coordenadas adecuadamente y extrae el CRS (sistema de referencia de coordenadas).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = rio.open_rasterio(LOCAL_PATH)
+crs = data.rio.crs
+data = data.rename({'x':'longitude', 'y':'latitude', 'band':'band'}).squeeze()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+crs
+```
+
+
+
+Antes de generar un gráfico, crea un mapa base utilizando mosaicos [ESRI](https://es.wikipedia.org/wiki/Esri).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Creates basemap
+base = gv.tile_sources.ESRI.opts(width=750, height=750, padding=0.1)
+```
+
+
+
+También utiliza diccionarios para capturar la mayor parte de las opciones de trazado que utilizarás más adelante junto con `.hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ frame_width=500,
+ frame_height=500,
+ aspect='equal',
+ cmap='hot_r',
+ clim=(0, 100),
+ alpha=0.8
+ )
+layout_opts = dict(
+ xlabel='Longitude',
+ ylabel='Latitude'
+ )
+```
+
+
+
+Por último, usa el método `DataArray.where` para filtrar los píxeles que faltan y los que no vieron ningún cambio en la vegetación; estos valores de píxeles serán reasignados como `nan` por lo que serán transparentes cuando el raster sea visualizado. También modifica ligeramente las opciones de `image_opts` y `layout_opts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_anom_max = data.where((data>0) & (data!=255))
+image_opts.update(crs=data.rio.crs)
+layout_opts.update(title=f"VEG_ANOM_MAX")
+```
+
+
+
+Estos cambios permiten generar una visualización útil.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_anom_max.hvplot.image(**image_opts).opts(**layout_opts) * base
+```
+
+
+
+En el gráfico resultante, los píxeles blancos y amarillos corresponden a regiones en las que se ha producido cierta deforestación, pero no mucha. Por el contrario, los píxeles oscuros y negros corresponden a regiones que han perdido casi toda la vegetación.
+
+
+
+***
+
+## Banda 2: Fecha de alteración inicial de la vegetación (VEG_DIST_DATE)
+
+
+
+Los productos DIST-ALERT contienen varias bandas (tal como se resume en la [ especificación de productos DIST](https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf)). La segunda banda que se analiza es la _fecha de alteración inicial de la vegetación_ en el último año. Esta se almacena como un número entero de 16 bits (Int16).
+
+El archivo `OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-DIST-DATE.tif` se almacena localmente. La [especificación de productos DIST](\(https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf\)) describe cómo utilizar las convenciones para la denominación de archivos. Aquí destaca la _fecha y hora de adquisición_ `20220815T185931`, por ejemplo, casi las 7 p.m. (UTC) del 15 de agosto del 2022.
+
+Cargar y reetiqueta el `DataArray` como antes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-DIST-DATE.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+data = data.rename({'x':'longitude', 'y':'latitude', 'band':'band'}).squeeze()
+```
+
+
+
+En esta banda en particular, el valor 0 indica que no ha habido alteraciones en el último año y -1 es un valor que indica que faltan datos. Cualquier valor positivo es el número de días desde el 31 de diciembre del 2020 en los que se midió la primera alteración en ese píxel. Filtrar los valores no positivos y conserva estos valores significativos utilizando `DataArray.where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_date = data.where(data>0)
+```
+
+
+
+Examina el rango de valores numéricos en `veg_dist_date` utilizando DataArray.min`and`DataArray.max`. Ambos métodos ignorarán los píxeles que contengan `nan\` (por sus siglas en inglés de _Not-a-Number_) al calcular el mínimo y el máximo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+d_min, d_max = int(veg_dist_date.min().item()), int(veg_dist_date.max().item())
+print(f'{d_min=}\t{d_max=}')
+```
+
+
+
+En este caso, los datos relevantes se encuentran entre 247 y 592. Recuerda que se trata del número de días transcurridos desde el 31 de diciembre del 2020, cuando se observó la primera alteración en el último año. Dado que estos datos se adquirieron el 15 de agosto del 2022, los únicos valores posibles estarían entre 227 y 592 días. Así que debes recalibrar los colores en la visualización
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(
+ clim=(d_min,d_max),
+ crs=data.rio.crs
+ )
+layout_opts.update(title=f"VEG_DIST_DATE")
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_date.hvplot.image(**image_opts).opts(**layout_opts) * base
+```
+
+
+
+Con este mapa de colores, los píxeles más claros mostraron algunos signos de deforestación hace cerca de un año. Por el contrario, los píxeles negros mostraron deforestación por primera vez cerca del momento de adquisición de los datos. Por tanto, esta banda es útil para seguir el avance de los incendios forestales a medida que arrasan los bosques.
+
+
+
+***
+
+
+
+## Banda 3: Estado de alteración de la vegetación (VEG_DIST_STATUS)
+
+
+
+
+
+Por último, se analiza una tercera banda de la familia de productos DIST-ALERT denominada _estado de alteración de la vegetación_. Estos valores de píxel se almacenan como enteros de 8 bits sin signo. Solo hay 6 valores distintos almacenados:
+
+- **0:** Sin alteración
+- **1:** Alteración provisional (**primera detección**) con cambio en la cubierta vegetal < 50%
+- **2:** Alteración confirmada (**detección recurrente**) con cambio en la cubierta vegetal < 50%
+- **3:** Alteración provisional con cambio en la cobertura vegetal ≥ 50%
+- **4:** Alteración confirmada con cambio en la cobertura vegetal ≥ 50%
+- **255**: Datos no disponibles
+
+El valor de un píxel se marca como cambiado provisionalmente cuando la pérdida de la cobertura vegetal (alteración) es observada por primera vez por un satélite. Si el cambio se vuelve a notar en posteriores adquisiciones HLS sobre dicho píxel, entonces el píxel se marca como confirmado.
+
+
+
+
+
+Se puede usar un archivo local como ejemplo de esta capa/banda particular de los datos DIST-ALERT. El código es el mismo que el anterior, pero observa que:
+
+- los datos filtrados reflejan los valores de píxel significativos para esta capa (por ejemplo, `data>0` and `data<5`), y
+- los valores del mapa de colores se reasignan en consecuencia (es decir, de 0 a 4).
+-
+
+Observa el uso de `FixedTicker` en la definición de una barra de colores más adecuada para un mapa de color discreto (es decir, categórico).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-DIST-STATUS.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+data = data.rename({'x':'longitude', 'y':'latitude', 'band':'band'}).squeeze()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_status = data.where((data>0)&(data<5))
+image_opts.update(crs=data.rio.crs)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+layout_opts.update(
+ title=f"VEG_DIST_STATUS",
+ clim=(0,4),
+ colorbar_opts={'ticker': FixedTicker(ticks=[0, 1, 2, 3, 4])}
+ )
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_status.hvplot.image(**image_opts).opts(**layout_opts) * base
+```
+
+
+
+Este mapa de colores continuo no resalta correctamente las características de este gráfico. Una mejor opción sería un mapa de colores _categórico_. Se mostrará como hacerlo en el próximo cuaderno computacional (con los productos de datos OPERA DSWx).
+
+
+
+***
diff --git a/book/es-419/03_Using_NASA_EarthData/02_Using_OPERA_DSWx_Products.md b/book/es-419/03_Using_NASA_EarthData/02_Using_OPERA_DSWx_Products.md
new file mode 100644
index 0000000..fc46e75
--- /dev/null
+++ b/book/es-419/03_Using_NASA_EarthData/02_Using_OPERA_DSWx_Products.md
@@ -0,0 +1,303 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Utilización del producto OPERA DSWx
+
+
+
+
+
+
+
+Del proyecto [Observational Products for End-Users from Remote Sensing Analysis (OPERA)](https://www.jpl.nasa.gov/go/opera) (en español, Productos de Observación para Usuarios Finales a partir del Analisis por Teledetección):
+
+> Iniciado en abril del 2021, el proyecto OPERA del _Jet Propulsion Laboratory_ (JPL) (en español, Laboratorio de Propulsión a Chorro) recopila datos satelitales ópticos y de radar para generar seis conjuntos de productos:
+>
+> - un conjunto de productos sobre la extensión de las aguas de la superficie terrestre a escala casi mundial
+> - un conjunto de productos de Alteraciones de la Superficie terrestre a escala casi mundial
+> - un producto con Corrección Radiométrica del Terreno a escala casi mundial
+> - un conjunto de productos _Coregistered Single Look Complex_ para Norteamérica
+> - un conjunto de productos de Desplazamiento para Norteamérica
+> - un conjunto de productos de Movimiento Vertical del Terreno en Norteamérica
+
+Es decir, OPERA es una iniciativa de la National Aeronautics and Space Administration (NASA, en español, Administración Nacional de Aeronáutica y del Espacio) que toma, por ejemplo, datos de teledetección óptica o radar recopilados desde satélites, y genera una variedad de conjuntos de datos preprocesados para uso público. Los productos de OPERA no son imágenes de satélite sin procesar, sino el resultado de una clasificación algorítmica para determinar, por ejemplo, qué regiones terrestres contienen agua o dónde se ha modificado la vegetación. Las imágenes de satélite sin procesar se recopilan a partir de mediciones realizadas por los instrumentos a bordo de las misiones de los satélites Sentinel-1 A/B, Sentinel-2 A/B y Landsat-8/9 (de ahí el término _Harmonized Landsat-Sentinel_" (HLS) (en español, Landsat-Sentinel Armonizadas) para en numerosas descripciones de productos).
+
+
+
+***
+
+## El producto OPERA _Land Surface Disturbance_ (DIST) (en español, Perturbación de la superficie terrestre)
+
+
+
+Uno de estos productos de datos de OPERA es el producto DIST (descrito con más detalle en la especificación del producto [OPERA DIST HLS](https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf)).
+Los productos DIST mapean la _perturbación de la vegetación_ (en concreto, la pérdida de cubierta vegetal por píxel HLS siempre que haya una disminución indicada) a partir de escenas armonizadas Landsat-8 y Sentinel-2 A/B (HLS). Una de las aplicaciones de estos datos es cuantificar los daños causados por los _incendios forestales_. El producto DIST_ALERT se publica a intervalos regulares (al igual que las imágenes HLS, aproximadamente cada 12 días en un determinado mosaico/región). El producto DIST_ANN resume las mediciones de las alteraciones a lo largo de un año.
+
+Los productos DIST cuantifican los datos de reflectancia de la superficie (RS) (en inglés, Surface Reflectance, SR) adquiridos a partir de imágenes terrestres operacionales _Operational Land Imager_ (OLI) (en español, Generador de Imágenes Terrestres Operacional) a bordo del satélite de teledetección Landsat-8 y del _Multi-Spectral Instrument_ (MSI) (en español, Instrumento Multiespectral) a bordo del satélite de teledetección Sentinel-2 A/B. Los productos de datos HLS DIST son archivos de tipo ráster, cada uno de ellos asociado a mosaicos de la superficie terrestre. Cada mosaico se representa mediante coordenadas cartográficas proyectadas alineadas con el [Sistema de Referencia de Cuadrículas Militares (MGRS, por sus siglas en inglés de _Military Grid Reference System_)](https://en.wikipedia.org/wiki/Military_Grid_Reference_System). Cada mosaico se divide en 3,660 filas y 3,660 columnas con un espaciado de píxeles de 30 metros (así que un mosaico es de $109.8\,\mathrm{km}$ largo en cada lado). Los mosaicos vecinos se solapan 4.900 metros en cada dirección (los detalles se describen detalladamente en la [especificación de producto DIST](https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf)).
+
+Los productos OPERA DIST se distribuyen como [GeoTIFFs optimizados para la nube](https://www.cogeo.org/); en la práctica, esto significa que las diferentes bandas se almacenan en archivos de formato TIFFs (TIFF, por sus siglas en inglés, _Tagged Image File Format_) distintos. La especificación TIFF permite el almacenamiento de matrices multidimensionales en un único archivo. El almacenamiento de bandas distintas en diferentes archivos TIFF permite que estos se descarguen de forma independiente.
+
+
+
+***
+
+
+
+## Banda 1: Valor máximo de la anomalía de pérdida de vegetación (VEG_ANOM_MAX)
+
+
+
+
+
+Examina un archivo local con un ejemplo de datos DIST-ALERT. El archivo contiene la primera banda de datos de alteración: la _anomalía de pérdida máxima de vegetación_. Para cada píxel, se trata de un valor entre 0% y 100% que representa la diferencia porcentual entre la cobertura vegetal que se observa actualmente y un valor de referencia histórico. Es decir, un valor de 100 corresponde a una pérdida total de vegetación en un píxel y un valor de 0 corresponde a que no hubo pérdida de vegetación. Los valores de los píxeles se almacenan como enteros sin signo de 8 bits (UInt8) porque los valores de los píxeles solo deben oscilar entre 0 y 100. Un valor del píxel de 255 indica que faltan datos, es decir, que los datos HLS no pudieron determinar un valor máximo de anomalía en la vegetación para ese píxel. Por supuesto, el uso de datos enteros sin signo de 8 bits es mucho más eficiente para el almacenamiento y para la transmisión de datos a través de una red (en comparación con, por ejemplo, datos de punto flotante de 32 o 64 bits).
+
+Empieza importando las librerías necesarias. Observa que también estamos importando la clase `FixedTicker` de la librería Bokeh para hacer que los gráficos interactivos sean un poco más atractivos.
+
+
+
+```{code-cell} python editable=true jupyter={"source_hidden": false} slideshow={"slide_type": ""}
+# Notebook dependencies
+import warnings
+warnings.filterwarnings('ignore')
+from pathlib import Path
+import rioxarray as rio
+import geoviews as gv
+gv.extension('bokeh')
+import hvplot.xarray
+from bokeh.models import FixedTicker
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+
+
+Lee los datos de un archivo local `'OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif'`. Antes de cargarlo, analiza los metadatos incluídos en el nombre del archivo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+filename = LOCAL_PATH.name
+print(filename)
+```
+
+
+
+Este nombre de archivo bastante largo incluye varios campos separados por caracteres de guión bajo (`_`). Podemos utilizar el método `str.split` de Python para ver más fácilmente los distintos campos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+filename.split('_') # Use the Python str.split method to view the distinct fields more easily.
+```
+
+
+
+Los archivos de los productos OPERA tienen un esquema de nombres particular (como se describe en la [especificación de producto DIST](https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf)). En la salida anterior, puedes extraer ciertos metadatos para este ejemplo:
+
+1. _Product_: `OPERA`;
+2. _Level_: `L3` ;
+3. _ProductType_: `DIST-ALERT-HLS` ;
+4. _TileID_: `T10TEM` (cadena de caracteres que hace referencia a un mosaico del [MGRS](https://en.wikipedia.org/wiki/Military_Grid_Reference_System));
+5. _AcquisitionDateTime_: `20220815T185931Z` (cadena que representa una marca de tiempo GMT para la adquisición de los datos);
+6. _ProductionDateTime_ : `20220817T153514Z` (cadena que representa una marca de tiempo GMT para cuando se generó el producto de los datos);
+7. _Sensor_: `S2A` (identificador del satélite que adquirió los datos sin procesar: `L8` (Landsat-8), `S2A` (Sentinel-2 A) o `S2B` (Sentinel-2 B);
+8. _Resolution_: `30` (por ejemplo, píxeles de longitud lateral $30\mathrm{m}$);
+9. _ProductVersion_: `v0.1` (versión del producto); y
+10. _LayerName_: `VEG-ANOM-MAX`
+
+Ten en cuenta que la NASA utiliza nomenclaturas convencionales como [Earthdata Search](https://search.earthdata.nasa.gov) para extraer datos significativos de los [_SpatioTemporal Asset Catalogs_ (STACs)](https://stacspec.org/) (en español, Catálogos de Activos Espaciales y Temporales). Más adelante se utilizarán estos campo— en particular _TileID_ y _LayerName_; para filtrar los resultados de la búsqueda antes de recuperar los datos remotos.
+
+
+
+
+
+Sube los datos de este archivo local en un `DataArray`, que es un tipo de dato de Xarray, utilizando `rioxarray.open_rasterio`. Reetiqueta las coordenadas adecuadamente y extrae el CRS (sistema de referencia de coordenadas).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = rio.open_rasterio(LOCAL_PATH)
+crs = data.rio.crs
+data = data.rename({'x':'longitude', 'y':'latitude', 'band':'band'}).squeeze()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+crs
+```
+
+
+
+Antes de generar un gráfico, crea un mapa base utilizando mosaicos [ESRI](https://es.wikipedia.org/wiki/Esri).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Creates basemap
+base = gv.tile_sources.ESRI.opts(width=750, height=750, padding=0.1)
+```
+
+
+
+También utiliza diccionarios para capturar la mayor parte de las opciones de trazado que utilizarás más adelante junto con `.hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ frame_width=500,
+ frame_height=500,
+ aspect='equal',
+ cmap='hot_r',
+ clim=(0, 100),
+ alpha=0.8
+ )
+layout_opts = dict(
+ xlabel='Longitude',
+ ylabel='Latitude'
+ )
+```
+
+
+
+Por último, usa el método `DataArray.where` para filtrar los píxeles que faltan y los que no vieron ningún cambio en la vegetación; estos valores de píxeles serán reasignados como `nan` por lo que serán transparentes cuando el raster sea visualizado. También modifica ligeramente las opciones de `image_opts` y `layout_opts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_anom_max = data.where((data>0) & (data!=255))
+image_opts.update(crs=data.rio.crs)
+layout_opts.update(title=f"VEG_ANOM_MAX")
+```
+
+
+
+Estos cambios permiten generar una visualización útil.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_anom_max.hvplot.image(**image_opts).opts(**layout_opts) * base
+```
+
+
+
+En el gráfico resultante, los píxeles blancos y amarillos corresponden a regiones en las que se ha producido cierta deforestación, pero no mucha. Por el contrario, los píxeles oscuros y negros corresponden a regiones que han perdido casi toda la vegetación.
+
+
+
+***
+
+## Banda 2: Fecha de alteración inicial de la vegetación (VEG_DIST_DATE)
+
+
+
+Los productos DIST-ALERT contienen varias bandas (tal como se resume en la [ especificación de productos DIST](https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf)). La segunda banda que se analiza es la _fecha de alteración inicial de la vegetación_ en el último año. Esta se almacena como un número entero de 16 bits (Int16).
+
+El archivo `OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-DIST-DATE.tif` se almacena localmente. La [especificación de productos DIST](\(https://d2pn8kiwq2w21t.cloudfront.net/documents/OPERA_DIST_HLS_Product_Specification_V1.pdf\)) describe cómo utilizar las convenciones para la denominación de archivos. Aquí destaca la _fecha y hora de adquisición_ `20220815T185931`, por ejemplo, casi las 7 p.m. (UTC) del 15 de agosto del 2022.
+
+Cargar y reetiqueta el `DataArray` como antes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-DIST-DATE.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+data = data.rename({'x':'longitude', 'y':'latitude', 'band':'band'}).squeeze()
+```
+
+
+
+En esta banda en particular, el valor 0 indica que no ha habido alteraciones en el último año y -1 es un valor que indica que faltan datos. Cualquier valor positivo es el número de días desde el 31 de diciembre del 2020 en los que se midió la primera alteración en ese píxel. Filtrar los valores no positivos y conserva estos valores significativos utilizando `DataArray.where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_date = data.where(data>0)
+```
+
+
+
+Examina el rango de valores numéricos en `veg_dist_date` utilizando DataArray.min`and`DataArray.max`. Ambos métodos ignorarán los píxeles que contengan `nan\` (por sus siglas en inglés de _Not-a-Number_) al calcular el mínimo y el máximo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+d_min, d_max = int(veg_dist_date.min().item()), int(veg_dist_date.max().item())
+print(f'{d_min=}\t{d_max=}')
+```
+
+
+
+En este caso, los datos relevantes se encuentran entre 247 y 592. Recuerda que se trata del número de días transcurridos desde el 31 de diciembre del 2020, cuando se observó la primera alteración en el último año. Dado que estos datos se adquirieron el 15 de agosto del 2022, los únicos valores posibles estarían entre 227 y 592 días. Así que debes recalibrar los colores en la visualización
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(
+ clim=(d_min,d_max),
+ crs=data.rio.crs
+ )
+layout_opts.update(title=f"VEG_DIST_DATE")
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_date.hvplot.image(**image_opts).opts(**layout_opts) * base
+```
+
+
+
+Con este mapa de colores, los píxeles más claros mostraron algunos signos de deforestación hace cerca de un año. Por el contrario, los píxeles negros mostraron deforestación por primera vez cerca del momento de adquisición de los datos. Por tanto, esta banda es útil para seguir el avance de los incendios forestales a medida que arrasan los bosques.
+
+
+
+***
+
+
+
+## Banda 3: Estado de alteración de la vegetación (VEG_DIST_STATUS)
+
+
+
+
+
+Por último, se analiza una tercera banda de la familia de productos DIST-ALERT denominada _estado de alteración de la vegetación_. Estos valores de píxel se almacenan como enteros de 8 bits sin signo. Solo hay 6 valores distintos almacenados:
+
+- **0:** Sin alteración
+- **1:** Alteración provisional (**primera detección**) con cambio en la cubierta vegetal < 50%
+- **2:** Alteración confirmada (**detección recurrente**) con cambio en la cubierta vegetal < 50%
+- **3:** Alteración provisional con cambio en la cobertura vegetal ≥ 50%
+- **4:** Alteración confirmada con cambio en la cobertura vegetal ≥ 50%
+- **255**: Datos no disponibles
+
+El valor de un píxel se marca como cambiado provisionalmente cuando la pérdida de la cobertura vegetal (alteración) es observada por primera vez por un satélite. Si el cambio se vuelve a notar en posteriores adquisiciones HLS sobre dicho píxel, entonces el píxel se marca como confirmado.
+
+
+
+
+
+Se puede usar un archivo local como ejemplo de esta capa/banda particular de los datos DIST-ALERT. El código es el mismo que el anterior, pero observa que:
+
+- los datos filtrados reflejan los valores de píxel significativos para esta capa (por ejemplo, `data>0` and `data<5`), y
+- los valores del mapa de colores se reasignan en consecuencia (es decir, de 0 a 4).
+-
+
+Observa el uso de `FixedTicker` en la definición de una barra de colores más adecuada para un mapa de color discreto (es decir, categórico).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-DIST-STATUS.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+data = data.rename({'x':'longitude', 'y':'latitude', 'band':'band'}).squeeze()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_status = data.where((data>0)&(data<5))
+image_opts.update(crs=data.rio.crs)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+layout_opts.update(
+ title=f"VEG_DIST_STATUS",
+ clim=(0,4),
+ colorbar_opts={'ticker': FixedTicker(ticks=[0, 1, 2, 3, 4])}
+ )
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_status.hvplot.image(**image_opts).opts(**layout_opts) * base
+```
+
+
+
+Este mapa de colores continuo no resalta correctamente las características de este gráfico. Una mejor opción sería un mapa de colores _categórico_. Se mostrará como hacerlo en el próximo cuaderno computacional (con los productos de datos OPERA DSWx).
+
+
+
+***
diff --git a/book/es-419/03_Using_NASA_EarthData/02_Using_OPERA_DSWx_Products.md b/book/es-419/03_Using_NASA_EarthData/02_Using_OPERA_DSWx_Products.md
new file mode 100644
index 0000000..fc46e75
--- /dev/null
+++ b/book/es-419/03_Using_NASA_EarthData/02_Using_OPERA_DSWx_Products.md
@@ -0,0 +1,303 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Utilización del producto OPERA DSWx
+
+
+
+
+  +
+
+El proyecto de Productos de Observación para Usuarios Finales a partir del Análisis por Teledetección (OPERA, por las siglas en inglés de _Observational Products for End-Users from Remote Sensing Analysis_).
+
+> Iniciado en abril del 2021, el proyecto OPERA del _Jet Propulsion Laboratory_ (JPL) (en español, Laboratorio de Propulsión a Chorro) recopila datos de instrumentos satelitales ópticos y de radar para generar seis conjuntos de productos:
+>
+> - un conjunto de productos sobre la Extensión del Agua de la Superficie terrestre a escala casi mundial
+> - un conjunto de productos de Alteraciones de la Superficie terrestre a escala casi mundial
+> - un producto con Corrección Radiométrica del Terreno a escala casi mundial
+> - un conjunto de productos _Coregistered Single Look xomplex_ para Norteamérica
+> - un conjunto de productos de Desplazamiento para Norteamérica
+> - un conjunto de productos de Movimiento Vertical del Terreno en Norteamérica
+
+Es decir, OPERA es una iniciativa de la _National Aeronautics and Space Administration_ (NASA)(en español, Administración Nacional de Aeronáutica y del Espacio) que toma, por ejemplo, datos de teledetección óptica o radar recopilados desde satélites, y genera una variedad de conjuntos de datos preprocesados para uso público. Los productos de OPERA no son imágenes de satélite sin procesar, sino el resultado de una clasificación algorítmica para determinar, por ejemplo, qué regiones terrestres contienen agua o dónde se ha desplazado la vegetación. Las imágenes de satélite sin procesar se recopilan a partir de mediciones realizadas por los instrumentos a bordo de las misiones de los satélites Sentinel-1 A/B, Sentinel-2 A/B y Landsat-8/9 (de ahí el término _HLS_ por las siglas en inglés de "_Harmonized Landsat-Sentinel_" (en español, armonización Landsat-Sentinel) en numerosas descripciones de productos).
+
+
+
+---
+
+## El producto OPERA _Dynamic Surface Water Extent_ (DSWx) (en español, Extensión de aguas superficiales dinámicas)
+
+
+
+Ya vimos la familia DIST (es decir, alteración de la superficie terrestre) con productos de datos OPERA. En este cuaderno computacional, analizaremos otro producto de datos OPERA: el producto _Dynamic Surface Water Extent (DSWx)_ (en español, Extensión de Aguas Superficiales Dinámicas). Este producto de datos resume la extensión de las aguas continentales (es decir, el agua en las masas de tierra en contraposición a la parte de un océano) que se puede utilizar para hacer un seguimiento de las inundaciones y las sequías. El producto DSWx se describe detalladamente en la [especificación del producto OPERA DSWx HLS](https://d2pn8kiwq2w21t.cloudfront.net/documents/ProductSpec_DSWX_URS309746.pdf).
+
+Los productos de datos DSWx se generan a partir de las mediciones de reflectancia superficial (RS) del HLS, específicamente, estas son efectuadas por el _Operational Land Imager_ (OLI) (en español,
+Generador de imágenes terrestres operacional) a bordo del satélite Landsat 8, el OLI-2 a bordo del satélite Landsat 9, y el MultiSpectral Instrument (MSI) (en español, Instrumento multiespectral) a bordo de los satélites Sentinel-2A/B. Al igual que los productos DIST, los productos DSWx consisten en datos ráster almacenados en formato GeoTIFF que utilizan el [_Military Grid Reference System_ (MGRS)](https://en.wikipedia.org/wiki/Military_Grid_Reference_System) (en español, Sistema de Referencia de Cuadrículas Militares) (los detalles se describen en la especificación del [producto DSWx](https://d2pn8kiwq2w21t.cloudfront.net/documents/ProductSpec_DSWX_URS309746.pdf). De nuevo, los productos OPERA DSWx se distribuyen como [GeoTIFF optimizados para la nube](https://www.cogeo.org/) que almacenan diferentes bandas/capas en archivos TIFF distintos.
+
+
+
+---
+
+
+
+## Banda 1: Clasificación del agua (WTR, por las abreviación en inglés de la palabra _Water_)
+
+
+
+Hay diez bandas o capas asociadas al producto de datos DSWx. En este tutorial, nos enfocaremos estrictamente en la primera banda—la capa de _clasificación del agua (WTR)_—pero los detalles de todas las bandas se dan en la [especificación del producto DSWx](https://d2pn8kiwq2w21t.cloudfront.net/documents/ProductSpec_DSWX_URS309746.pdf). Por ejemplo, la banda 3 es la _Capa de Confianza (CONF)_ que proporciona, para cada píxel, valores cuantitativos que describen el grado de confianza en las categorías dadas en la banda 1 (la Capa de clasificación del agua). La banda 4 es una capa de _Diagnóstico (DIAG)_ que codifica, para cada píxel, cuál de las cinco pruebas resultó positiva para obtener el valor del píxel correspondiente en la capa CONF.
+
+La capa de clasificación del agua consiste en datos ráster enteros de 8 bits sin signo (UInt8) que representan si un píxel contiene aguas continentales (por ejemplo, parte de un embalse, un lago, un río, etc., pero no agua asociada con el mar abierto). Los valores de esta capa ráster se calculan a partir de las imágenes crudas adquiridas por el satélite, asignando a los píxeles uno de los 7 valores enteros positivos que analizaremos a continuación.
+
+---
+
+### Análisis de una capa WTR de ejemplo
+
+Comencemos importando las librerías necesarias y cargando un archivo adecuado en un Xarray `DataArray`. El archivo en cuestión contiene datos ráster relativos al [embalse del lago Powelr](https://en.wikipedia.org/wiki/Lake_Powell), en el río Colorado, en los Estados Unidos.
+
+```{code-cell} python
+import warnings
+warnings.filterwarnings('ignore')
+from pathlib import Path
+import numpy as np, pandas as pd, xarray as xr
+import rioxarray as rio
+```
+
+```{code-cell} python
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+gv.extension('bokeh')
+from bokeh.models import FixedTicker
+```
+
+```{code-cell} python
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DSWx-HLS_T12SVG_20230411T180222Z_20230414T030945Z_L8_30_v1.0_B01_WTR.tif')
+b01_wtr = rio.open_rasterio(LOCAL_PATH).rename({'x':'longitude', 'y':'latitude'}).squeeze()
+```
+
+Recuerda que usar el `repr` para `b01_wtr` en este cuaderno computacional Jupyter es bastante conveniente.
+
+- Al desplegar la pestaña `Attributes`, podemos ver todos los metadatos asociados a los datos adquiridos.
+- Al desplegar la pestaña `Coordinates`, podemos analizar todos los arreglos asociados a esos valores de coordenadas.
+
+```{code-cell} python
+# Examine data
+b01_wtr
+```
+
+Vamos a examinar la distribución de los valores del píxel en `b01_wtr` utilizando el método Pandas `Series.value_counts`.
+
+```{code-cell} python
+counts = pd.Series(b01_wtr.values.flatten()).value_counts().sort_index()
+display(counts)
+```
+
+Estos valores de píxel son _datos categóricos_. En específico, los valores de píxel válidos y sus significados—según la [especificación del producto DSWx](https://d2pn8kiwq2w21t.cloudfront.net/documents/ProductSpec_DSWX_URS309746.pdf) —son los siguientes:
+
+- **0**: Sin agua—cualquier área con datos de reflectancia válidos que no sea de una de las otras categorías permitidas (agua abierta, agua superficial parcial, nieve/hielo, nube/sombra de nube, o océano enmascarado).
+- **1**: Agua abierta—cualquier píxel que sea completamente agua sin obstrucciones para el sensor, incluyendo obstrucciones por vegetación, terreno y edificios.
+- **2**: Agua parcialmente superficial—un área que es por lo menos 50% y menos de 100% agua abierta (por ejemplo, sumideros inundados, vegetación flotante, y píxeles bisecados por líneas costeras).
+- **252**: Nieve/Hielo.
+- **253**: Nube o sombra de nube—un área oscurecida por, o adyacente a, una nube o sombra de nube.
+- **254**: Océano enmascarado—un área identificada como océano utilizando una base de datos de la línea costera con un margen añadido.
+- **255**: Valor de relleno (datos faltantes).
+
+---
+
+### Realización de una primera visualización
+
+Hagamos un primer gráfico aproximado de los datos ráster utilizando `hvplot.image`. Como de costumbre, instanciamos un objeto `view` que corta un subconjunto más pequeño de píxeles para hacer que la imagen se renderice rápidamente.
+
+```{code-cell} python
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ project=True
+ )
+layout_opts = dict(
+ xlabel='Longitude',
+ ylabel='Latitude',
+ aspect='equal',
+ )
+
+steps = 100
+subset = slice(0, None, steps)
+view = b01_wtr.isel(longitude=subset, latitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts)
+```
+
+El mapa de colores predeterminado no revela muy bien las características ráster. Además, observa que el eje de la barra de colores cubre el rango numérico de 0 a 255 (aproximadamente), aunque la mayoría de esos valores de los píxeles (es decir, de `3` a `251`) no aparecen realmente en los datos. Anotar una imagen ráster de datos categóricos con una leyenda puede tener más sentido que utilizar una barra de colores. Sin embargo, actualmente, `hvplot.image` no soporta el uso de una leyenda. Por lo tanto, para este tutorial, nos limitaremos a utilizar una barra de colores. Antes de asignar un mapa de colores y etiquetas apropiadas para la barra de colores, tiene sentido limpiar los valores de los píxeles.
+
+---
+
+### Reasignación de los valores de los píxeles
+
+Queremos reasignar los valores ráster de los píxeles a un rango más estrecho (por ejemplo, de `0` a `5` en vez de `0` a `255`) para crear una barra de color sensible. Para ello, empezaremos copiando los valores del `DataArray` `b01_wtr` en otro llamado `new_data`, y creando un arreglo llamado `values` para contener el rango completo de valores de píxel permitidos.
+
+```{code-cell} python
+new_data = b01_wtr.copy(deep=True)
+values = np.array([0, 1, 2, 252, 253, 254, 255], dtype=np.uint8)
+print(values)
+```
+
+Primero debemos decidir cómo tratar los datos que faltan, es decir, los píxeles con el valor `255` en este ráster. Vamos a elegir tratar los píxeles de datos que faltan igual que los píxeles `"Sin agua"`. Podemos utilizar el método `DataArray.where` para reasignar los píxeles con valor `null_val` (por ejemplo, 255 en la celda de código siguiente) al valor de reemplazo `transparent_val` (por ejemplo, `0` en este caso). Anticipando que incluiremos este código en una función más adelante, incluimos el cálculo en un bloque `if` condicionado a un valor booleano `replace_null`.
+
+```{code-cell} python
+null_val = 255
+transparent_val = 0
+replace_null = True
+if replace_null:
+ new_data = new_data.where(new_data!=null_val, other=transparent_val)
+ values = values[np.where(values!=null_val)]
+
+print(np.unique(new_data.values))
+```
+
+Observa que `values` ya no incluye `null_val`. A continuación, instanciamos un arreglo `new_values` para almacenar los valores de píxel de reemplazo.
+
+```{code-cell} python
+n_values = len(values)
+start_val = 0
+new_values = np.arange(start=start_val, stop=start_val+n_values, dtype=values.dtype)
+assert transparent_val in new_values, f"{transparent_val=} not in {new_values}"
+print(values)
+print(new_values)
+```
+
+Ahora combinamos `values` y `new_values` en un diccionario `relabel`, y usamos el diccionario para modificar las entradas de `new_data`.
+
+```{code-cell} python
+relabel = dict(zip(values, new_values))
+for old, new in relabel.items():
+ if new==old: continue
+ new_data = new_data.where(new_data!=old, other=new)
+```
+
+Podemos encapsular la lógica de las celdas anteriores en una función de utilidad `relabel_pixels` que condensa un amplio rango de valores categóricos de píxeles en uno más ajustado que se mostrará mejor con un mapa de colores.
+
+```{code-cell} python
+# utility to remap pixel values to a sequence of contiguous integers
+def relabel_pixels(data, values, null_val=255, transparent_val=0, replace_null=True, start=0):
+ """
+ This function accepts a DataArray with a finite number of categorical values as entries.
+ It reassigns the pixel labels to a sequence of consecutive integers starting from start.
+ data: Xarray DataArray with finitely many categories in its array of values.
+ null_val: (default 255) Pixel value used to flag missing data and/or exceptions.
+ transparent_val: (default 0) Pixel value that will be fully transparent when rendered.
+ replace_null: (default True) Maps null_value->transparent_value everywhere in data.
+ start: (default 0) starting range of consecutive integer values for new labels.
+ The values returned are:
+ new_data: Xarray DataArray containing pixels with new values
+ relabel: dictionary associating old pixel values with new pixel values
+ """
+ new_data = data.copy(deep=True)
+ if values:
+ values = np.sort(np.array(values, dtype=np.uint8))
+ else:
+ values = np.sort(np.unique(data.values.flatten()))
+ if replace_null:
+ new_data = new_data.where(new_data!=null_val, other=transparent_val)
+ values = values[np.where(values!=null_val)]
+ n_values = len(values)
+ new_values = np.arange(start=start, stop=start+n_values, dtype=values.dtype)
+ assert transparent_val in new_values, f"{transparent_val=} not in {new_values}"
+ relabel = dict(zip(values, new_values))
+ for old, new in relabel.items():
+ if new==old: continue
+ new_data = new_data.where(new_data!=old, other=new)
+ return new_data, relabel
+```
+
+Apliquemos la función que acabamos de definir a `b01_wtr` y comprobemos que los valores de los píxeles cambiaron como queríamos.
+
+```{code-cell} python
+values = [0, 1, 2, 252, 253, 254, 255]
+print(f"Before applying relabel_pixels: {np.unique(b01_wtr.values)}")
+print(f"Original pixel values expected: {values}")
+b01_wtr, relabel = relabel_pixels(b01_wtr, values=values)
+print(f"After applying relabel_pixels: {np.unique(b01_wtr.values)}")
+```
+
+Observe que el valor de píxel `5` no aparece en el arreglo reetiquetada porque el valor de píxel `254` (para los píxeles de "Océano enmascarado") no aparece en el archivo GeoTIFF original. Esto está bien. El código escrito a continuación seguirá produciendo toda la variedad de posibles valores para los píxeles (y colores) en su barra de colores.
+
+---
+
+### Definición de un mapa de color y trazado con una barra de color
+
+Ahora estamos listos para definir un mapa de colores. Definimos el diccionario `COLORS` de forma que las etiquetas de píxel de `new_values` sean las claves del diccionario y algunas tuplas de color _Red Green Blue Alpha_ (RGBA) (en español, rojo verde azul alfa) utilizadas frecuentemente para este tipo de datos sean los valores del diccionario. Utilizaremos variantes del código de la siguiente celda para actualizar `layout_opts` de forma que los gráficos generados para varias capas/bandas de los productos de datos DSWx tengan leyendas adecuadas.
+
+```{code-cell} python
+COLORS = {
+0: (255, 255, 255, 0.0), # No Water
+1: (0, 0, 255, 1.0), # Open Water
+2: (180, 213, 244, 1.0), # Partial Surface Water
+3: ( 0, 255, 255, 1.0), # Snow/Ice
+4: (175, 175, 175, 1.0), # Cloud/Cloud Shadow
+5: ( 0, 0, 127, 0.5), # Ocean Masked
+}
+
+c_labels = ["No Water", "Open Water", "Partial Surface Water", "Snow/Ice", "Cloud/Cloud Shadow", "Ocean Masked"]
+c_ticks = list(COLORS.keys())
+limits = (c_ticks[0]-0.5, c_ticks[-1]+0.5)
+
+print(c_ticks)
+print(c_labels)
+```
+
+Para utilizar este mapa de colores, estas marcas y estas etiquetas en una barra de colores, creamos un diccionario `c_bar_opts` que contiene los objetos que hay que pasar al motor de renderizado Bokeh.
+
+```{code-cell} python editable=true slideshow={"slide_type": ""}
+c_bar_opts = dict( ticker=FixedTicker(ticks=c_ticks),
+ major_label_overrides=dict(zip(c_ticks, c_labels)),
+ major_tick_line_width=0, )
+```
+
+Debemos actualizar los diccionarios `image_opts` y `layout_opts` para incluir los datos relevantes para el mapa de colores.
+
+```{code-cell} python editable=true slideshow={"slide_type": ""}
+image_opts.update({ 'cmap': list(COLORS.values()),
+ 'clim': limits,
+ 'colorbar': True
+ })
+
+layout_opts.update(dict(title='B01_WTR', colorbar_opts=c_bar_opts))
+```
+
+Finalmente, podemos renderizar un gráfico rápido para asegurarnos de que la barra de colores se produce con etiquetas adecuadas.
+
+```{code-cell} python
+steps = 100
+subset = slice(0, None, steps)
+view = b01_wtr.isel(longitude=subset, latitude=subset)
+view.hvplot.image( **image_opts).opts(frame_width=500, frame_height=500, **layout_opts)
+```
+
+Por último, podemos definir un mapa base, esta vez utilizando mosaicos de [ESRI](https://www.esri.com). Esta vez, graficaremos el ráster a resolución completa (es decir, no nos molestaremos en utilizar `isel` para seleccionar primero un corte de menor resolución del ráster).
+
+```{code-cell} python editable=true slideshow={"slide_type": ""}
+# Creates basemap
+base = gv.tile_sources.EsriTerrain.opts(padding=0.1, alpha=0.25)
+b01_wtr.hvplot(**image_opts).opts(**layout_opts) * base
+```
+
+
+
+Este cuaderno computacional proporciona una visión general de cómo visualizar datos extraídos de productos de datos OPERA DSWx almacenados localmente. Ahora estamos listos para automatizar la búsqueda de dichos productos en la nube utilizando la API PySTAC.
+
+----
+
+
diff --git a/book/es-419/03_Using_NASA_EarthData/03_Using_PySTAC.md b/book/es-419/03_Using_NASA_EarthData/03_Using_PySTAC.md
new file mode 100644
index 0000000..8cf249a
--- /dev/null
+++ b/book/es-419/03_Using_NASA_EarthData/03_Using_PySTAC.md
@@ -0,0 +1,575 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Uso de la API de PySTAC
+
+
+
+En el sitio web [Earthdata Search](https://search.earthdata.nasa.gov) de la NASA se puede buscar una gran cantidad de datos. El enlace anterior se conecta a una interfaz gráfica de usuario (GUI, por sus siglas en inglés de _Graphical User Interface_) para buscar en los [catálogos activos espaciotemporales (STACs, por sus siglas en inglés de _SpatioTemporal Asset Catalogs_)](https://stacspec.org/) al especificar un área de interés (AOI, por sus siglas en inglés de _Area of Interest_) y una _ventana temporal_ o un _intervalo de fechas_.
+
+En pos de la reproducibilidad, se busca que las personas usuarias sean capaces de buscar en los catálogos de activos de manera programática. Aquí es donde entra en juego la librería [PySTAC](https://pystac.readthedocs.io/en/stable/).
+
+
+
+***
+
+## Esquema de las etapas del análisis
+
+
+
+- Identificación de los parámetros de búsqueda
+ - AOI, ventana temporal
+ - Endpoint, proveedor, identificador del catálogo ("nombre corto")
+- Obtención de los resultados de la búsqueda
+ - Exploración de datos, análisis para identificar características, bandas de interés
+ - Almacenar los resultados en un DataFrame para facilitar la exploración
+- Explorar y refinar los resultados de la búsqueda
+ - Identificar los granos de mayor valor
+ - Filtrar los granos anómalos con una contribución mínima
+ - Combinar los granos filtrados correspondientes en un DataFrame
+ - Identificar el tipo de salida que se quiere obtener
+- Procesamiento de los datos para generar resultados relevantes
+ - Descargar los granos relevantes en un tipo de dato DataArray de la libreria Xarray, apilados adecuadamente
+ - Realizar los cálculos intermedios necesarios
+ - Integrar los fragmentos de datos relevantes en una visualización
+
+
+
+***
+
+## Identificar los parámetros de búsqueda
+
+### Definir el AOI y el rango de fechas
+
+
+
+Comenzaremos tomando en cuenta un ejemplo concreto. [Las fuertes lluvias afectaron gravemente al sureste de Texas en mayo de 2024](https://www.texastribune.org/2024/05/03/texas-floods-weather-harris-county/), provocando [inundaciones y causando importantes daños materiales y humanos](https://www.texastribune.org/series/east-texas-floods-2024/).
+
+Como es usual, se requiere la importación de ciertas librerías relevantes. Las dos primeras celdas son familiares (relacionadas con las herramientas de análisis y la visualización de los datos que ya se examinaron). La tercera celda incluye la importación de la biblioteca `pystac_client` y de la biblioteca `gdal`, seguidas de algunos ajustes necesarios para utilizar la [Biblioteca de Abstracción de Datos Geoespaciales (GDAL, por sus siglas en inglés, Geospatial Data Abstraction Library)](https://gdal.org). Estos detalles en la configuración permiten que las sesiones de tu cuaderno computacional interactúen sin problemas con las fuentes remotas de datos geoespaciales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+# data wrangling imports
+import numpy as np
+import pandas as pd
+import xarray as xr
+import rioxarray as rio
+import rasterio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas
+import hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# STAC imports to retrieve cloud data
+from pystac_client import Client
+from osgeo import gdal
+# GDAL setup for accessing cloud data
+gdal.SetConfigOption('GDAL_HTTP_COOKIEFILE','~/.cookies.txt')
+gdal.SetConfigOption('GDAL_HTTP_COOKIEJAR', '~/.cookies.txt')
+gdal.SetConfigOption('GDAL_DISABLE_READDIR_ON_OPEN','EMPTY_DIR')
+gdal.SetConfigOption('CPL_VSIL_CURL_ALLOWED_EXTENSIONS','TIF, TIFF')
+```
+
+
+
+A continuación, definiremos los parámetros de búsqueda geográfica para poder recuperar los datos correspondientes a ese evento de inundación. Esto consiste en especificar un _AOI_ y un _intervalo de fechas_.
+
+- El AOI se especifica como un rectángulo de coordenadas de longitud-latitud en una única 4-tupla con la forma
+ $$({\mathtt{longitude}}_{\mathrm{min}},{\mathtt{latitude}}_{\mathrm{min}},{\mathtt{longitude}}_{\mathrm{max}},{\mathtt{latitude}}_{\mathrm{max}}),$$
+ por ejemplo, las coordenadas de la esquina inferior izquierda seguidas de las coordenadas de la esquina superior derecha.
+- El intervalo de fechas se especifica como una cadena de la forma
+ $${\mathtt{date}_{\mathrm{start}}}/{\mathtt{date}_{\mathrm{end}}},$$
+ donde las fechas se especifican en el formato estándar `YYYY-MM-DD`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Center of the AOI
+livingston_tx_lonlat = (-95.09,30.69) # (lon, lat) form
+```
+
+
+
+Escribiremos algunas funciones cortas para encapsular la lógica de nuestros flujos de trabajo genéricos. Para el código de investigación, estas se colocarían en archivos de Python. Por practicidad, incrustaremos las funciones en este cuaderno y en otros para que puedan ejecutarse correctamente con dependencias mínimas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle with given center of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding-box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to plot an AOI or bounding-box
+def plot_bbox(bbox):
+ '''Given bounding-box, returns GeoViews plot of Rectangle & Point at center
+ + bbox: bounding-box specified as (lon_min, lat_min, lon_max, lat_max)
+ Assume longitude-latitude coordinates.
+ '''
+ # These plot options are fixed but can be over-ridden
+ point_opts = opts.Points(size=12, alpha=0.25, color='blue')
+ rect_opts = opts.Rectangles(line_width=0, alpha=0.1, color='red')
+ lon_lat = (0.5*sum(bbox[::2]), 0.5*sum(bbox[1::2]))
+ return (gv.Points([lon_lat]) * gv.Rectangles([bbox])).opts(point_opts, rect_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+AOI = make_bbox(livingston_tx_lonlat, 0.5, 0.25)
+basemap = gv.tile_sources.OSM.opts(width=500, height=500)
+plot_bbox(AOI) * basemap
+```
+
+
+
+Agreguemos un intervalo de fechas. Las inundaciones ocurrieron principalmente entre el 30 de abril y el 2 de mayo. Estableceremos una ventana temporal más larga que cubra los meses de abril y mayo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+start_date, stop_date = '2024-04-01', '2024-05-31'
+DATE_RANGE = f'{start_date}/{stop_date}'
+```
+
+
+
+Por último, se crea un un diccionario `search_params` que almacene el AOI y el intervalo de fechas. Este diccionario se utilizará para buscar datos en los STACs.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+search_params = dict(bbox=AOI, datetime=DATE_RANGE)
+print(search_params)
+```
+
+***
+
+## Obtención de los resultados de búsqueda
+
+### Ejecución de una búsqueda con la API PySTAC
+
+
+
+Para iniciar una búsqueda de datos se necesitan tres datos más: el _Endpoint_ (una URL), el _Proveedor_ (una cadena que representa una ruta que extiende el _Endpoint_) y los _Identificadores de la colección_ (una lista de cadenas que hacen referencia a catálogos específicos). Generalmente, debemos probar con el [sitio web de Earthdata Search](https://search.earthdata.nasa.gov) de la NASA para determinar correctamente los valores para los productos de datos específicos que queremos recuperar. El [repositorio de GitHub de la NASA CMR STAC también supervisa los problemas](https://github.com/nasa/cmr-stac/issues) relacionados con la API para las consultas de búsqueda de EarthData Cloud.
+
+Para la búsqueda de productos de datos DSWx que se quiere ejecutar, estos parámetros son los que se definen en la siguiente celda de código.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+ENDPOINT = 'https://cmr.earthdata.nasa.gov/stac' # base URL for the STAC to search
+PROVIDER = 'POCLOUD'
+COLLECTIONS = ["OPERA_L3_DSWX-HLS_V1_1.0"]
+# Update the dictionary opts with list of collections to search
+search_params.update(collections=COLLECTIONS)
+print(search_params)
+```
+
+
+
+Una vez que se definieron los parámetros de búsqueda en el diccionario de Python `search_params`, se puede instanciar un `Cliente` y buscar en el catálogo espacio-temporal de activos utilizando el método `Client.search`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+catalog = Client.open(f'{ENDPOINT}/{PROVIDER}/')
+search_results = catalog.search(**search_params)
+print(f'{type(search_results)=}\n',search_results)
+```
+
+
+
+El objeto `search_results` que se obtuvo al llamar al método `search` es del tipo `ItemSearch`. Para recuperar los resultados, invocamos al método `items` y convertimos el resultado en una `list` de Python que asociaremos al identificador `granules`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+granules = list(search_results.items())
+print(f"Number of granules found with tiles overlapping given AOI: {len(granules)}")
+```
+
+
+
+Se analiza el contenido de la lista `granules`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+granule = granules[0]
+print(f'{type(granule)=}')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+granule
+```
+
+
+
+El objeto `granule` tiene una representación de salida enriquecida en este cuaderno computacional de Jupyter. Podemos ampliar los atributos en la celda de salida haciendo clic en los triángulos.
+
+
+
+El término _grano_ se refiere a una colección de archivos de datos (datos ráster en este caso), todos ellos asociados a datos sin procesar adquiridos por un satélite concreto en una fecha y hora fija sobre un mosaico geográfico concreto. Hay una gran variedad de atributos interesantes asociados con este grano.
+
+- properties['datetime']: una cadena que representa la hora de adquisición de los datos de los archivos de datos ráster de este grano,
+- properties['eo:cloud_cover']: el porcentaje de píxeles oscurecidos por nubes y sombras de nubes en los archivos de datos ráster de este grano, y
+- `assets`: un `dict` de Python cuyos valores resumen las bandas o niveles de los datos ráster asociados con este gránulo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f"{type(granule.properties)=}\n")
+print(f"{granule.properties['datetime']=}\n")
+print(f"{granule.properties['eo:cloud_cover']=}\n")
+print(f"{type(granule.assets)=}\n")
+print(f"{granule.assets.keys()=}\n")
+```
+
+
+
+Cada objeto en `granule.assets` es una instancia de la clase `Asset` que tiene un atributo `href`. Es el atributo `href` el que nos indica dónde localizar un archivo GeoTiff asociado con el activo de este gránulo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+for a in granule.assets:
+ print(f"{a=}\t{type(granule.assets[a])=}")
+ print(f"{granule.assets[a].href=}\n\n")
+```
+
+
+
+Además, el `Item` tiene un atributo `.id` que almacena una cadena de caracteres. Al igual que ocurre con los nombres de archivos asociados a los productos OPERA, esta cadena `.id` contiene el identificador de un mosaico geográfico MGRS. Podemos extraer ese identificador aplicando manipulación de cadenas con Python al atributo `.id` del gránulo. Se realiza y se almacena el resultado en la variable `tile_id`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(granule.id)
+tile_id = granule.id.split('_')[3]
+print(f"{tile_id=}")
+```
+
+***
+
+### Resumiendo los resultados de la búsqueda en un DataFrame
+
+
+
+Los detalles de los resultados de la búsqueda son complicados de analizar de esta manera. Se extraen algunos campos concretos de los gránulos obtenidos en un `DataFrame` de Pandas utilizando una función de Python. Definiremos la función aquí y la reutilizaremos en cuadernos posteriores.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# utility to extract search results into a Pandas DataFrame
+def search_to_dataframe(search):
+ '''Constructs Pandas DataFrame from PySTAC Earthdata search results.
+ DataFrame columns are determined from search item properties and assets.
+ 'asset': string identifying an Asset type associated with a granule
+ 'href': data URL for file associated with the Asset in a given row.'''
+ granules = list(search.items())
+ assert granules, "Error: empty list of search results"
+ props = list({prop for g in granules for prop in g.properties.keys()})
+ tile_ids = map(lambda granule: granule.id.split('_')[3], granules)
+ rows = (([g.properties.get(k, None) for k in props] + [a, g.assets[a].href, t])
+ for g, t in zip(granules,tile_ids) for a in g.assets )
+ df = pd.concat(map(lambda x: pd.DataFrame(x, index=props+['asset','href', 'tile_id']).T, rows),
+ axis=0, ignore_index=True)
+ assert len(df), "Empty DataFrame"
+ return df
+```
+
+
+
+Invocar `search_to_dataframe` en `search_results` codifica la mayor parte de la información importante de la búsqueda como un Pandas `DataFrame`, tal como se muestra a continuación.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = search_to_dataframe(search_results)
+df.head()
+```
+
+
+
+El método `DataFrame.info` nos permite examinar la estructura de este DataFrame.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.info()
+```
+
+
+
+Se limpia el DataFrame que contiene los resultados de búsqueda. Esto podría estar incluido en una función, pero vale la pena saber cómo se hace esto con Pandas de manera interactiva.
+
+En primer lugar, para estos resultados, solo es necesaria una columna `Datetime`, se pueden eliminar las demás.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = df.drop(['start_datetime', 'end_datetime'], axis=1)
+df.info()
+```
+
+
+
+A continuación, se arregla el esquema del `DataFrame` `df` convirtiendo las columnas en tipos de datos sensibles. También será conveniente utilizar la marca de tiempo de la adquisición como índice del DataFrame. Se realiza utilizando el método `DataFrame.set_index`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df['datetime'] = pd.DatetimeIndex(df['datetime'])
+df['eo:cloud_cover'] = df['eo:cloud_cover'].astype(np.float16)
+str_cols = ['asset', 'href', 'tile_id']
+for col in str_cols:
+ df[col] = df[col].astype(pd.StringDtype())
+df = df.set_index('datetime').sort_index()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.info()
+```
+
+
+
+Como resultado se obtiene un DataFrame con un esquema conciso que se puede utilizar para manipulaciones posteriores. Agrupar los resultados de la búsqueda STAC en un `DataFrame` de Pandas de forma razonable es un poco complicado. Varias de las manipulaciones anteriores podrían haberse incluido en la función `search_to_dataframe`. Pero, dado que los resultados de búsqueda de la API de STAC aún están evolucionando, actualmente es mejor ser flexible y utilizar Pandas de forma interactiva para trabajar con los resultados de búsqueda. Se verá esto con más detalle en ejemplos posteriores.
+
+
+
+***
+
+## Explorar y refinar los resultados de búsqueda
+
+
+
+Si se examina la columna numérica `eo:cloud_cover` del DataFrame `df`, se pueden recopilar estadísticas utilizando agregaciones estándar y el método `DataFrame.agg`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df['eo:cloud_cover'].agg(['min','mean','median','max'])
+```
+
+
+
+Observa que hay varias entradas `nan` en esta columna. Las funciones de agregación estadística de Pandas suelen ser "`nan`-aware", esto significa que ignoran implícitamente las entradas `nan` al calcular las estadísticas.
+
+
+
+### Filtrado del DataFrame de búsqueda con Pandas
+
+
+
+Como primera operación de filtrado, se mantienen solo las filas para las que la cobertura de las nubes es inferior al 50%.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df_clear = df.loc[df['eo:cloud_cover']<50]
+df_clear
+```
+
+
+
+Para esta consulta de búsqueda, cada gránulo DSWX comprende datos ráster para diez bandas o niveles. Se puede ver esto aplicando el método Pandas `Series.value_counts` a la columna `asset`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df_clear.asset.value_counts()
+```
+
+
+
+Se filtran las filas que corresponden a la banda `B01_WTR` del producto de datos DSWx. La función de Pandas `DataFrame.str` hace que esta operación sea sencilla. Se nombra al `DataFrame` filtrado como `b01_wtr`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+b01_wtr = df_clear.loc[df_clear.asset.str.contains('B01_WTR')]
+b01_wtr.info()
+b01_wtr.asset.value_counts()
+```
+
+
+
+También se puede observar que hay varios mosaicos geográficos asociados a los gránulos encontrados que intersecan el AOI proporcionado.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+b01_wtr.tile_id.value_counts()
+```
+
+
+
+Recuerda que estos códigos se refieren a mosaicos geográficos MGRS especificados en un sistema de coordenadas concreto. Como se identifican estos códigos en la columna `tile_id`, se puede filtrar las filas que corresponden, por ejemplo, a los archivos recopilados sobre el mosaico T15RUQ del MGRS:
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+b01_wtr_t15ruq = b01_wtr.loc[b01_wtr.tile_id=='T15RUQ']
+b01_wtr_t15ruq
+```
+
+
+
+Se obtiene un `DataFrame` `b01_wtr_t15ruq` mucho más corto que resume las ubicaciones remotas de los archivos (por ejemplo, GeoTiffs) que almacenan datos ráster para la banda de aguas superficiales `B01_WTR` en el mosaico MGRS `T15RUQ` recopilados en varias marcas de tiempo que se encuentran dentro de la ventana de tiempo que especificamos. Se puede utilizar este DataFrame para descargar esos archivos para su análisis o visualización.
+
+
+
+***
+
+## Procesamiento de datos para obtener resultados relevantes
+
+### Apilamiento de los datos
+
+
+
+Se cuenta con un `DataFrame` que identifica archivos remotos específicos de datos ráster. El siguiente paso es combinar estos datos ráster en una estructura de datos adecuada para el análisis. El Xarray `DataArray` es adecuado en este caso. La combinación puede generarse utilizando la función `concat` de Xarray. La función `urls_to_stack` en la siguiente celda es larga pero no es complicada. Toma un `DataFrame` con marcas de tiempo en el índice y una columna etiquetada `href` de las URL, lee los archivos asociados a esas URL uno a uno, y apila las matrices bidimensionales relevantes de datos ráster en una matriz tridimensional.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+def urls_to_stack(granule_dataframe):
+ '''Processes DataFrame of PySTAC search results (with OPERA tile URLs) &
+ returns stacked Xarray DataArray (dimensions time, latitude, & longitude)'''
+
+ stack = []
+ for i, row in granule_dataframe.iterrows():
+ with rasterio.open(row.href) as ds:
+ # extract CRS string
+ crs = str(ds.crs).split(':')[-1]
+ # extract the image spatial extent (xmin, ymin, xmax, ymax)
+ xmin, ymin, xmax, ymax = ds.bounds
+ # the x and y resolution of the image is available in image metadata
+ x_res = np.abs(ds.transform[0])
+ y_res = np.abs(ds.transform[4])
+ # read the data
+ img = ds.read()
+ # Ensure img has three dimensions (bands, y, x)
+ if img.ndim == 2:
+ img = np.expand_dims(img, axis=0)
+ lon = np.arange(xmin, xmax, x_res)
+ lat = np.arange(ymax, ymin, -y_res)
+ bands = np.arange(img.shape[0])
+ da = xr.DataArray(
+ data=img,
+ dims=["band", "lat", "lon"],
+ coords=dict(
+ lon=(["lon"], lon),
+ lat=(["lat"], lat),
+ time=i,
+ band=bands
+ ),
+ attrs=dict(
+ description="OPERA DSWx B01",
+ units=None,
+ ),
+ )
+ da.rio.write_crs(crs, inplace=True)
+ stack.append(da)
+ return xr.concat(stack, dim='time').squeeze()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+stack = urls_to_stack(b01_wtr_t15ruq)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+### Crear una visualización de los datos
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Define a colormap with RGBA tuples
+COLORS = [(150, 150, 150, 0.1)]*256 # Setting all values to gray with low opacity
+COLORS[0] = (0, 255, 0, 0.1) # Not-water class to green
+COLORS[1] = (0, 0, 255, 1) # Open surface water
+COLORS[2] = (0, 0, 255, 1) # Partial surface water
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='lon',
+ y='lat',
+ project=True,
+ rasterize=True,
+ cmap=COLORS,
+ colorbar=False,
+ tiles = gv.tile_sources.OSM,
+ widget_location='bottom',
+ frame_width=500,
+ frame_height=500,
+ xlabel='Longitude (degrees)',
+ ylabel='Latitude (degrees)',
+ title = 'DSWx data for May 2024 Texas floods',
+ fontscale=1.25
+ )
+```
+
+
+
+Visualizar las imágenes completas puede consumir mucha memoria. Se utiliza el método Xarray `DataArray.isel` para extraer un trozo del arreglo `stack` con menos píxeles. Esto permitirá un rápido renderizado y desplazamiento.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+view = stack.isel(lon=slice(3000,None), lat=slice(3000,None))
+view.hvplot.image(**image_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack.hvplot.image(**image_opts) # Construct view from all slices.
+```
+
+
+
+Antes de continuar, recuerda apagar el kernel de este cuaderno computacional para liberar memoria para otros cálculos.
+
+
+
+***
+
+
+
+Este cuaderno computacional proporciona principalmente un ejemplo para ilustrar el uso de la API de PySTAC.
+
+En los siguientes cuadernos computacionales, utilizaremos este flujo de trabajo general:
+
+1. Establecer una consulta de búsqueda mediante la identificación de un _AOI_ particular y un _intervalo de fechas_.
+2. Identificar un _endpoint_, un _proveedor_ y un _catálogo de activos_ adecuados, y ejecutar la búsqueda utilizando `pystac.Client`.
+3. Convertir los resultados de la búsqueda en un DataFrame de Pandas que contenga los principales campos de interés.
+4. Utilizar el DataFrame resultante para filtrar los archivos de datos remotos más relevantes necesarios para el análisis y/o la visualización.
+5. Ejecutar el análisis y/o la visualización utilizando el DataFrame para recuperar los datos requeridos.
+
+
diff --git a/book/es-419/04_Case_Studies/00_Template.md b/book/es-419/04_Case_Studies/00_Template.md
new file mode 100644
index 0000000..55ea976
--- /dev/null
+++ b/book/es-419/04_Case_Studies/00_Template.md
@@ -0,0 +1,232 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Plantilla para el uso de del servicio cloud ofrecido por EarthData
+
+## Esquema de los pasos para el análisis
+
+
+
+- Identificación de los parámetros de búsqueda
+ - Área de interés (AOI, por las siglas en inglés de _area of interest_) y ventana temporal
+ - _Endpoint_, proveedor, identificador del catálogo ("nombre corto")
+- Obtención de los resultados de la búsqueda
+ - Exploracion, análisis para identificar características, bandas de interés
+ - Almacenar los resultados en un DataFrame para facilitar la exploración
+- Explorar y refinar los resultados de la búsqueda
+ - Identificar los gránulos de mayor valor
+ - Filtrar los gránulos atípicos con mínima contribución
+ - Combinar los gránulos filtrados relevantes en un DataFrame
+ - Identificar el tipo de salida a generar
+- Procesar los datos para obtener resultados relevantes
+ - Descargar los gránulos relevantes en Xarray DataArray, apilados adecuadamente
+ - Realizar los cálculos intermedios necesarios
+ - Combinar los datos relevantes en una visualización
+
+
+
+***
+
+### Importación preliminar de librerías
+
+```python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+# data wrangling imports
+import numpy as np
+import pandas as pd
+import xarray as xr
+import rioxarray as rio
+import rasterio
+```
+
+```python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas
+import hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```python jupyter={"source_hidden": false}
+# STAC imports to retrieve cloud data
+from pystac_client import Client
+from osgeo import gdal
+# GDAL setup for accessing cloud data
+gdal.SetConfigOption('GDAL_HTTP_COOKIEFILE','~/.cookies.txt')
+gdal.SetConfigOption('GDAL_HTTP_COOKIEJAR', '~/.cookies.txt')
+gdal.SetConfigOption('GDAL_DISABLE_READDIR_ON_OPEN','EMPTY_DIR')
+gdal.SetConfigOption('CPL_VSIL_CURL_ALLOWED_EXTENSIONS','TIF, TIFF')
+```
+
+### Funciones prácticas
+
+Estas funciones podrían incluirse en archivos de módulos para proyectos de investigación más evolucionados. Para fines didácticos, se incluyen en este cuaderno computacional.
+
+```python jupyter={"source_hidden": false}
+# simple utility to make a rectangle with given center of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding-box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+```python jupyter={"source_hidden": false}
+# simple utility to plot an AOI or bounding-box
+def plot_bbox(bbox):
+ '''Given bounding-box, returns GeoViews plot of Rectangle & Point at center
+ + bbox: bounding-box specified as (lon_min, lat_min, lon_max, lat_max)
+ Assume longitude-latitude coordinates.
+ '''
+ # These plot options are fixed but can be over-ridden
+ point_opts = opts.Points(size=12, alpha=0.25, color='blue')
+ rect_opts = opts.Rectangles(line_width=0, alpha=0.1, color='red')
+ lon_lat = (0.5*sum(bbox[::2]), 0.5*sum(bbox[1::2]))
+ return (gv.Points([lon_lat]) * gv.Rectangles([bbox])).opts(point_opts, rect_opts)
+```
+
+```python jupyter={"source_hidden": false}
+# utility to extract search results into a Pandas DataFrame
+def search_to_dataframe(search):
+ '''Constructs Pandas DataFrame from PySTAC Earthdata search results.
+ DataFrame columns are determined from search item properties and assets.
+ 'asset': string identifying an Asset type associated with a granule
+ 'href': data URL for file associated with the Asset in a given row.'''
+ granules = list(search.items())
+ assert granules, "Error: empty list of search results"
+ props = list({prop for g in granules for prop in g.properties.keys()})
+ tile_ids = map(lambda granule: granule.id.split('_')[3], granules)
+ rows = (([g.properties.get(k, None) for k in props] + [a, g.assets[a].href, t])
+ for g, t in zip(granules,tile_ids) for a in g.assets )
+ df = pd.concat(map(lambda x: pd.DataFrame(x, index=props+['asset','href', 'tile_id']).T, rows),
+ axis=0, ignore_index=True)
+ assert len(df), "Empty DataFrame"
+ return df
+```
+
+```python jupyter={"source_hidden": false}
+# utility to process DataFrame of search results & return DataArray of stacked raster images
+def urls_to_stack(granule_dataframe):
+ '''Processes DataFrame of PySTAC search results (with OPERA tile URLs) &
+ returns stacked Xarray DataArray (dimensions time, latitude, & longitude)'''
+
+ stack = []
+ for i, row in granule_dataframe.iterrows():
+ with rasterio.open(row.href) as ds:
+ # extract CRS string
+ crs = str(ds.crs).split(':')[-1]
+ # extract the image spatial extent (xmin, ymin, xmax, ymax)
+ xmin, ymin, xmax, ymax = ds.bounds
+ # the x and y resolution of the image is available in image metadata
+ x_res = np.abs(ds.transform[0])
+ y_res = np.abs(ds.transform[4])
+ # read the data
+ img = ds.read()
+ # Ensure img has three dimensions (bands, y, x)
+ if img.ndim == 2:
+ img = np.expand_dims(img, axis=0)
+ lon = np.arange(xmin, xmax, x_res)
+ lat = np.arange(ymax, ymin, -y_res)
+ bands = np.arange(img.shape[0])
+ da = xr.DataArray(
+ data=img,
+ dims=["band", "lat", "lon"],
+ coords=dict(
+ lon=(["lon"], lon),
+ lat=(["lat"], lat),
+ time=i,
+ band=bands
+ ),
+ attrs=dict(
+ description="OPERA DSWx B01",
+ units=None,
+ ),
+ )
+ da.rio.write_crs(crs, inplace=True)
+ stack.append(da)
+ return xr.concat(stack, dim='time').squeeze()
+```
+
+***
+
+## Identificación de los parámetros de búsqueda
+
+```python jupyter={"source_hidden": false}
+AOI = ...
+DATE_RANGE = ...
+```
+
+```python jupyter={"source_hidden": false}
+# Optionally plot the AOI
+```
+
+```python jupyter={"source_hidden": false}
+search_params = dict(bbox=AOI, datetime=DATE_RANGE)
+print(search_params)
+```
+
+***
+
+## Obtención de los resultados de la búsqueda
+
+```python jupyter={"source_hidden": false}
+ENDPOINT = ...
+PROVIDER = ...
+COLLECTIONS = ...
+# Update the dictionary opts with list of collections to search
+search_params.update(collections=COLLECTIONS)
+print(search_params)
+```
+
+```python jupyter={"source_hidden": false}
+catalog = Client.open(f'{ENDPOINT}/{PROVIDER}/')
+search_results = catalog.search(**search_params)
+```
+
+```python
+df = search_to_dataframe(search_results)
+df.head()
+```
+
+Limpiar el DataFrame `df` de forma que tenga sentido (por ejemplo, eliminando columnas/filas innecesarias, convirtiendo columnas en tipos de datos fijos, estableciendo un índice, etc.).
+
+```python
+```
+
+***
+
+## Exploración y refinamiento de los resultados de la búsqueda
+
+
+
+Consiste en filtrar filas o columnas adecuadamente para limitar los resultados de la búsqueda a los archivos de datos ráster más relevantes para el análisis y/o la visualización. Esto puede significar enfocarse en determinadas regiones geográficos, bandas específicas del producto de datos, determinadas fechas o períodos, etc.
+
+
+
+```python
+```
+
+***
+
+## Procesamiento de los datos para obtener resultados relevantes
+
+Esto puede incluir apilar matrices bidimensionales en una matriz tridimensional, combinar imágenes ráster de mosaicos adyacentes en uno solo, etc.
+
+```python
+```
+
+***
diff --git a/book/es-419/04_Case_Studies/01_Greece_Wildfires.md b/book/es-419/04_Case_Studies/01_Greece_Wildfires.md
new file mode 100644
index 0000000..ae9b379
--- /dev/null
+++ b/book/es-419/04_Case_Studies/01_Greece_Wildfires.md
@@ -0,0 +1,481 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Incendios forestales en Grecia
+
+
+
+En este ejemplo, recuperaremos los datos asociados a los [incendios forestales en Grecia en el 2023](https://es.wikipedia.org/wiki/Incendios_forestales_en_Grecia_de_2023) para comprender su evolución y extensión. Generaremos una serie temporal asociada a estos datos y dos visualizaciones del evento.
+
+En particular, analizaremos la zona que está alrededor de la ciudad de [Alexandroupolis](https://es.wikipedia.org/wiki/Alejandr%C3%B3polis), que se vio gravemente afectada por incendios forestales, con la consiguiente pérdida de vidas, propiedades y zonas boscosas.
+
+---
+
+
+
+## Esquema de las etapas del análisis
+
+
+
+- Identificación de los parámetros de búsqueda
+ - AOI, ventana temporal
+ - _Endpoint_, proveedor, identificador del catálogo ("nombre corto")
+- Obtención de los resultados de la búsqueda
+ - Inspección, análisis para identificar características, bandas de interés
+ - Almacenamos los resultados en un DataFrame para facilitar la exploración
+- Exploración y refinamiento de los resultados de la búsqueda
+ - Identificar los gránulos de mayor valor
+ - Filtrar los gránulos extraños con una contribución mínima
+ - Integrar los gránulos filtrados en un DataFrame
+ - Identificar el tipo de resultado que se quiere generar
+- Procesamiento de los datos para generar resultados relevantes
+ - Descargar los gránulos relevantes en Xarray DataArray, apilados adecuadamente
+ - Llevar a cabo los cálculos intermedios necesarios
+ - Unir los fragmentos de datos relevantes en la visualización
+
+---
+
+
+
+### Importación preliminar
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+import numpy as np, pandas as pd, xarray as xr
+import rioxarray as rio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# STAC imports to retrieve cloud data
+from pystac_client import Client
+from osgeo import gdal
+# GDAL setup for accessing cloud data
+gdal.SetConfigOption('GDAL_HTTP_COOKIEFILE','~/.cookies.txt')
+gdal.SetConfigOption('GDAL_HTTP_COOKIEJAR', '~/.cookies.txt')
+gdal.SetConfigOption('GDAL_DISABLE_READDIR_ON_OPEN','EMPTY_DIR')
+gdal.SetConfigOption('CPL_VSIL_CURL_ALLOWED_EXTENSIONS','TIF, TIFF')
+```
+
+### Funciones prácticas
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle with given center of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding-box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to plot an AOI or bounding-box
+def plot_bbox(bbox):
+ '''Given bounding-box, returns GeoViews plot of Rectangle & Point at center
+ + bbox: bounding-box specified as (lon_min, lat_min, lon_max, lat_max)
+ Assume longitude-latitude coordinates.
+ '''
+ # These plot options are fixed but can be over-ridden
+ point_opts = opts.Points(size=12, alpha=0.25, color='blue')
+ rect_opts = opts.Rectangles(line_width=2, alpha=0.1, color='red')
+ lon_lat = (0.5*sum(bbox[::2]), 0.5*sum(bbox[1::2]))
+ return (gv.Points([lon_lat]) * gv.Rectangles([bbox])).opts(point_opts, rect_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# utility to extract search results into a Pandas DataFrame
+def search_to_dataframe(search):
+ '''Constructs Pandas DataFrame from PySTAC Earthdata search results.
+ DataFrame columns are determined from search item properties and assets.
+ 'asset': string identifying an Asset type associated with a granule
+ 'href': data URL for file associated with the Asset in a given row.'''
+ granules = list(search.items())
+ assert granules, "Error: empty list of search results"
+ props = list({prop for g in granules for prop in g.properties.keys()})
+ tile_ids = map(lambda granule: granule.id.split('_')[3], granules)
+ rows = (([g.properties.get(k, None) for k in props] + [a, g.assets[a].href, t])
+ for g, t in zip(granules,tile_ids) for a in g.assets )
+ df = pd.concat(map(lambda x: pd.DataFrame(x, index=props+['asset','href', 'tile_id']).T, rows),
+ axis=0, ignore_index=True)
+ assert len(df), "Empty DataFrame"
+ return df
+```
+
+
+
+Estas funciones podrían incluirse en archivos modular para proyectos de investigación más evolucionados. Para fines didácticos, se incluyen en este cuaderno computacional.
+
+---
+
+
+
+## Identificación de los parámetros de búsqueda
+
+```{code-cell} python jupyter={"source_hidden": false}
+dadia_forest = (26.18, 41.08)
+AOI = make_bbox(dadia_forest, 0.1, 0.1)
+DATE_RANGE = '2023-08-01/2023-09-30'.split('/')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Optionally plot the AOI
+basemap = gv.tile_sources.ESRI(width=500, height=500, padding=0.1, alpha=0.25)
+plot_bbox(AOI) * basemap
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+search_params = dict(bbox=AOI, datetime=DATE_RANGE)
+print(search_params)
+```
+
+---
+
+## Obtención de los resultados de búsqueda
+
+```{code-cell} python jupyter={"source_hidden": false}
+ENDPOINT = 'https://cmr.earthdata.nasa.gov/stac'
+PROVIDER = 'LPCLOUD'
+COLLECTIONS = ["OPERA_L3_DIST-ALERT-HLS_V1_1"]
+# Update the dictionary opts with list of collections to search
+search_params.update(collections=COLLECTIONS)
+print(search_params)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+catalog = Client.open(f'{ENDPOINT}/{PROVIDER}/')
+search_results = catalog.search(**search_params)
+```
+
+
+
+Como de costumbre, codificaremos el resultado de la búsqueda en un Pandas `DataFrame`, analizaremos los resultados, y haremos algunas transformaciones para limpiarlos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+df = search_to_dataframe(search_results)
+df.head()
+```
+
+
+
+Limpiaremos el `DataFrame` `df` de las formas típicas:
+
+- convertiendo la columna `datetime` en `DatetimeIndex`,
+- eliminando columnas de tipo `datetime` extrañas,
+- renombrando la columna `eo:cloud_cover` como `cloud_cover`,
+- convirtiendo la columna `cloud_cover` en valores de punto flotante, y
+- convertiendo las columnas restantes en cadenas de caracteres, y
+- estableciendo la columna `datetime` como `Index`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = df.drop(['end_datetime', 'start_datetime'], axis=1)
+df.datetime = pd.DatetimeIndex(df.datetime)
+df = df.rename(columns={'eo:cloud_cover':'cloud_cover'})
+df['cloud_cover'] = df['cloud_cover'].astype(np.float16)
+for col in ['asset', 'href', 'tile_id']:
+ df[col] = df[col].astype(pd.StringDtype())
+df = df.set_index('datetime').sort_index()
+df.info()
+```
+
+---
+
+## Exploración y refinamiento de los resultados de la búsqueda
+
+
+
+Examinemos el `DataFrame` `df` para comprender mejor los resultados de la búsqueda. En primer lugar, veamos cuántos mosaicos geográficos diferentes aparecen en los resultados de la búsqueda.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.tile_id.value_counts()
+```
+
+
+
+Entonces, el AOI se encuentra estrictamente dentro de un único mosaico geográfico MGRS llamado `T35TMF`. Analicemos la columna `asset`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.asset.value_counts().sort_values(ascending=False)
+```
+
+
+
+Algunos de los nombres de estos activos no son tan simples y ordenados como los que encontramos con los productos de datos DIST-ALERT. Sin embargo, podemos identificar fácilmente las subcadenas de caracteres útiles. En este caso, elegimos sólo las filas en las que la columna `asset` incluye `'VEG-DIST-STATUS'` como subcadena de caracteres.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+idx_veg_dist_status = df.asset.str.contains('VEG-DIST-STATUS')
+idx_veg_dist_status
+```
+
+
+
+Podemos utilizar esta `Series` booleana con el método de acceso `.loc` de Pandas para filtrar solo las filas que queremos (por ejemplo, las que se conectan a archivos de datos ráster que almacenan la banda `VEG-DIST-STATUS`). Posteriormente podemos eliminar la columna `asset` (que ya no es necesaria).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_status = df.loc[idx_veg_dist_status]
+veg_dist_status = veg_dist_status.drop('asset', axis=1)
+veg_dist_status
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(len(veg_dist_status))
+```
+
+
+
+Observe que algunas de las filas tienen la misma fecha pero horas diferentes (lo cual corresponde a varias observaciones en el mismo día del calendario UTC). Podemos agregar las URL en listas _remuestreando_ la serie temporal por día. Posteriormente podremos visualizar el resultado.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+by_day = veg_dist_status.resample('1d').href.apply(list)
+display(by_day)
+by_day.map(len).hvplot.scatter(grid=True).opts(title='# of observations')
+```
+
+
+
+Limpiemos la `Series` `by_day` filtrando las filas que tienen listas vacías (es decir, fechas en las que no se adquirieron datos).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+by_day = by_day.loc[by_day.map(bool)]
+by_day.map(len).hvplot.scatter(ylim=(0,2.1), grid=True).opts(title="# of observations")
+```
+
+
+
+Ahora podemos utilizar la serie `by_day` remuestreada para extraer datos ráster para su análisis.
+
+---
+
+
+
+## Procesamiento de los datos
+
+
+
+El incendio forestal cerca de Alexandroupolis comenzó alrededor del 21 de agosto y se propagó rápidamente, afectando en particular al cercano bosque de Dadia. En primer lugar, vamos a ensamblar un "cubo de datos" (por ejemplo, un arreglo apilado de rásters) a partir de los archivos remotos indexados en la serie Pandas `by_day`. Empezaremos seleccionando y cargando uno de los archivos GeoTIFF remotos para extraer los metadatos que se aplican a todos los rásteres asociados con este evento y este mosaico MGRS.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+href = by_day[0][0]
+data = rio.open_rasterio(href).rename(dict(x='longitude', y='latitude'))
+crs = data.rio.crs
+shape = data.shape
+```
+
+
+
+Antes de construir un `DataArray` apilado dentro de un bucle, definiremos un diccionario de Python llamado `template` que se utilizará para instanciar los cortes del arreglo. El diccionario `template` almacenará los metadatos extraídos del archivo GeoTIFF, especialmente las coordenadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+template = dict()
+template['coords'] = data.coords.copy()
+del template['coords']['band']
+template['coords'].update({'time': by_day.index.values})
+template['dims'] = ['time', 'longitude', 'latitude']
+template['attrs'] = dict(description=f"OPERA DSWX: VEG-DIST-STATUS", units=None)
+
+print(template)
+```
+
+
+
+Utilizaremos un bucle para construir un arreglo apilado de rásteres a partir de la serie Pandas `by_day` (cuyas entradas son listas de cadenas de caracteres, es decir, URIs). Si la lista tiene un único elemento, la URL se puede leer directamente utilizando `rasterio.open`; de lo contrario, la función [`rasterio.merge.merge`](https://rasterio.readthedocs.io/en/latest/api/rasterio.merge.html) combina varios archivos de datos ráster adquiridos el mismo día en una única imagen ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import rasterio
+from rasterio.merge import merge
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+rasters = []
+for date, hrefs in by_day.items():
+ n_files = len(hrefs)
+ if n_files > 1:
+ print(f"Merging {n_files} files for {date.strftime('%Y-%m-%d')}...")
+ raster, _ = merge(hrefs)
+ else:
+ print(f"Opening {n_files} file for {date.strftime('%Y-%m-%d')}...")
+ with rasterio.open(hrefs[0]) as ds:
+ raster = ds.read()
+ rasters.append(np.reshape(raster, newshape=shape))
+```
+
+
+
+Los datos acumulados en la lista de `rasters` se almacenan como arreglos de NumPy. Así, en vez de llamar a `xarray.concat`, realizamos una llamada a `numpy.concatenate` dentro de una llamada al constructor `xarray.DataArray`. Vinculamos el objeto creado al identificador `stack`, asegurándonos de incluir también la información CRS.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = xr.DataArray(data=np.concatenate(rasters, axis=0), **template)
+stack.rio.write_crs(crs, inplace=True)
+stack
+```
+
+
+
+La pila `DataArray` `stack` tiene `time`, `longitude` y `latitude` como principales dimensiones de coordenadas. Podemos utilizarla para hacer algunos cálculos y generar visualizaciones.
+
+---
+
+
+
+### Visualización del área dañada
+
+
+
+Para empezar, utilicemos los datos en `stack` para calcular la superficie total dañada. Los datos en `stack` provienen de la banda `VEG-DIST-STATUS` del producto DIST-ALERT. Interpretamos los valores de los píxeles en esta banda de la siguiente manera:
+
+- **0:** Sin alteración
+- **1:** La primera detección de alteraciones con cambio en la cobertura vegetal $<50\%$
+- **2:** Detección provisional de alteraciones con cambio en la cobertura vegetal $<50\%$
+- **3:** Detección confirmada de alteraciones con cambio en la cobertura vegetal $<50\%$
+- **4:** La primera detección de alteraciones con cambio en la cobertura vegetal $\ge50\%$
+- **5:** Detección provisional de alteraciones con cambio en la cobertura vegetal $\ge50\%$
+- **6:** Detección confirmada de alteraciones con cambio en la cobertura vegetal $\ge50\%$
+- **7:** Detección finalizada de alteraciones con cambio en la cobertura vegetal $<50\%$
+- **8:** Detección finalizada de alteraciones con cambio en la cobertura vegetal $\ge50\%$
+
+El valor del píxel particular que queremos marcar es 6, es decir, un píxel en el que se confirmó que por lo menos el 50% de la cubierta vegetal está dañada y en el que la alteración continúa activamente. Podemos utilizar el método `.sum` para sumar todos los píxeles con valor `6` y el método `.to_series` para representar la suma como una serie de Pandas indexada en el tiempo. También definimos `conversion_factor` que toma en cuenta el área de cada píxel en $\mathrm{km}^2$ (recordemos que cada píxel tiene un área de $30\mathrm{m}\times30\mathrm{m}$).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+pixel_val = 6
+conversion_factor = (30/1_000)**2 / pixel_val
+damage_area = stack.where(stack==pixel_val, other=0).sum(axis=(1,2))
+damage_area = damage_area.to_series() * conversion_factor
+damage_area
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot_title = 'Damaged Area (km²)'
+line_plot_opts = dict(title=plot_title, grid=True, color='r')
+damage_area.hvplot.line(**line_plot_opts)
+```
+
+
+
+Observando el gráfico anterior, parece que los incendios forestales comenzaron alrededor del 21 de agosto y se propagaron rápidamente.
+
+---
+
+
+
+### Visualización de fragmentos temporales seleccionados
+
+
+
+El bosque cercano de Dadia se vio especialmente afectado por los incendios. Para comprobarlo, trazaremos los datos ráster para ver la distribución espacial de los píxeles dañados en tres fechas concretas: 2 de agosto, 26 de agosto y 18 de septiembre. Una vez más, resaltaremos solamente los píxeles que tengan valor 6 en los datos ráster. Podemos extraer fácilmente esas fechas específicas de la serie temporal `by_day` utilizando una lista de fechas (por ejemplo, `dates_of_interest` en la siguiente celda).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+dates_of_interest = ['2023-08-01', '2023-08-26', '2023-09-18']
+print(dates_of_interest)
+snapshots = stack.sel(time=dates_of_interest)
+snapshots
+```
+
+
+
+Vamos a hacer una secuencia estática de los gráficos. Empezaremos definiendo algunas opciones estándar almacenadas en diccionarios.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ crs=crs,
+ shared_axes=False,
+ colorbar=False,
+ aspect='equal',
+ )
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+```
+
+
+
+Construiremos un mapa de colores utilizando un diccionario de valores RGBA (por ejemplo, tuplas con tres entradas enteras entre 0 y 255, y una cuarta entrada de punto flotante entre 0.0 y 1.0 para la transparencia).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+COLORS = { k:(0,0,0,0.0) for k in range(256) }
+COLORS.update({6: (255,0,0,1.0)})
+image_opts.update(cmap=list(COLORS.values()))
+```
+
+
+
+Como siempre, empezaremos por cortar imágenes más pequeñas para asegurarnos de que
+`hvplot.image` funciona correctamente. Podemos reducir el valor del parámetro `steps` a `1` o `None` para obtener las imágenes renderizadas en resolución completa.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 100
+subset = slice(0,None, steps)
+view = snapshots.isel(longitude=subset, latitude=subset)
+(view.hvplot.image(**image_opts).opts(**layout_opts) * basemap).layout()
+```
+
+
+
+Si eliminamos la llamada a `.layout`, podemos producir un desplazamiento interactivo que muestre el progreso del incendio forestal utilizando todos los rásteres en `stack`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 100
+subset = slice(0,None, steps)
+view = stack.isel(longitude=subset, latitude=subset,)
+(view.hvplot.image(**image_opts).opts(**layout_opts) * basemap)
+```
+
+---
diff --git a/book/es-419/04_Case_Studies/02_Lake_Mead_Mosaicking.md b/book/es-419/04_Case_Studies/02_Lake_Mead_Mosaicking.md
new file mode 100644
index 0000000..404c2bf
--- /dev/null
+++ b/book/es-419/04_Case_Studies/02_Lake_Mead_Mosaicking.md
@@ -0,0 +1,501 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.4
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Generación de un mosaico del lago Mead
+
+
+
+El [Lake Mead](https://es.wikipedia.org/wiki/Lago_Mead) es un embalse de agua que se encuentra en el suroeste de los Estados Unidos y es importante para el riego en esa zona. El lago ha tenido una importante sequía durante la última década y, en particular, entre 2020 y 2023. En este cuaderno computacional, buscaremos datos GeoTIFF relacionados con este lago y sintetizaremos varios archivos ráster para producir una visualización.
+
+***
+
+
+
+## Esquema de los pasos para el análisis
+
+
+
+- Identificar los parámetros de búsqueda
+ - AOI y ventana temporal
+ - _Endpoint_, proveedor, identificador del catálogo ("nombre corto")
+- Obtención de los resultados de búsqueda
+ - Exploracion, análisis para identificar características, bandas de interés
+ - Almacenar los resultados en un DataFrame para facilitar la exploración
+- Exploración y refinamiento de los resultados de la búsqueda
+ - Identificar los gránulos de mayor valor
+ - Filtrar los gránulos atípicos con contribución mínima
+ - Combinar los gránulos filtrados en un DataFrame
+ - Identificar el tipo de salida a generar
+- Procesamiento de los datos para obtener resultados relevantes
+ - Descargar los gránulos relevantes en Xarray DataArray, apilados adecuadamente
+ - Realizar los cálculos intermedios necesarios
+ - Unir los datos relevantes en una visualización
+
+
+
+***
+
+### Importación preliminar
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+import numpy as np, pandas as pd, xarray as xr
+import rioxarray as rio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+from bokeh.models import FixedTicker
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# STAC imports to retrieve cloud data
+from pystac_client import Client
+from osgeo import gdal
+# GDAL setup for accessing cloud data
+gdal.SetConfigOption('GDAL_HTTP_COOKIEFILE','~/.cookies.txt')
+gdal.SetConfigOption('GDAL_HTTP_COOKIEJAR', '~/.cookies.txt')
+gdal.SetConfigOption('GDAL_DISABLE_READDIR_ON_OPEN','EMPTY_DIR')
+gdal.SetConfigOption('CPL_VSIL_CURL_ALLOWED_EXTENSIONS','TIF, TIFF')
+```
+
+### Funciones prácticas
+
+
+
+Estas funciones podrían incluirse en archivos modulares para proyectos de investigación más evolucionados. Para fines didácticos, se incluyen en este cuaderno computacional.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle with given center of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding-box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to plot an AOI or bounding-box
+def plot_bbox(bbox):
+ '''Given bounding-box, returns GeoViews plot of Rectangle & Point at center
+ + bbox: bounding-box specified as (lon_min, lat_min, lon_max, lat_max)
+ Assume longitude-latitude coordinates.
+ '''
+ # These plot options are fixed but can be over-ridden
+ point_opts = opts.Points(size=12, alpha=0.25, color='blue')
+ rect_opts = opts.Rectangles(line_width=0, alpha=0.1, color='red')
+ lon_lat = (0.5*sum(bbox[::2]), 0.5*sum(bbox[1::2]))
+ return (gv.Points([lon_lat]) * gv.Rectangles([bbox])).opts(point_opts, rect_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# utility to extract search results into a Pandas DataFrame
+def search_to_dataframe(search):
+ '''Constructs Pandas DataFrame from PySTAC Earthdata search results.
+ DataFrame columns are determined from search item properties and assets.
+ 'asset': string identifying an Asset type associated with a granule
+ 'href': data URL for file associated with the Asset in a given row.'''
+ granules = list(search.items())
+ assert granules, "Error: empty list of search results"
+ props = list({prop for g in granules for prop in g.properties.keys()})
+ tile_ids = map(lambda granule: granule.id.split('_')[3], granules)
+ rows = (([g.properties.get(k, None) for k in props] + [a, g.assets[a].href, t])
+ for g, t in zip(granules,tile_ids) for a in g.assets )
+ df = pd.concat(map(lambda x: pd.DataFrame(x, index=props+['asset','href', 'tile_id']).T, rows),
+ axis=0, ignore_index=True)
+ assert len(df), "Empty DataFrame"
+ return df
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# utility to remap pixel values to a sequence of contiguous integers
+def relabel_pixels(data, values, null_val=255, transparent_val=0, replace_null=True, start=0):
+ """
+ This function accepts a DataArray with a finite number of categorical values as entries.
+ It reassigns the pixel labels to a sequence of consecutive integers starting from start.
+ data: Xarray DataArray with finitely many categories in its array of values.
+ null_val: (default 255) Pixel value used to flag missing data and/or exceptions.
+ transparent_val: (default 0) Pixel value that will be fully transparent when rendered.
+ replace_null: (default True) Maps null_value->transparent_value everywhere in data.
+ start: (default 0) starting range of consecutive integer values for new labels.
+ The values returned are:
+ new_data: Xarray DataArray containing pixels with new values
+ relabel: dictionary associating old pixel values with new pixel values
+ """
+ new_data = data.copy(deep=True)
+ if values:
+ values = np.sort(np.array(values, dtype=np.uint8))
+ else:
+ values = np.sort(np.unique(data.values.flatten()))
+ if replace_null:
+ new_data = new_data.where(new_data!=null_val, other=transparent_val)
+ values = values[np.where(values!=null_val)]
+ n_values = len(values)
+ new_values = np.arange(start=start, stop=start+n_values, dtype=values.dtype)
+ assert transparent_val in new_values, f"{transparent_val=} not in {new_values}"
+ relabel = dict(zip(values, new_values))
+ for old, new in relabel.items():
+ if new==old: continue
+ new_data = new_data.where(new_data!=old, other=new)
+ return new_data, relabel
+```
+
+***
+
+## Identificación de los parámetros de búsqueda
+
+
+
+Identificaremos un punto geográfico cerca de la orilla norte del [lago Mead](https://es.wikipedia.org/wiki/Lago_Mead), haremos un cuadro delimitador y elegiremos un intervalo de fechas que cubra marzo y parte de abril del 2023.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+lake_mead = (-114.754, 36.131)
+AOI = make_bbox(lake_mead, 0.1, 0.1)
+DATE_RANGE = "2023-03-01/2023-04-15"
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Optionally plot the AOI
+basemap = gv.tile_sources.OSM(width=500, height=500, padding=0.1)
+plot_bbox(AOI) * basemap
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+search_params = dict(bbox=AOI, datetime=DATE_RANGE)
+print(search_params)
+```
+
+***
+
+## Obtención de los resultados de búsqueda
+
+
+
+Como siempre, especificaremos el _endpoint_ de búsqueda, el proveedor y el catálogo. Para la familia de productos de datos DSWx son los siguientes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+ENDPOINT = 'https://cmr.earthdata.nasa.gov/stac'
+PROVIDER = 'POCLOUD'
+COLLECTIONS = ["OPERA_L3_DSWX-HLS_V1_1.0"]
+# Update the dictionary opts with list of collections to search
+search_params.update(collections=COLLECTIONS)
+print(search_params)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+catalog = Client.open(f'{ENDPOINT}/{PROVIDER}/')
+search_results = catalog.search(**search_params)
+```
+
+
+
+Convertimos los resultados de la búsqueda en un `DataFrame` para facilitar su lectura.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = search_to_dataframe(search_results)
+display(df.head())
+df.info()
+```
+
+
+
+Limpiaremos el DataFrame `df` de maneras estándar:
+
+- eliminando las columnas `start_datetime` y `end_datetime`,
+- renombrando la columna `eo:cloud_cover`,
+- convirtiendo las columnas a tipos de datos adecuados, y
+- asignando la columna `datetime` como el índice.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.datetime = pd.DatetimeIndex(df.datetime)
+df = df.drop(['start_datetime', 'end_datetime'], axis=1)
+df = df.rename({'eo:cloud_cover':'cloud_cover'}, axis=1)
+df['cloud_cover'] = df['cloud_cover'].astype(np.float16)
+for col in ['asset', 'href', 'tile_id']:
+ df[col] = df[col].astype(pd.StringDtype())
+df = df.set_index('datetime').sort_index()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+display(df.head())
+df.info()
+```
+
+***
+
+## Exploración y refinamiento de los resultados de la búsqueda
+
+
+
+Podemos observar la columna `assets` para ver cuáles bandas diferentes están disponibles en los resultados devueltos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.asset.value_counts()
+```
+
+
+
+La banda `0_B01_WTR` es con la que queremos trabajar posteriormente.
+
+También podemos ver cuánta nubosidad hay en los resultados de nuestra búsqueda.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.cloud_cover.agg(['min','mean','median','max'])
+```
+
+
+
+Podemos extraer las filas seleccionadas del `DataFrame` usando `Series` booleanas. Específicamente, seleccionaremos las filas que tengan menos de 10% de nubosidad y en las que el `asset` sea la banda `0_B01_WTR`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+c1 = (df.cloud_cover <= 10)
+c2 = (df.asset.str.contains('B01_WTR'))
+b01_wtr = df.loc[c1 & c2].drop(['asset', 'cloud_cover'], axis=1)
+b01_wtr
+```
+
+
+
+Por último, podemos ver cuántos mosaicos MGRS diferentes intersecan nuestro AOI.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+b01_wtr.tile_id.value_counts()
+```
+
+
+
+Hay cuatro mosaicos geográficos distintos que intersecan este AOI en particular.
+
+***
+
+
+
+## Procesamiento de los datos para obtener resultados relevantes
+
+
+
+Esta vez, utilizaremos una técnica llamada _creación de mosaicos_ para combinar datos ráster de mosaicos adyacentes en un conjunto único de datos ráster. Esto requiere la función `rasterio.merge.merge` como antes. También necesitaremos la función `rasterio.transform.array_bounds` para alinear correctamente las coordenadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import rasterio
+from rasterio.merge import merge
+from rasterio.transform import array_bounds
+```
+
+
+
+Ya hemos utilizado la función `merge` para combinar distintos conjuntos de datos ráster asociados a un único mosaico MGRS. En esta ocasión, los datos ráster combinados provendrán de mosaicos MGRS adyacentes. Al llamar a la función `merge` en la siguiente celda de código, la columna `b01_wtr.href` se tratará como una lista de URL ([Localizadores Uniformes de Recursos](https://es.wikipedia.org/wiki/Localizador_de_recursos_uniforme) (URL, por su siglas en inglés de _Uniform Resource Locator_)). Para cada URL de la lista, se descargará y se procesará un archivo GeoTIFF. El resultado neto es un mosaico de imágenes, es decir, un ráster fusionado que contiene una combinación de todos los rásteres. Los detalles del algoritmo de fusión se describen en la [documentación del módulo `rasterio.merge`](https://rasterio.readthedocs.io/en/latest/api/rasterio.merge.html).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+mosaicked_img, mosaic_transform = merge(b01_wtr.href)
+```
+
+
+
+La salida consiste de nuevo en un arreglo de NumPy y una transformación de coordenadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f"{type(mosaicked_img)=}\n")
+print(f"{mosaicked_img.shape=}\n")
+print(f"{type(mosaic_transform)=}\n")
+print(f"{mosaic_transform=}\n")
+```
+
+
+
+Las entradas de `mosaic_transform` describen una [_transformación afín_](https://es.wikipedia.org/wiki/Transformaci%C3%B3n_af%C3%ADn) de coordenadas de píxel a coordenadas UTM continuas. En particular:
+
+- la entrada `mosaic_transform[0]` es el ancho horizontal de cada píxel en metros, y
+- la entrada `mosaic_transform[4]` es la altura vertical de cada píxel en metros.
+
+Observa también que, en este caso, `mosaic_transform[4]` es un valor negativo (es decir, `mosaic_transform[4]==-30.0`). Esto nos dice que la orientación del eje vertical de coordenadas continuas se opone a la orientación del eje vertical de coordenadas de píxeles, es decir, la coordenada continua vertical disminuye en dirección descendente, a diferencia de la coordenada vertical de píxeles.
+
+Cuando pasamos el objeto `mosaic_transform` a la función `rasterio.transform.array_bounds`, el valor que se devuelve es un cuadro delimitador, es decir, una tupla de la forma `(x_min, y_min, x_max, y_max)` que describe las esquinas inferior izquierda y superior derecha de la imagen en mosaico resultante en coordenadas continuas UTM.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+bounds = array_bounds(*mosaicked_img.shape[1:], mosaic_transform)
+
+bounds
+```
+
+
+
+La combinación de toda la información anterior nos permite reconstruir las coordenadas UTM continuas asociadas a cada píxel. Computaremos arreglos para estas coordenadas continuas y las etiquetaremos como `longitude` y `latitude`. Estas coordenadas serían más precisas si se llamaran `easting` y `northing`, pero utilizaremos las etiquetas `longitude` y `latitude` respectivamente cuando adjuntemos los arreglos de coordenadas a un Xarray `DataArray`. Elegimos estas etiquetas porque, cuando los datos ráster se visualizan con `hvplot.image`, la salida utilizará coordenadas longitud-latitud.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+longitude = np.arange(bounds[0], bounds[2], mosaic_transform[0])
+latitude = np.arange(bounds[3], bounds[1], mosaic_transform[4])
+```
+
+
+
+Almacenamos la imagen en mosaico y los metadatos relevantes en un Xarray `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = xr.DataArray(
+ data=mosaicked_img,
+ dims=["band", "latitude", "longitude"],
+ coords=dict(
+ longitude=(["longitude"], longitude),
+ latitude=(["latitude"], latitude),
+ ),
+ attrs=dict(
+ description="OPERA DSWx B01",
+ units=None,
+ ),
+ )
+raster
+```
+
+
+
+Necesitamos adjuntar un objeto CRS al objeto `raster`. Para ello, utilizaremos `rasterio.open` para cargar los metadatos relevantes de uno de los gránulos asociados a `b01_wtr` (deberían ser los mismos para todos estos archivos).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(b01_wtr.href[0]) as ds:
+ crs = ds.crs
+
+raster.rio.write_crs(crs, inplace=True)
+print(raster.rio.crs)
+```
+
+
+
+En el código de investigación, podríamos agrupar los comandos anteriores en una función y guardarla en un módulo. No lo haremos aquí porque, para los propósitos de este tutorial, es preferible asegurarse de que podemos analizar la salida de varias líneas de código interactivamente.
+
+Con todos los pasos anteriores completados, estamos listos para producir una visualización del mosaico. Reetiquetaremos los valores de los píxeles para que la barra de colores del resultado final sea más prolija.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster, relabel = relabel_pixels(raster, values=[0,1,2,253,254,255])
+```
+
+
+
+Vamos a definir las opciones de imagen, las opciones de diseño, y un mapa de color en los diccionarios como lo hicimos anteriormente para generar una única visualización.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ crs=raster.rio.crs
+ )
+layout_opts = dict(
+ xlabel='Longitude',
+ ylabel='Latitude',
+ )
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Define a colormap using RGBA values; these need to be written manually here...
+COLORS = {
+0: (255, 255, 255, 0.0), # No Water
+1: (0, 0, 255, 1.0), # Open Water
+2: (180, 213, 244, 1.0), # Partial Surface Water
+3: ( 0, 255, 255, 1.0), # Snow/Ice
+4: (175, 175, 175, 1.0), # Cloud/Cloud Shadow
+5: ( 0, 0, 127, 0.5), # Ocean Masked
+}
+
+c_labels = ["Not water", "Open water", "Partial Surface Water", "Snow/Ice",
+ "Cloud/Cloud Shadow", "Ocean Masked"]
+c_ticks = list(COLORS.keys())
+limits = (c_ticks[0]-0.5, c_ticks[-1]+0.5)
+
+c_bar_opts = dict( ticker=FixedTicker(ticks=c_ticks),
+ major_label_overrides=dict(zip(c_ticks, c_labels)),
+ major_tick_line_width=0, )
+
+image_opts.update({ 'cmap': list(COLORS.values()),
+ 'clim': limits,
+ })
+
+layout_opts.update(dict(colorbar_opts=c_bar_opts))
+```
+
+
+
+Definiremos el mapa base como un objeto separado para superponerlo usando el operador `*`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.ESRI(frame_width=500, frame_height=500, padding=0.05, alpha=0.25)
+```
+
+
+
+Por último, podemos utilizar la función `slice` de Python para extraer rápidamente las imágenes reducidas antes de tratar de ver la imagen completa. Recuerda que reducir el valor de `steps` a `1` (o `None`) visualiza el ráster a resolución completa.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+steps = 1
+view = raster.isel(longitude=slice(0,None,steps), latitude=slice(0,None,steps)).squeeze()
+
+view.hvplot.image(**image_opts).opts(**layout_opts) * basemap
+```
+
+
+
+Este ráster es mucho más grande de los que analizamos anteriormente (requiere aproximadamente 4 veces más espacio de almacenamiento). Este proceso podría ser iterado para hacer un deslizador que muestre los resultados fusionados de mosaicos vecinos en diferentes momentos. Esto, por supuesto, requiere que haya suficiente memoria disponible.
+
+***
+
+
+
+
+El proyecto de Productos de Observación para Usuarios Finales a partir del Análisis por Teledetección (OPERA, por las siglas en inglés de _Observational Products for End-Users from Remote Sensing Analysis_).
+
+> Iniciado en abril del 2021, el proyecto OPERA del _Jet Propulsion Laboratory_ (JPL) (en español, Laboratorio de Propulsión a Chorro) recopila datos de instrumentos satelitales ópticos y de radar para generar seis conjuntos de productos:
+>
+> - un conjunto de productos sobre la Extensión del Agua de la Superficie terrestre a escala casi mundial
+> - un conjunto de productos de Alteraciones de la Superficie terrestre a escala casi mundial
+> - un producto con Corrección Radiométrica del Terreno a escala casi mundial
+> - un conjunto de productos _Coregistered Single Look xomplex_ para Norteamérica
+> - un conjunto de productos de Desplazamiento para Norteamérica
+> - un conjunto de productos de Movimiento Vertical del Terreno en Norteamérica
+
+Es decir, OPERA es una iniciativa de la _National Aeronautics and Space Administration_ (NASA)(en español, Administración Nacional de Aeronáutica y del Espacio) que toma, por ejemplo, datos de teledetección óptica o radar recopilados desde satélites, y genera una variedad de conjuntos de datos preprocesados para uso público. Los productos de OPERA no son imágenes de satélite sin procesar, sino el resultado de una clasificación algorítmica para determinar, por ejemplo, qué regiones terrestres contienen agua o dónde se ha desplazado la vegetación. Las imágenes de satélite sin procesar se recopilan a partir de mediciones realizadas por los instrumentos a bordo de las misiones de los satélites Sentinel-1 A/B, Sentinel-2 A/B y Landsat-8/9 (de ahí el término _HLS_ por las siglas en inglés de "_Harmonized Landsat-Sentinel_" (en español, armonización Landsat-Sentinel) en numerosas descripciones de productos).
+
+
+
+---
+
+## El producto OPERA _Dynamic Surface Water Extent_ (DSWx) (en español, Extensión de aguas superficiales dinámicas)
+
+
+
+Ya vimos la familia DIST (es decir, alteración de la superficie terrestre) con productos de datos OPERA. En este cuaderno computacional, analizaremos otro producto de datos OPERA: el producto _Dynamic Surface Water Extent (DSWx)_ (en español, Extensión de Aguas Superficiales Dinámicas). Este producto de datos resume la extensión de las aguas continentales (es decir, el agua en las masas de tierra en contraposición a la parte de un océano) que se puede utilizar para hacer un seguimiento de las inundaciones y las sequías. El producto DSWx se describe detalladamente en la [especificación del producto OPERA DSWx HLS](https://d2pn8kiwq2w21t.cloudfront.net/documents/ProductSpec_DSWX_URS309746.pdf).
+
+Los productos de datos DSWx se generan a partir de las mediciones de reflectancia superficial (RS) del HLS, específicamente, estas son efectuadas por el _Operational Land Imager_ (OLI) (en español,
+Generador de imágenes terrestres operacional) a bordo del satélite Landsat 8, el OLI-2 a bordo del satélite Landsat 9, y el MultiSpectral Instrument (MSI) (en español, Instrumento multiespectral) a bordo de los satélites Sentinel-2A/B. Al igual que los productos DIST, los productos DSWx consisten en datos ráster almacenados en formato GeoTIFF que utilizan el [_Military Grid Reference System_ (MGRS)](https://en.wikipedia.org/wiki/Military_Grid_Reference_System) (en español, Sistema de Referencia de Cuadrículas Militares) (los detalles se describen en la especificación del [producto DSWx](https://d2pn8kiwq2w21t.cloudfront.net/documents/ProductSpec_DSWX_URS309746.pdf). De nuevo, los productos OPERA DSWx se distribuyen como [GeoTIFF optimizados para la nube](https://www.cogeo.org/) que almacenan diferentes bandas/capas en archivos TIFF distintos.
+
+
+
+---
+
+
+
+## Banda 1: Clasificación del agua (WTR, por las abreviación en inglés de la palabra _Water_)
+
+
+
+Hay diez bandas o capas asociadas al producto de datos DSWx. En este tutorial, nos enfocaremos estrictamente en la primera banda—la capa de _clasificación del agua (WTR)_—pero los detalles de todas las bandas se dan en la [especificación del producto DSWx](https://d2pn8kiwq2w21t.cloudfront.net/documents/ProductSpec_DSWX_URS309746.pdf). Por ejemplo, la banda 3 es la _Capa de Confianza (CONF)_ que proporciona, para cada píxel, valores cuantitativos que describen el grado de confianza en las categorías dadas en la banda 1 (la Capa de clasificación del agua). La banda 4 es una capa de _Diagnóstico (DIAG)_ que codifica, para cada píxel, cuál de las cinco pruebas resultó positiva para obtener el valor del píxel correspondiente en la capa CONF.
+
+La capa de clasificación del agua consiste en datos ráster enteros de 8 bits sin signo (UInt8) que representan si un píxel contiene aguas continentales (por ejemplo, parte de un embalse, un lago, un río, etc., pero no agua asociada con el mar abierto). Los valores de esta capa ráster se calculan a partir de las imágenes crudas adquiridas por el satélite, asignando a los píxeles uno de los 7 valores enteros positivos que analizaremos a continuación.
+
+---
+
+### Análisis de una capa WTR de ejemplo
+
+Comencemos importando las librerías necesarias y cargando un archivo adecuado en un Xarray `DataArray`. El archivo en cuestión contiene datos ráster relativos al [embalse del lago Powelr](https://en.wikipedia.org/wiki/Lake_Powell), en el río Colorado, en los Estados Unidos.
+
+```{code-cell} python
+import warnings
+warnings.filterwarnings('ignore')
+from pathlib import Path
+import numpy as np, pandas as pd, xarray as xr
+import rioxarray as rio
+```
+
+```{code-cell} python
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+gv.extension('bokeh')
+from bokeh.models import FixedTicker
+```
+
+```{code-cell} python
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DSWx-HLS_T12SVG_20230411T180222Z_20230414T030945Z_L8_30_v1.0_B01_WTR.tif')
+b01_wtr = rio.open_rasterio(LOCAL_PATH).rename({'x':'longitude', 'y':'latitude'}).squeeze()
+```
+
+Recuerda que usar el `repr` para `b01_wtr` en este cuaderno computacional Jupyter es bastante conveniente.
+
+- Al desplegar la pestaña `Attributes`, podemos ver todos los metadatos asociados a los datos adquiridos.
+- Al desplegar la pestaña `Coordinates`, podemos analizar todos los arreglos asociados a esos valores de coordenadas.
+
+```{code-cell} python
+# Examine data
+b01_wtr
+```
+
+Vamos a examinar la distribución de los valores del píxel en `b01_wtr` utilizando el método Pandas `Series.value_counts`.
+
+```{code-cell} python
+counts = pd.Series(b01_wtr.values.flatten()).value_counts().sort_index()
+display(counts)
+```
+
+Estos valores de píxel son _datos categóricos_. En específico, los valores de píxel válidos y sus significados—según la [especificación del producto DSWx](https://d2pn8kiwq2w21t.cloudfront.net/documents/ProductSpec_DSWX_URS309746.pdf) —son los siguientes:
+
+- **0**: Sin agua—cualquier área con datos de reflectancia válidos que no sea de una de las otras categorías permitidas (agua abierta, agua superficial parcial, nieve/hielo, nube/sombra de nube, o océano enmascarado).
+- **1**: Agua abierta—cualquier píxel que sea completamente agua sin obstrucciones para el sensor, incluyendo obstrucciones por vegetación, terreno y edificios.
+- **2**: Agua parcialmente superficial—un área que es por lo menos 50% y menos de 100% agua abierta (por ejemplo, sumideros inundados, vegetación flotante, y píxeles bisecados por líneas costeras).
+- **252**: Nieve/Hielo.
+- **253**: Nube o sombra de nube—un área oscurecida por, o adyacente a, una nube o sombra de nube.
+- **254**: Océano enmascarado—un área identificada como océano utilizando una base de datos de la línea costera con un margen añadido.
+- **255**: Valor de relleno (datos faltantes).
+
+---
+
+### Realización de una primera visualización
+
+Hagamos un primer gráfico aproximado de los datos ráster utilizando `hvplot.image`. Como de costumbre, instanciamos un objeto `view` que corta un subconjunto más pequeño de píxeles para hacer que la imagen se renderice rápidamente.
+
+```{code-cell} python
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ project=True
+ )
+layout_opts = dict(
+ xlabel='Longitude',
+ ylabel='Latitude',
+ aspect='equal',
+ )
+
+steps = 100
+subset = slice(0, None, steps)
+view = b01_wtr.isel(longitude=subset, latitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts)
+```
+
+El mapa de colores predeterminado no revela muy bien las características ráster. Además, observa que el eje de la barra de colores cubre el rango numérico de 0 a 255 (aproximadamente), aunque la mayoría de esos valores de los píxeles (es decir, de `3` a `251`) no aparecen realmente en los datos. Anotar una imagen ráster de datos categóricos con una leyenda puede tener más sentido que utilizar una barra de colores. Sin embargo, actualmente, `hvplot.image` no soporta el uso de una leyenda. Por lo tanto, para este tutorial, nos limitaremos a utilizar una barra de colores. Antes de asignar un mapa de colores y etiquetas apropiadas para la barra de colores, tiene sentido limpiar los valores de los píxeles.
+
+---
+
+### Reasignación de los valores de los píxeles
+
+Queremos reasignar los valores ráster de los píxeles a un rango más estrecho (por ejemplo, de `0` a `5` en vez de `0` a `255`) para crear una barra de color sensible. Para ello, empezaremos copiando los valores del `DataArray` `b01_wtr` en otro llamado `new_data`, y creando un arreglo llamado `values` para contener el rango completo de valores de píxel permitidos.
+
+```{code-cell} python
+new_data = b01_wtr.copy(deep=True)
+values = np.array([0, 1, 2, 252, 253, 254, 255], dtype=np.uint8)
+print(values)
+```
+
+Primero debemos decidir cómo tratar los datos que faltan, es decir, los píxeles con el valor `255` en este ráster. Vamos a elegir tratar los píxeles de datos que faltan igual que los píxeles `"Sin agua"`. Podemos utilizar el método `DataArray.where` para reasignar los píxeles con valor `null_val` (por ejemplo, 255 en la celda de código siguiente) al valor de reemplazo `transparent_val` (por ejemplo, `0` en este caso). Anticipando que incluiremos este código en una función más adelante, incluimos el cálculo en un bloque `if` condicionado a un valor booleano `replace_null`.
+
+```{code-cell} python
+null_val = 255
+transparent_val = 0
+replace_null = True
+if replace_null:
+ new_data = new_data.where(new_data!=null_val, other=transparent_val)
+ values = values[np.where(values!=null_val)]
+
+print(np.unique(new_data.values))
+```
+
+Observa que `values` ya no incluye `null_val`. A continuación, instanciamos un arreglo `new_values` para almacenar los valores de píxel de reemplazo.
+
+```{code-cell} python
+n_values = len(values)
+start_val = 0
+new_values = np.arange(start=start_val, stop=start_val+n_values, dtype=values.dtype)
+assert transparent_val in new_values, f"{transparent_val=} not in {new_values}"
+print(values)
+print(new_values)
+```
+
+Ahora combinamos `values` y `new_values` en un diccionario `relabel`, y usamos el diccionario para modificar las entradas de `new_data`.
+
+```{code-cell} python
+relabel = dict(zip(values, new_values))
+for old, new in relabel.items():
+ if new==old: continue
+ new_data = new_data.where(new_data!=old, other=new)
+```
+
+Podemos encapsular la lógica de las celdas anteriores en una función de utilidad `relabel_pixels` que condensa un amplio rango de valores categóricos de píxeles en uno más ajustado que se mostrará mejor con un mapa de colores.
+
+```{code-cell} python
+# utility to remap pixel values to a sequence of contiguous integers
+def relabel_pixels(data, values, null_val=255, transparent_val=0, replace_null=True, start=0):
+ """
+ This function accepts a DataArray with a finite number of categorical values as entries.
+ It reassigns the pixel labels to a sequence of consecutive integers starting from start.
+ data: Xarray DataArray with finitely many categories in its array of values.
+ null_val: (default 255) Pixel value used to flag missing data and/or exceptions.
+ transparent_val: (default 0) Pixel value that will be fully transparent when rendered.
+ replace_null: (default True) Maps null_value->transparent_value everywhere in data.
+ start: (default 0) starting range of consecutive integer values for new labels.
+ The values returned are:
+ new_data: Xarray DataArray containing pixels with new values
+ relabel: dictionary associating old pixel values with new pixel values
+ """
+ new_data = data.copy(deep=True)
+ if values:
+ values = np.sort(np.array(values, dtype=np.uint8))
+ else:
+ values = np.sort(np.unique(data.values.flatten()))
+ if replace_null:
+ new_data = new_data.where(new_data!=null_val, other=transparent_val)
+ values = values[np.where(values!=null_val)]
+ n_values = len(values)
+ new_values = np.arange(start=start, stop=start+n_values, dtype=values.dtype)
+ assert transparent_val in new_values, f"{transparent_val=} not in {new_values}"
+ relabel = dict(zip(values, new_values))
+ for old, new in relabel.items():
+ if new==old: continue
+ new_data = new_data.where(new_data!=old, other=new)
+ return new_data, relabel
+```
+
+Apliquemos la función que acabamos de definir a `b01_wtr` y comprobemos que los valores de los píxeles cambiaron como queríamos.
+
+```{code-cell} python
+values = [0, 1, 2, 252, 253, 254, 255]
+print(f"Before applying relabel_pixels: {np.unique(b01_wtr.values)}")
+print(f"Original pixel values expected: {values}")
+b01_wtr, relabel = relabel_pixels(b01_wtr, values=values)
+print(f"After applying relabel_pixels: {np.unique(b01_wtr.values)}")
+```
+
+Observe que el valor de píxel `5` no aparece en el arreglo reetiquetada porque el valor de píxel `254` (para los píxeles de "Océano enmascarado") no aparece en el archivo GeoTIFF original. Esto está bien. El código escrito a continuación seguirá produciendo toda la variedad de posibles valores para los píxeles (y colores) en su barra de colores.
+
+---
+
+### Definición de un mapa de color y trazado con una barra de color
+
+Ahora estamos listos para definir un mapa de colores. Definimos el diccionario `COLORS` de forma que las etiquetas de píxel de `new_values` sean las claves del diccionario y algunas tuplas de color _Red Green Blue Alpha_ (RGBA) (en español, rojo verde azul alfa) utilizadas frecuentemente para este tipo de datos sean los valores del diccionario. Utilizaremos variantes del código de la siguiente celda para actualizar `layout_opts` de forma que los gráficos generados para varias capas/bandas de los productos de datos DSWx tengan leyendas adecuadas.
+
+```{code-cell} python
+COLORS = {
+0: (255, 255, 255, 0.0), # No Water
+1: (0, 0, 255, 1.0), # Open Water
+2: (180, 213, 244, 1.0), # Partial Surface Water
+3: ( 0, 255, 255, 1.0), # Snow/Ice
+4: (175, 175, 175, 1.0), # Cloud/Cloud Shadow
+5: ( 0, 0, 127, 0.5), # Ocean Masked
+}
+
+c_labels = ["No Water", "Open Water", "Partial Surface Water", "Snow/Ice", "Cloud/Cloud Shadow", "Ocean Masked"]
+c_ticks = list(COLORS.keys())
+limits = (c_ticks[0]-0.5, c_ticks[-1]+0.5)
+
+print(c_ticks)
+print(c_labels)
+```
+
+Para utilizar este mapa de colores, estas marcas y estas etiquetas en una barra de colores, creamos un diccionario `c_bar_opts` que contiene los objetos que hay que pasar al motor de renderizado Bokeh.
+
+```{code-cell} python editable=true slideshow={"slide_type": ""}
+c_bar_opts = dict( ticker=FixedTicker(ticks=c_ticks),
+ major_label_overrides=dict(zip(c_ticks, c_labels)),
+ major_tick_line_width=0, )
+```
+
+Debemos actualizar los diccionarios `image_opts` y `layout_opts` para incluir los datos relevantes para el mapa de colores.
+
+```{code-cell} python editable=true slideshow={"slide_type": ""}
+image_opts.update({ 'cmap': list(COLORS.values()),
+ 'clim': limits,
+ 'colorbar': True
+ })
+
+layout_opts.update(dict(title='B01_WTR', colorbar_opts=c_bar_opts))
+```
+
+Finalmente, podemos renderizar un gráfico rápido para asegurarnos de que la barra de colores se produce con etiquetas adecuadas.
+
+```{code-cell} python
+steps = 100
+subset = slice(0, None, steps)
+view = b01_wtr.isel(longitude=subset, latitude=subset)
+view.hvplot.image( **image_opts).opts(frame_width=500, frame_height=500, **layout_opts)
+```
+
+Por último, podemos definir un mapa base, esta vez utilizando mosaicos de [ESRI](https://www.esri.com). Esta vez, graficaremos el ráster a resolución completa (es decir, no nos molestaremos en utilizar `isel` para seleccionar primero un corte de menor resolución del ráster).
+
+```{code-cell} python editable=true slideshow={"slide_type": ""}
+# Creates basemap
+base = gv.tile_sources.EsriTerrain.opts(padding=0.1, alpha=0.25)
+b01_wtr.hvplot(**image_opts).opts(**layout_opts) * base
+```
+
+
+
+Este cuaderno computacional proporciona una visión general de cómo visualizar datos extraídos de productos de datos OPERA DSWx almacenados localmente. Ahora estamos listos para automatizar la búsqueda de dichos productos en la nube utilizando la API PySTAC.
+
+----
+
+
diff --git a/book/es-419/03_Using_NASA_EarthData/03_Using_PySTAC.md b/book/es-419/03_Using_NASA_EarthData/03_Using_PySTAC.md
new file mode 100644
index 0000000..8cf249a
--- /dev/null
+++ b/book/es-419/03_Using_NASA_EarthData/03_Using_PySTAC.md
@@ -0,0 +1,575 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Uso de la API de PySTAC
+
+
+
+En el sitio web [Earthdata Search](https://search.earthdata.nasa.gov) de la NASA se puede buscar una gran cantidad de datos. El enlace anterior se conecta a una interfaz gráfica de usuario (GUI, por sus siglas en inglés de _Graphical User Interface_) para buscar en los [catálogos activos espaciotemporales (STACs, por sus siglas en inglés de _SpatioTemporal Asset Catalogs_)](https://stacspec.org/) al especificar un área de interés (AOI, por sus siglas en inglés de _Area of Interest_) y una _ventana temporal_ o un _intervalo de fechas_.
+
+En pos de la reproducibilidad, se busca que las personas usuarias sean capaces de buscar en los catálogos de activos de manera programática. Aquí es donde entra en juego la librería [PySTAC](https://pystac.readthedocs.io/en/stable/).
+
+
+
+***
+
+## Esquema de las etapas del análisis
+
+
+
+- Identificación de los parámetros de búsqueda
+ - AOI, ventana temporal
+ - Endpoint, proveedor, identificador del catálogo ("nombre corto")
+- Obtención de los resultados de la búsqueda
+ - Exploración de datos, análisis para identificar características, bandas de interés
+ - Almacenar los resultados en un DataFrame para facilitar la exploración
+- Explorar y refinar los resultados de la búsqueda
+ - Identificar los granos de mayor valor
+ - Filtrar los granos anómalos con una contribución mínima
+ - Combinar los granos filtrados correspondientes en un DataFrame
+ - Identificar el tipo de salida que se quiere obtener
+- Procesamiento de los datos para generar resultados relevantes
+ - Descargar los granos relevantes en un tipo de dato DataArray de la libreria Xarray, apilados adecuadamente
+ - Realizar los cálculos intermedios necesarios
+ - Integrar los fragmentos de datos relevantes en una visualización
+
+
+
+***
+
+## Identificar los parámetros de búsqueda
+
+### Definir el AOI y el rango de fechas
+
+
+
+Comenzaremos tomando en cuenta un ejemplo concreto. [Las fuertes lluvias afectaron gravemente al sureste de Texas en mayo de 2024](https://www.texastribune.org/2024/05/03/texas-floods-weather-harris-county/), provocando [inundaciones y causando importantes daños materiales y humanos](https://www.texastribune.org/series/east-texas-floods-2024/).
+
+Como es usual, se requiere la importación de ciertas librerías relevantes. Las dos primeras celdas son familiares (relacionadas con las herramientas de análisis y la visualización de los datos que ya se examinaron). La tercera celda incluye la importación de la biblioteca `pystac_client` y de la biblioteca `gdal`, seguidas de algunos ajustes necesarios para utilizar la [Biblioteca de Abstracción de Datos Geoespaciales (GDAL, por sus siglas en inglés, Geospatial Data Abstraction Library)](https://gdal.org). Estos detalles en la configuración permiten que las sesiones de tu cuaderno computacional interactúen sin problemas con las fuentes remotas de datos geoespaciales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+# data wrangling imports
+import numpy as np
+import pandas as pd
+import xarray as xr
+import rioxarray as rio
+import rasterio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas
+import hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# STAC imports to retrieve cloud data
+from pystac_client import Client
+from osgeo import gdal
+# GDAL setup for accessing cloud data
+gdal.SetConfigOption('GDAL_HTTP_COOKIEFILE','~/.cookies.txt')
+gdal.SetConfigOption('GDAL_HTTP_COOKIEJAR', '~/.cookies.txt')
+gdal.SetConfigOption('GDAL_DISABLE_READDIR_ON_OPEN','EMPTY_DIR')
+gdal.SetConfigOption('CPL_VSIL_CURL_ALLOWED_EXTENSIONS','TIF, TIFF')
+```
+
+
+
+A continuación, definiremos los parámetros de búsqueda geográfica para poder recuperar los datos correspondientes a ese evento de inundación. Esto consiste en especificar un _AOI_ y un _intervalo de fechas_.
+
+- El AOI se especifica como un rectángulo de coordenadas de longitud-latitud en una única 4-tupla con la forma
+ $$({\mathtt{longitude}}_{\mathrm{min}},{\mathtt{latitude}}_{\mathrm{min}},{\mathtt{longitude}}_{\mathrm{max}},{\mathtt{latitude}}_{\mathrm{max}}),$$
+ por ejemplo, las coordenadas de la esquina inferior izquierda seguidas de las coordenadas de la esquina superior derecha.
+- El intervalo de fechas se especifica como una cadena de la forma
+ $${\mathtt{date}_{\mathrm{start}}}/{\mathtt{date}_{\mathrm{end}}},$$
+ donde las fechas se especifican en el formato estándar `YYYY-MM-DD`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Center of the AOI
+livingston_tx_lonlat = (-95.09,30.69) # (lon, lat) form
+```
+
+
+
+Escribiremos algunas funciones cortas para encapsular la lógica de nuestros flujos de trabajo genéricos. Para el código de investigación, estas se colocarían en archivos de Python. Por practicidad, incrustaremos las funciones en este cuaderno y en otros para que puedan ejecutarse correctamente con dependencias mínimas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle with given center of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding-box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to plot an AOI or bounding-box
+def plot_bbox(bbox):
+ '''Given bounding-box, returns GeoViews plot of Rectangle & Point at center
+ + bbox: bounding-box specified as (lon_min, lat_min, lon_max, lat_max)
+ Assume longitude-latitude coordinates.
+ '''
+ # These plot options are fixed but can be over-ridden
+ point_opts = opts.Points(size=12, alpha=0.25, color='blue')
+ rect_opts = opts.Rectangles(line_width=0, alpha=0.1, color='red')
+ lon_lat = (0.5*sum(bbox[::2]), 0.5*sum(bbox[1::2]))
+ return (gv.Points([lon_lat]) * gv.Rectangles([bbox])).opts(point_opts, rect_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+AOI = make_bbox(livingston_tx_lonlat, 0.5, 0.25)
+basemap = gv.tile_sources.OSM.opts(width=500, height=500)
+plot_bbox(AOI) * basemap
+```
+
+
+
+Agreguemos un intervalo de fechas. Las inundaciones ocurrieron principalmente entre el 30 de abril y el 2 de mayo. Estableceremos una ventana temporal más larga que cubra los meses de abril y mayo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+start_date, stop_date = '2024-04-01', '2024-05-31'
+DATE_RANGE = f'{start_date}/{stop_date}'
+```
+
+
+
+Por último, se crea un un diccionario `search_params` que almacene el AOI y el intervalo de fechas. Este diccionario se utilizará para buscar datos en los STACs.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+search_params = dict(bbox=AOI, datetime=DATE_RANGE)
+print(search_params)
+```
+
+***
+
+## Obtención de los resultados de búsqueda
+
+### Ejecución de una búsqueda con la API PySTAC
+
+
+
+Para iniciar una búsqueda de datos se necesitan tres datos más: el _Endpoint_ (una URL), el _Proveedor_ (una cadena que representa una ruta que extiende el _Endpoint_) y los _Identificadores de la colección_ (una lista de cadenas que hacen referencia a catálogos específicos). Generalmente, debemos probar con el [sitio web de Earthdata Search](https://search.earthdata.nasa.gov) de la NASA para determinar correctamente los valores para los productos de datos específicos que queremos recuperar. El [repositorio de GitHub de la NASA CMR STAC también supervisa los problemas](https://github.com/nasa/cmr-stac/issues) relacionados con la API para las consultas de búsqueda de EarthData Cloud.
+
+Para la búsqueda de productos de datos DSWx que se quiere ejecutar, estos parámetros son los que se definen en la siguiente celda de código.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+ENDPOINT = 'https://cmr.earthdata.nasa.gov/stac' # base URL for the STAC to search
+PROVIDER = 'POCLOUD'
+COLLECTIONS = ["OPERA_L3_DSWX-HLS_V1_1.0"]
+# Update the dictionary opts with list of collections to search
+search_params.update(collections=COLLECTIONS)
+print(search_params)
+```
+
+
+
+Una vez que se definieron los parámetros de búsqueda en el diccionario de Python `search_params`, se puede instanciar un `Cliente` y buscar en el catálogo espacio-temporal de activos utilizando el método `Client.search`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+catalog = Client.open(f'{ENDPOINT}/{PROVIDER}/')
+search_results = catalog.search(**search_params)
+print(f'{type(search_results)=}\n',search_results)
+```
+
+
+
+El objeto `search_results` que se obtuvo al llamar al método `search` es del tipo `ItemSearch`. Para recuperar los resultados, invocamos al método `items` y convertimos el resultado en una `list` de Python que asociaremos al identificador `granules`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+granules = list(search_results.items())
+print(f"Number of granules found with tiles overlapping given AOI: {len(granules)}")
+```
+
+
+
+Se analiza el contenido de la lista `granules`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+granule = granules[0]
+print(f'{type(granule)=}')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+granule
+```
+
+
+
+El objeto `granule` tiene una representación de salida enriquecida en este cuaderno computacional de Jupyter. Podemos ampliar los atributos en la celda de salida haciendo clic en los triángulos.
+
+
+
+El término _grano_ se refiere a una colección de archivos de datos (datos ráster en este caso), todos ellos asociados a datos sin procesar adquiridos por un satélite concreto en una fecha y hora fija sobre un mosaico geográfico concreto. Hay una gran variedad de atributos interesantes asociados con este grano.
+
+- properties['datetime']: una cadena que representa la hora de adquisición de los datos de los archivos de datos ráster de este grano,
+- properties['eo:cloud_cover']: el porcentaje de píxeles oscurecidos por nubes y sombras de nubes en los archivos de datos ráster de este grano, y
+- `assets`: un `dict` de Python cuyos valores resumen las bandas o niveles de los datos ráster asociados con este gránulo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f"{type(granule.properties)=}\n")
+print(f"{granule.properties['datetime']=}\n")
+print(f"{granule.properties['eo:cloud_cover']=}\n")
+print(f"{type(granule.assets)=}\n")
+print(f"{granule.assets.keys()=}\n")
+```
+
+
+
+Cada objeto en `granule.assets` es una instancia de la clase `Asset` que tiene un atributo `href`. Es el atributo `href` el que nos indica dónde localizar un archivo GeoTiff asociado con el activo de este gránulo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+for a in granule.assets:
+ print(f"{a=}\t{type(granule.assets[a])=}")
+ print(f"{granule.assets[a].href=}\n\n")
+```
+
+
+
+Además, el `Item` tiene un atributo `.id` que almacena una cadena de caracteres. Al igual que ocurre con los nombres de archivos asociados a los productos OPERA, esta cadena `.id` contiene el identificador de un mosaico geográfico MGRS. Podemos extraer ese identificador aplicando manipulación de cadenas con Python al atributo `.id` del gránulo. Se realiza y se almacena el resultado en la variable `tile_id`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(granule.id)
+tile_id = granule.id.split('_')[3]
+print(f"{tile_id=}")
+```
+
+***
+
+### Resumiendo los resultados de la búsqueda en un DataFrame
+
+
+
+Los detalles de los resultados de la búsqueda son complicados de analizar de esta manera. Se extraen algunos campos concretos de los gránulos obtenidos en un `DataFrame` de Pandas utilizando una función de Python. Definiremos la función aquí y la reutilizaremos en cuadernos posteriores.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# utility to extract search results into a Pandas DataFrame
+def search_to_dataframe(search):
+ '''Constructs Pandas DataFrame from PySTAC Earthdata search results.
+ DataFrame columns are determined from search item properties and assets.
+ 'asset': string identifying an Asset type associated with a granule
+ 'href': data URL for file associated with the Asset in a given row.'''
+ granules = list(search.items())
+ assert granules, "Error: empty list of search results"
+ props = list({prop for g in granules for prop in g.properties.keys()})
+ tile_ids = map(lambda granule: granule.id.split('_')[3], granules)
+ rows = (([g.properties.get(k, None) for k in props] + [a, g.assets[a].href, t])
+ for g, t in zip(granules,tile_ids) for a in g.assets )
+ df = pd.concat(map(lambda x: pd.DataFrame(x, index=props+['asset','href', 'tile_id']).T, rows),
+ axis=0, ignore_index=True)
+ assert len(df), "Empty DataFrame"
+ return df
+```
+
+
+
+Invocar `search_to_dataframe` en `search_results` codifica la mayor parte de la información importante de la búsqueda como un Pandas `DataFrame`, tal como se muestra a continuación.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = search_to_dataframe(search_results)
+df.head()
+```
+
+
+
+El método `DataFrame.info` nos permite examinar la estructura de este DataFrame.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.info()
+```
+
+
+
+Se limpia el DataFrame que contiene los resultados de búsqueda. Esto podría estar incluido en una función, pero vale la pena saber cómo se hace esto con Pandas de manera interactiva.
+
+En primer lugar, para estos resultados, solo es necesaria una columna `Datetime`, se pueden eliminar las demás.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = df.drop(['start_datetime', 'end_datetime'], axis=1)
+df.info()
+```
+
+
+
+A continuación, se arregla el esquema del `DataFrame` `df` convirtiendo las columnas en tipos de datos sensibles. También será conveniente utilizar la marca de tiempo de la adquisición como índice del DataFrame. Se realiza utilizando el método `DataFrame.set_index`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df['datetime'] = pd.DatetimeIndex(df['datetime'])
+df['eo:cloud_cover'] = df['eo:cloud_cover'].astype(np.float16)
+str_cols = ['asset', 'href', 'tile_id']
+for col in str_cols:
+ df[col] = df[col].astype(pd.StringDtype())
+df = df.set_index('datetime').sort_index()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.info()
+```
+
+
+
+Como resultado se obtiene un DataFrame con un esquema conciso que se puede utilizar para manipulaciones posteriores. Agrupar los resultados de la búsqueda STAC en un `DataFrame` de Pandas de forma razonable es un poco complicado. Varias de las manipulaciones anteriores podrían haberse incluido en la función `search_to_dataframe`. Pero, dado que los resultados de búsqueda de la API de STAC aún están evolucionando, actualmente es mejor ser flexible y utilizar Pandas de forma interactiva para trabajar con los resultados de búsqueda. Se verá esto con más detalle en ejemplos posteriores.
+
+
+
+***
+
+## Explorar y refinar los resultados de búsqueda
+
+
+
+Si se examina la columna numérica `eo:cloud_cover` del DataFrame `df`, se pueden recopilar estadísticas utilizando agregaciones estándar y el método `DataFrame.agg`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df['eo:cloud_cover'].agg(['min','mean','median','max'])
+```
+
+
+
+Observa que hay varias entradas `nan` en esta columna. Las funciones de agregación estadística de Pandas suelen ser "`nan`-aware", esto significa que ignoran implícitamente las entradas `nan` al calcular las estadísticas.
+
+
+
+### Filtrado del DataFrame de búsqueda con Pandas
+
+
+
+Como primera operación de filtrado, se mantienen solo las filas para las que la cobertura de las nubes es inferior al 50%.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df_clear = df.loc[df['eo:cloud_cover']<50]
+df_clear
+```
+
+
+
+Para esta consulta de búsqueda, cada gránulo DSWX comprende datos ráster para diez bandas o niveles. Se puede ver esto aplicando el método Pandas `Series.value_counts` a la columna `asset`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df_clear.asset.value_counts()
+```
+
+
+
+Se filtran las filas que corresponden a la banda `B01_WTR` del producto de datos DSWx. La función de Pandas `DataFrame.str` hace que esta operación sea sencilla. Se nombra al `DataFrame` filtrado como `b01_wtr`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+b01_wtr = df_clear.loc[df_clear.asset.str.contains('B01_WTR')]
+b01_wtr.info()
+b01_wtr.asset.value_counts()
+```
+
+
+
+También se puede observar que hay varios mosaicos geográficos asociados a los gránulos encontrados que intersecan el AOI proporcionado.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+b01_wtr.tile_id.value_counts()
+```
+
+
+
+Recuerda que estos códigos se refieren a mosaicos geográficos MGRS especificados en un sistema de coordenadas concreto. Como se identifican estos códigos en la columna `tile_id`, se puede filtrar las filas que corresponden, por ejemplo, a los archivos recopilados sobre el mosaico T15RUQ del MGRS:
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+b01_wtr_t15ruq = b01_wtr.loc[b01_wtr.tile_id=='T15RUQ']
+b01_wtr_t15ruq
+```
+
+
+
+Se obtiene un `DataFrame` `b01_wtr_t15ruq` mucho más corto que resume las ubicaciones remotas de los archivos (por ejemplo, GeoTiffs) que almacenan datos ráster para la banda de aguas superficiales `B01_WTR` en el mosaico MGRS `T15RUQ` recopilados en varias marcas de tiempo que se encuentran dentro de la ventana de tiempo que especificamos. Se puede utilizar este DataFrame para descargar esos archivos para su análisis o visualización.
+
+
+
+***
+
+## Procesamiento de datos para obtener resultados relevantes
+
+### Apilamiento de los datos
+
+
+
+Se cuenta con un `DataFrame` que identifica archivos remotos específicos de datos ráster. El siguiente paso es combinar estos datos ráster en una estructura de datos adecuada para el análisis. El Xarray `DataArray` es adecuado en este caso. La combinación puede generarse utilizando la función `concat` de Xarray. La función `urls_to_stack` en la siguiente celda es larga pero no es complicada. Toma un `DataFrame` con marcas de tiempo en el índice y una columna etiquetada `href` de las URL, lee los archivos asociados a esas URL uno a uno, y apila las matrices bidimensionales relevantes de datos ráster en una matriz tridimensional.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+def urls_to_stack(granule_dataframe):
+ '''Processes DataFrame of PySTAC search results (with OPERA tile URLs) &
+ returns stacked Xarray DataArray (dimensions time, latitude, & longitude)'''
+
+ stack = []
+ for i, row in granule_dataframe.iterrows():
+ with rasterio.open(row.href) as ds:
+ # extract CRS string
+ crs = str(ds.crs).split(':')[-1]
+ # extract the image spatial extent (xmin, ymin, xmax, ymax)
+ xmin, ymin, xmax, ymax = ds.bounds
+ # the x and y resolution of the image is available in image metadata
+ x_res = np.abs(ds.transform[0])
+ y_res = np.abs(ds.transform[4])
+ # read the data
+ img = ds.read()
+ # Ensure img has three dimensions (bands, y, x)
+ if img.ndim == 2:
+ img = np.expand_dims(img, axis=0)
+ lon = np.arange(xmin, xmax, x_res)
+ lat = np.arange(ymax, ymin, -y_res)
+ bands = np.arange(img.shape[0])
+ da = xr.DataArray(
+ data=img,
+ dims=["band", "lat", "lon"],
+ coords=dict(
+ lon=(["lon"], lon),
+ lat=(["lat"], lat),
+ time=i,
+ band=bands
+ ),
+ attrs=dict(
+ description="OPERA DSWx B01",
+ units=None,
+ ),
+ )
+ da.rio.write_crs(crs, inplace=True)
+ stack.append(da)
+ return xr.concat(stack, dim='time').squeeze()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+stack = urls_to_stack(b01_wtr_t15ruq)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+### Crear una visualización de los datos
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Define a colormap with RGBA tuples
+COLORS = [(150, 150, 150, 0.1)]*256 # Setting all values to gray with low opacity
+COLORS[0] = (0, 255, 0, 0.1) # Not-water class to green
+COLORS[1] = (0, 0, 255, 1) # Open surface water
+COLORS[2] = (0, 0, 255, 1) # Partial surface water
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='lon',
+ y='lat',
+ project=True,
+ rasterize=True,
+ cmap=COLORS,
+ colorbar=False,
+ tiles = gv.tile_sources.OSM,
+ widget_location='bottom',
+ frame_width=500,
+ frame_height=500,
+ xlabel='Longitude (degrees)',
+ ylabel='Latitude (degrees)',
+ title = 'DSWx data for May 2024 Texas floods',
+ fontscale=1.25
+ )
+```
+
+
+
+Visualizar las imágenes completas puede consumir mucha memoria. Se utiliza el método Xarray `DataArray.isel` para extraer un trozo del arreglo `stack` con menos píxeles. Esto permitirá un rápido renderizado y desplazamiento.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+view = stack.isel(lon=slice(3000,None), lat=slice(3000,None))
+view.hvplot.image(**image_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack.hvplot.image(**image_opts) # Construct view from all slices.
+```
+
+
+
+Antes de continuar, recuerda apagar el kernel de este cuaderno computacional para liberar memoria para otros cálculos.
+
+
+
+***
+
+
+
+Este cuaderno computacional proporciona principalmente un ejemplo para ilustrar el uso de la API de PySTAC.
+
+En los siguientes cuadernos computacionales, utilizaremos este flujo de trabajo general:
+
+1. Establecer una consulta de búsqueda mediante la identificación de un _AOI_ particular y un _intervalo de fechas_.
+2. Identificar un _endpoint_, un _proveedor_ y un _catálogo de activos_ adecuados, y ejecutar la búsqueda utilizando `pystac.Client`.
+3. Convertir los resultados de la búsqueda en un DataFrame de Pandas que contenga los principales campos de interés.
+4. Utilizar el DataFrame resultante para filtrar los archivos de datos remotos más relevantes necesarios para el análisis y/o la visualización.
+5. Ejecutar el análisis y/o la visualización utilizando el DataFrame para recuperar los datos requeridos.
+
+
diff --git a/book/es-419/04_Case_Studies/00_Template.md b/book/es-419/04_Case_Studies/00_Template.md
new file mode 100644
index 0000000..55ea976
--- /dev/null
+++ b/book/es-419/04_Case_Studies/00_Template.md
@@ -0,0 +1,232 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Plantilla para el uso de del servicio cloud ofrecido por EarthData
+
+## Esquema de los pasos para el análisis
+
+
+
+- Identificación de los parámetros de búsqueda
+ - Área de interés (AOI, por las siglas en inglés de _area of interest_) y ventana temporal
+ - _Endpoint_, proveedor, identificador del catálogo ("nombre corto")
+- Obtención de los resultados de la búsqueda
+ - Exploracion, análisis para identificar características, bandas de interés
+ - Almacenar los resultados en un DataFrame para facilitar la exploración
+- Explorar y refinar los resultados de la búsqueda
+ - Identificar los gránulos de mayor valor
+ - Filtrar los gránulos atípicos con mínima contribución
+ - Combinar los gránulos filtrados relevantes en un DataFrame
+ - Identificar el tipo de salida a generar
+- Procesar los datos para obtener resultados relevantes
+ - Descargar los gránulos relevantes en Xarray DataArray, apilados adecuadamente
+ - Realizar los cálculos intermedios necesarios
+ - Combinar los datos relevantes en una visualización
+
+
+
+***
+
+### Importación preliminar de librerías
+
+```python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+# data wrangling imports
+import numpy as np
+import pandas as pd
+import xarray as xr
+import rioxarray as rio
+import rasterio
+```
+
+```python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas
+import hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```python jupyter={"source_hidden": false}
+# STAC imports to retrieve cloud data
+from pystac_client import Client
+from osgeo import gdal
+# GDAL setup for accessing cloud data
+gdal.SetConfigOption('GDAL_HTTP_COOKIEFILE','~/.cookies.txt')
+gdal.SetConfigOption('GDAL_HTTP_COOKIEJAR', '~/.cookies.txt')
+gdal.SetConfigOption('GDAL_DISABLE_READDIR_ON_OPEN','EMPTY_DIR')
+gdal.SetConfigOption('CPL_VSIL_CURL_ALLOWED_EXTENSIONS','TIF, TIFF')
+```
+
+### Funciones prácticas
+
+Estas funciones podrían incluirse en archivos de módulos para proyectos de investigación más evolucionados. Para fines didácticos, se incluyen en este cuaderno computacional.
+
+```python jupyter={"source_hidden": false}
+# simple utility to make a rectangle with given center of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding-box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+```python jupyter={"source_hidden": false}
+# simple utility to plot an AOI or bounding-box
+def plot_bbox(bbox):
+ '''Given bounding-box, returns GeoViews plot of Rectangle & Point at center
+ + bbox: bounding-box specified as (lon_min, lat_min, lon_max, lat_max)
+ Assume longitude-latitude coordinates.
+ '''
+ # These plot options are fixed but can be over-ridden
+ point_opts = opts.Points(size=12, alpha=0.25, color='blue')
+ rect_opts = opts.Rectangles(line_width=0, alpha=0.1, color='red')
+ lon_lat = (0.5*sum(bbox[::2]), 0.5*sum(bbox[1::2]))
+ return (gv.Points([lon_lat]) * gv.Rectangles([bbox])).opts(point_opts, rect_opts)
+```
+
+```python jupyter={"source_hidden": false}
+# utility to extract search results into a Pandas DataFrame
+def search_to_dataframe(search):
+ '''Constructs Pandas DataFrame from PySTAC Earthdata search results.
+ DataFrame columns are determined from search item properties and assets.
+ 'asset': string identifying an Asset type associated with a granule
+ 'href': data URL for file associated with the Asset in a given row.'''
+ granules = list(search.items())
+ assert granules, "Error: empty list of search results"
+ props = list({prop for g in granules for prop in g.properties.keys()})
+ tile_ids = map(lambda granule: granule.id.split('_')[3], granules)
+ rows = (([g.properties.get(k, None) for k in props] + [a, g.assets[a].href, t])
+ for g, t in zip(granules,tile_ids) for a in g.assets )
+ df = pd.concat(map(lambda x: pd.DataFrame(x, index=props+['asset','href', 'tile_id']).T, rows),
+ axis=0, ignore_index=True)
+ assert len(df), "Empty DataFrame"
+ return df
+```
+
+```python jupyter={"source_hidden": false}
+# utility to process DataFrame of search results & return DataArray of stacked raster images
+def urls_to_stack(granule_dataframe):
+ '''Processes DataFrame of PySTAC search results (with OPERA tile URLs) &
+ returns stacked Xarray DataArray (dimensions time, latitude, & longitude)'''
+
+ stack = []
+ for i, row in granule_dataframe.iterrows():
+ with rasterio.open(row.href) as ds:
+ # extract CRS string
+ crs = str(ds.crs).split(':')[-1]
+ # extract the image spatial extent (xmin, ymin, xmax, ymax)
+ xmin, ymin, xmax, ymax = ds.bounds
+ # the x and y resolution of the image is available in image metadata
+ x_res = np.abs(ds.transform[0])
+ y_res = np.abs(ds.transform[4])
+ # read the data
+ img = ds.read()
+ # Ensure img has three dimensions (bands, y, x)
+ if img.ndim == 2:
+ img = np.expand_dims(img, axis=0)
+ lon = np.arange(xmin, xmax, x_res)
+ lat = np.arange(ymax, ymin, -y_res)
+ bands = np.arange(img.shape[0])
+ da = xr.DataArray(
+ data=img,
+ dims=["band", "lat", "lon"],
+ coords=dict(
+ lon=(["lon"], lon),
+ lat=(["lat"], lat),
+ time=i,
+ band=bands
+ ),
+ attrs=dict(
+ description="OPERA DSWx B01",
+ units=None,
+ ),
+ )
+ da.rio.write_crs(crs, inplace=True)
+ stack.append(da)
+ return xr.concat(stack, dim='time').squeeze()
+```
+
+***
+
+## Identificación de los parámetros de búsqueda
+
+```python jupyter={"source_hidden": false}
+AOI = ...
+DATE_RANGE = ...
+```
+
+```python jupyter={"source_hidden": false}
+# Optionally plot the AOI
+```
+
+```python jupyter={"source_hidden": false}
+search_params = dict(bbox=AOI, datetime=DATE_RANGE)
+print(search_params)
+```
+
+***
+
+## Obtención de los resultados de la búsqueda
+
+```python jupyter={"source_hidden": false}
+ENDPOINT = ...
+PROVIDER = ...
+COLLECTIONS = ...
+# Update the dictionary opts with list of collections to search
+search_params.update(collections=COLLECTIONS)
+print(search_params)
+```
+
+```python jupyter={"source_hidden": false}
+catalog = Client.open(f'{ENDPOINT}/{PROVIDER}/')
+search_results = catalog.search(**search_params)
+```
+
+```python
+df = search_to_dataframe(search_results)
+df.head()
+```
+
+Limpiar el DataFrame `df` de forma que tenga sentido (por ejemplo, eliminando columnas/filas innecesarias, convirtiendo columnas en tipos de datos fijos, estableciendo un índice, etc.).
+
+```python
+```
+
+***
+
+## Exploración y refinamiento de los resultados de la búsqueda
+
+
+
+Consiste en filtrar filas o columnas adecuadamente para limitar los resultados de la búsqueda a los archivos de datos ráster más relevantes para el análisis y/o la visualización. Esto puede significar enfocarse en determinadas regiones geográficos, bandas específicas del producto de datos, determinadas fechas o períodos, etc.
+
+
+
+```python
+```
+
+***
+
+## Procesamiento de los datos para obtener resultados relevantes
+
+Esto puede incluir apilar matrices bidimensionales en una matriz tridimensional, combinar imágenes ráster de mosaicos adyacentes en uno solo, etc.
+
+```python
+```
+
+***
diff --git a/book/es-419/04_Case_Studies/01_Greece_Wildfires.md b/book/es-419/04_Case_Studies/01_Greece_Wildfires.md
new file mode 100644
index 0000000..ae9b379
--- /dev/null
+++ b/book/es-419/04_Case_Studies/01_Greece_Wildfires.md
@@ -0,0 +1,481 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Incendios forestales en Grecia
+
+
+
+En este ejemplo, recuperaremos los datos asociados a los [incendios forestales en Grecia en el 2023](https://es.wikipedia.org/wiki/Incendios_forestales_en_Grecia_de_2023) para comprender su evolución y extensión. Generaremos una serie temporal asociada a estos datos y dos visualizaciones del evento.
+
+En particular, analizaremos la zona que está alrededor de la ciudad de [Alexandroupolis](https://es.wikipedia.org/wiki/Alejandr%C3%B3polis), que se vio gravemente afectada por incendios forestales, con la consiguiente pérdida de vidas, propiedades y zonas boscosas.
+
+---
+
+
+
+## Esquema de las etapas del análisis
+
+
+
+- Identificación de los parámetros de búsqueda
+ - AOI, ventana temporal
+ - _Endpoint_, proveedor, identificador del catálogo ("nombre corto")
+- Obtención de los resultados de la búsqueda
+ - Inspección, análisis para identificar características, bandas de interés
+ - Almacenamos los resultados en un DataFrame para facilitar la exploración
+- Exploración y refinamiento de los resultados de la búsqueda
+ - Identificar los gránulos de mayor valor
+ - Filtrar los gránulos extraños con una contribución mínima
+ - Integrar los gránulos filtrados en un DataFrame
+ - Identificar el tipo de resultado que se quiere generar
+- Procesamiento de los datos para generar resultados relevantes
+ - Descargar los gránulos relevantes en Xarray DataArray, apilados adecuadamente
+ - Llevar a cabo los cálculos intermedios necesarios
+ - Unir los fragmentos de datos relevantes en la visualización
+
+---
+
+
+
+### Importación preliminar
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+import numpy as np, pandas as pd, xarray as xr
+import rioxarray as rio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# STAC imports to retrieve cloud data
+from pystac_client import Client
+from osgeo import gdal
+# GDAL setup for accessing cloud data
+gdal.SetConfigOption('GDAL_HTTP_COOKIEFILE','~/.cookies.txt')
+gdal.SetConfigOption('GDAL_HTTP_COOKIEJAR', '~/.cookies.txt')
+gdal.SetConfigOption('GDAL_DISABLE_READDIR_ON_OPEN','EMPTY_DIR')
+gdal.SetConfigOption('CPL_VSIL_CURL_ALLOWED_EXTENSIONS','TIF, TIFF')
+```
+
+### Funciones prácticas
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle with given center of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding-box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to plot an AOI or bounding-box
+def plot_bbox(bbox):
+ '''Given bounding-box, returns GeoViews plot of Rectangle & Point at center
+ + bbox: bounding-box specified as (lon_min, lat_min, lon_max, lat_max)
+ Assume longitude-latitude coordinates.
+ '''
+ # These plot options are fixed but can be over-ridden
+ point_opts = opts.Points(size=12, alpha=0.25, color='blue')
+ rect_opts = opts.Rectangles(line_width=2, alpha=0.1, color='red')
+ lon_lat = (0.5*sum(bbox[::2]), 0.5*sum(bbox[1::2]))
+ return (gv.Points([lon_lat]) * gv.Rectangles([bbox])).opts(point_opts, rect_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# utility to extract search results into a Pandas DataFrame
+def search_to_dataframe(search):
+ '''Constructs Pandas DataFrame from PySTAC Earthdata search results.
+ DataFrame columns are determined from search item properties and assets.
+ 'asset': string identifying an Asset type associated with a granule
+ 'href': data URL for file associated with the Asset in a given row.'''
+ granules = list(search.items())
+ assert granules, "Error: empty list of search results"
+ props = list({prop for g in granules for prop in g.properties.keys()})
+ tile_ids = map(lambda granule: granule.id.split('_')[3], granules)
+ rows = (([g.properties.get(k, None) for k in props] + [a, g.assets[a].href, t])
+ for g, t in zip(granules,tile_ids) for a in g.assets )
+ df = pd.concat(map(lambda x: pd.DataFrame(x, index=props+['asset','href', 'tile_id']).T, rows),
+ axis=0, ignore_index=True)
+ assert len(df), "Empty DataFrame"
+ return df
+```
+
+
+
+Estas funciones podrían incluirse en archivos modular para proyectos de investigación más evolucionados. Para fines didácticos, se incluyen en este cuaderno computacional.
+
+---
+
+
+
+## Identificación de los parámetros de búsqueda
+
+```{code-cell} python jupyter={"source_hidden": false}
+dadia_forest = (26.18, 41.08)
+AOI = make_bbox(dadia_forest, 0.1, 0.1)
+DATE_RANGE = '2023-08-01/2023-09-30'.split('/')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Optionally plot the AOI
+basemap = gv.tile_sources.ESRI(width=500, height=500, padding=0.1, alpha=0.25)
+plot_bbox(AOI) * basemap
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+search_params = dict(bbox=AOI, datetime=DATE_RANGE)
+print(search_params)
+```
+
+---
+
+## Obtención de los resultados de búsqueda
+
+```{code-cell} python jupyter={"source_hidden": false}
+ENDPOINT = 'https://cmr.earthdata.nasa.gov/stac'
+PROVIDER = 'LPCLOUD'
+COLLECTIONS = ["OPERA_L3_DIST-ALERT-HLS_V1_1"]
+# Update the dictionary opts with list of collections to search
+search_params.update(collections=COLLECTIONS)
+print(search_params)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+catalog = Client.open(f'{ENDPOINT}/{PROVIDER}/')
+search_results = catalog.search(**search_params)
+```
+
+
+
+Como de costumbre, codificaremos el resultado de la búsqueda en un Pandas `DataFrame`, analizaremos los resultados, y haremos algunas transformaciones para limpiarlos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+df = search_to_dataframe(search_results)
+df.head()
+```
+
+
+
+Limpiaremos el `DataFrame` `df` de las formas típicas:
+
+- convertiendo la columna `datetime` en `DatetimeIndex`,
+- eliminando columnas de tipo `datetime` extrañas,
+- renombrando la columna `eo:cloud_cover` como `cloud_cover`,
+- convirtiendo la columna `cloud_cover` en valores de punto flotante, y
+- convertiendo las columnas restantes en cadenas de caracteres, y
+- estableciendo la columna `datetime` como `Index`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = df.drop(['end_datetime', 'start_datetime'], axis=1)
+df.datetime = pd.DatetimeIndex(df.datetime)
+df = df.rename(columns={'eo:cloud_cover':'cloud_cover'})
+df['cloud_cover'] = df['cloud_cover'].astype(np.float16)
+for col in ['asset', 'href', 'tile_id']:
+ df[col] = df[col].astype(pd.StringDtype())
+df = df.set_index('datetime').sort_index()
+df.info()
+```
+
+---
+
+## Exploración y refinamiento de los resultados de la búsqueda
+
+
+
+Examinemos el `DataFrame` `df` para comprender mejor los resultados de la búsqueda. En primer lugar, veamos cuántos mosaicos geográficos diferentes aparecen en los resultados de la búsqueda.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.tile_id.value_counts()
+```
+
+
+
+Entonces, el AOI se encuentra estrictamente dentro de un único mosaico geográfico MGRS llamado `T35TMF`. Analicemos la columna `asset`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.asset.value_counts().sort_values(ascending=False)
+```
+
+
+
+Algunos de los nombres de estos activos no son tan simples y ordenados como los que encontramos con los productos de datos DIST-ALERT. Sin embargo, podemos identificar fácilmente las subcadenas de caracteres útiles. En este caso, elegimos sólo las filas en las que la columna `asset` incluye `'VEG-DIST-STATUS'` como subcadena de caracteres.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+idx_veg_dist_status = df.asset.str.contains('VEG-DIST-STATUS')
+idx_veg_dist_status
+```
+
+
+
+Podemos utilizar esta `Series` booleana con el método de acceso `.loc` de Pandas para filtrar solo las filas que queremos (por ejemplo, las que se conectan a archivos de datos ráster que almacenan la banda `VEG-DIST-STATUS`). Posteriormente podemos eliminar la columna `asset` (que ya no es necesaria).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+veg_dist_status = df.loc[idx_veg_dist_status]
+veg_dist_status = veg_dist_status.drop('asset', axis=1)
+veg_dist_status
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(len(veg_dist_status))
+```
+
+
+
+Observe que algunas de las filas tienen la misma fecha pero horas diferentes (lo cual corresponde a varias observaciones en el mismo día del calendario UTC). Podemos agregar las URL en listas _remuestreando_ la serie temporal por día. Posteriormente podremos visualizar el resultado.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+by_day = veg_dist_status.resample('1d').href.apply(list)
+display(by_day)
+by_day.map(len).hvplot.scatter(grid=True).opts(title='# of observations')
+```
+
+
+
+Limpiemos la `Series` `by_day` filtrando las filas que tienen listas vacías (es decir, fechas en las que no se adquirieron datos).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+by_day = by_day.loc[by_day.map(bool)]
+by_day.map(len).hvplot.scatter(ylim=(0,2.1), grid=True).opts(title="# of observations")
+```
+
+
+
+Ahora podemos utilizar la serie `by_day` remuestreada para extraer datos ráster para su análisis.
+
+---
+
+
+
+## Procesamiento de los datos
+
+
+
+El incendio forestal cerca de Alexandroupolis comenzó alrededor del 21 de agosto y se propagó rápidamente, afectando en particular al cercano bosque de Dadia. En primer lugar, vamos a ensamblar un "cubo de datos" (por ejemplo, un arreglo apilado de rásters) a partir de los archivos remotos indexados en la serie Pandas `by_day`. Empezaremos seleccionando y cargando uno de los archivos GeoTIFF remotos para extraer los metadatos que se aplican a todos los rásteres asociados con este evento y este mosaico MGRS.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+href = by_day[0][0]
+data = rio.open_rasterio(href).rename(dict(x='longitude', y='latitude'))
+crs = data.rio.crs
+shape = data.shape
+```
+
+
+
+Antes de construir un `DataArray` apilado dentro de un bucle, definiremos un diccionario de Python llamado `template` que se utilizará para instanciar los cortes del arreglo. El diccionario `template` almacenará los metadatos extraídos del archivo GeoTIFF, especialmente las coordenadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+template = dict()
+template['coords'] = data.coords.copy()
+del template['coords']['band']
+template['coords'].update({'time': by_day.index.values})
+template['dims'] = ['time', 'longitude', 'latitude']
+template['attrs'] = dict(description=f"OPERA DSWX: VEG-DIST-STATUS", units=None)
+
+print(template)
+```
+
+
+
+Utilizaremos un bucle para construir un arreglo apilado de rásteres a partir de la serie Pandas `by_day` (cuyas entradas son listas de cadenas de caracteres, es decir, URIs). Si la lista tiene un único elemento, la URL se puede leer directamente utilizando `rasterio.open`; de lo contrario, la función [`rasterio.merge.merge`](https://rasterio.readthedocs.io/en/latest/api/rasterio.merge.html) combina varios archivos de datos ráster adquiridos el mismo día en una única imagen ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import rasterio
+from rasterio.merge import merge
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+rasters = []
+for date, hrefs in by_day.items():
+ n_files = len(hrefs)
+ if n_files > 1:
+ print(f"Merging {n_files} files for {date.strftime('%Y-%m-%d')}...")
+ raster, _ = merge(hrefs)
+ else:
+ print(f"Opening {n_files} file for {date.strftime('%Y-%m-%d')}...")
+ with rasterio.open(hrefs[0]) as ds:
+ raster = ds.read()
+ rasters.append(np.reshape(raster, newshape=shape))
+```
+
+
+
+Los datos acumulados en la lista de `rasters` se almacenan como arreglos de NumPy. Así, en vez de llamar a `xarray.concat`, realizamos una llamada a `numpy.concatenate` dentro de una llamada al constructor `xarray.DataArray`. Vinculamos el objeto creado al identificador `stack`, asegurándonos de incluir también la información CRS.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = xr.DataArray(data=np.concatenate(rasters, axis=0), **template)
+stack.rio.write_crs(crs, inplace=True)
+stack
+```
+
+
+
+La pila `DataArray` `stack` tiene `time`, `longitude` y `latitude` como principales dimensiones de coordenadas. Podemos utilizarla para hacer algunos cálculos y generar visualizaciones.
+
+---
+
+
+
+### Visualización del área dañada
+
+
+
+Para empezar, utilicemos los datos en `stack` para calcular la superficie total dañada. Los datos en `stack` provienen de la banda `VEG-DIST-STATUS` del producto DIST-ALERT. Interpretamos los valores de los píxeles en esta banda de la siguiente manera:
+
+- **0:** Sin alteración
+- **1:** La primera detección de alteraciones con cambio en la cobertura vegetal $<50\%$
+- **2:** Detección provisional de alteraciones con cambio en la cobertura vegetal $<50\%$
+- **3:** Detección confirmada de alteraciones con cambio en la cobertura vegetal $<50\%$
+- **4:** La primera detección de alteraciones con cambio en la cobertura vegetal $\ge50\%$
+- **5:** Detección provisional de alteraciones con cambio en la cobertura vegetal $\ge50\%$
+- **6:** Detección confirmada de alteraciones con cambio en la cobertura vegetal $\ge50\%$
+- **7:** Detección finalizada de alteraciones con cambio en la cobertura vegetal $<50\%$
+- **8:** Detección finalizada de alteraciones con cambio en la cobertura vegetal $\ge50\%$
+
+El valor del píxel particular que queremos marcar es 6, es decir, un píxel en el que se confirmó que por lo menos el 50% de la cubierta vegetal está dañada y en el que la alteración continúa activamente. Podemos utilizar el método `.sum` para sumar todos los píxeles con valor `6` y el método `.to_series` para representar la suma como una serie de Pandas indexada en el tiempo. También definimos `conversion_factor` que toma en cuenta el área de cada píxel en $\mathrm{km}^2$ (recordemos que cada píxel tiene un área de $30\mathrm{m}\times30\mathrm{m}$).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+pixel_val = 6
+conversion_factor = (30/1_000)**2 / pixel_val
+damage_area = stack.where(stack==pixel_val, other=0).sum(axis=(1,2))
+damage_area = damage_area.to_series() * conversion_factor
+damage_area
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot_title = 'Damaged Area (km²)'
+line_plot_opts = dict(title=plot_title, grid=True, color='r')
+damage_area.hvplot.line(**line_plot_opts)
+```
+
+
+
+Observando el gráfico anterior, parece que los incendios forestales comenzaron alrededor del 21 de agosto y se propagaron rápidamente.
+
+---
+
+
+
+### Visualización de fragmentos temporales seleccionados
+
+
+
+El bosque cercano de Dadia se vio especialmente afectado por los incendios. Para comprobarlo, trazaremos los datos ráster para ver la distribución espacial de los píxeles dañados en tres fechas concretas: 2 de agosto, 26 de agosto y 18 de septiembre. Una vez más, resaltaremos solamente los píxeles que tengan valor 6 en los datos ráster. Podemos extraer fácilmente esas fechas específicas de la serie temporal `by_day` utilizando una lista de fechas (por ejemplo, `dates_of_interest` en la siguiente celda).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+dates_of_interest = ['2023-08-01', '2023-08-26', '2023-09-18']
+print(dates_of_interest)
+snapshots = stack.sel(time=dates_of_interest)
+snapshots
+```
+
+
+
+Vamos a hacer una secuencia estática de los gráficos. Empezaremos definiendo algunas opciones estándar almacenadas en diccionarios.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ crs=crs,
+ shared_axes=False,
+ colorbar=False,
+ aspect='equal',
+ )
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+```
+
+
+
+Construiremos un mapa de colores utilizando un diccionario de valores RGBA (por ejemplo, tuplas con tres entradas enteras entre 0 y 255, y una cuarta entrada de punto flotante entre 0.0 y 1.0 para la transparencia).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+COLORS = { k:(0,0,0,0.0) for k in range(256) }
+COLORS.update({6: (255,0,0,1.0)})
+image_opts.update(cmap=list(COLORS.values()))
+```
+
+
+
+Como siempre, empezaremos por cortar imágenes más pequeñas para asegurarnos de que
+`hvplot.image` funciona correctamente. Podemos reducir el valor del parámetro `steps` a `1` o `None` para obtener las imágenes renderizadas en resolución completa.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 100
+subset = slice(0,None, steps)
+view = snapshots.isel(longitude=subset, latitude=subset)
+(view.hvplot.image(**image_opts).opts(**layout_opts) * basemap).layout()
+```
+
+
+
+Si eliminamos la llamada a `.layout`, podemos producir un desplazamiento interactivo que muestre el progreso del incendio forestal utilizando todos los rásteres en `stack`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 100
+subset = slice(0,None, steps)
+view = stack.isel(longitude=subset, latitude=subset,)
+(view.hvplot.image(**image_opts).opts(**layout_opts) * basemap)
+```
+
+---
diff --git a/book/es-419/04_Case_Studies/02_Lake_Mead_Mosaicking.md b/book/es-419/04_Case_Studies/02_Lake_Mead_Mosaicking.md
new file mode 100644
index 0000000..404c2bf
--- /dev/null
+++ b/book/es-419/04_Case_Studies/02_Lake_Mead_Mosaicking.md
@@ -0,0 +1,501 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.4
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Generación de un mosaico del lago Mead
+
+
+
+El [Lake Mead](https://es.wikipedia.org/wiki/Lago_Mead) es un embalse de agua que se encuentra en el suroeste de los Estados Unidos y es importante para el riego en esa zona. El lago ha tenido una importante sequía durante la última década y, en particular, entre 2020 y 2023. En este cuaderno computacional, buscaremos datos GeoTIFF relacionados con este lago y sintetizaremos varios archivos ráster para producir una visualización.
+
+***
+
+
+
+## Esquema de los pasos para el análisis
+
+
+
+- Identificar los parámetros de búsqueda
+ - AOI y ventana temporal
+ - _Endpoint_, proveedor, identificador del catálogo ("nombre corto")
+- Obtención de los resultados de búsqueda
+ - Exploracion, análisis para identificar características, bandas de interés
+ - Almacenar los resultados en un DataFrame para facilitar la exploración
+- Exploración y refinamiento de los resultados de la búsqueda
+ - Identificar los gránulos de mayor valor
+ - Filtrar los gránulos atípicos con contribución mínima
+ - Combinar los gránulos filtrados en un DataFrame
+ - Identificar el tipo de salida a generar
+- Procesamiento de los datos para obtener resultados relevantes
+ - Descargar los gránulos relevantes en Xarray DataArray, apilados adecuadamente
+ - Realizar los cálculos intermedios necesarios
+ - Unir los datos relevantes en una visualización
+
+
+
+***
+
+### Importación preliminar
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+import numpy as np, pandas as pd, xarray as xr
+import rioxarray as rio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+from bokeh.models import FixedTicker
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# STAC imports to retrieve cloud data
+from pystac_client import Client
+from osgeo import gdal
+# GDAL setup for accessing cloud data
+gdal.SetConfigOption('GDAL_HTTP_COOKIEFILE','~/.cookies.txt')
+gdal.SetConfigOption('GDAL_HTTP_COOKIEJAR', '~/.cookies.txt')
+gdal.SetConfigOption('GDAL_DISABLE_READDIR_ON_OPEN','EMPTY_DIR')
+gdal.SetConfigOption('CPL_VSIL_CURL_ALLOWED_EXTENSIONS','TIF, TIFF')
+```
+
+### Funciones prácticas
+
+
+
+Estas funciones podrían incluirse en archivos modulares para proyectos de investigación más evolucionados. Para fines didácticos, se incluyen en este cuaderno computacional.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle with given center of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding-box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to plot an AOI or bounding-box
+def plot_bbox(bbox):
+ '''Given bounding-box, returns GeoViews plot of Rectangle & Point at center
+ + bbox: bounding-box specified as (lon_min, lat_min, lon_max, lat_max)
+ Assume longitude-latitude coordinates.
+ '''
+ # These plot options are fixed but can be over-ridden
+ point_opts = opts.Points(size=12, alpha=0.25, color='blue')
+ rect_opts = opts.Rectangles(line_width=0, alpha=0.1, color='red')
+ lon_lat = (0.5*sum(bbox[::2]), 0.5*sum(bbox[1::2]))
+ return (gv.Points([lon_lat]) * gv.Rectangles([bbox])).opts(point_opts, rect_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# utility to extract search results into a Pandas DataFrame
+def search_to_dataframe(search):
+ '''Constructs Pandas DataFrame from PySTAC Earthdata search results.
+ DataFrame columns are determined from search item properties and assets.
+ 'asset': string identifying an Asset type associated with a granule
+ 'href': data URL for file associated with the Asset in a given row.'''
+ granules = list(search.items())
+ assert granules, "Error: empty list of search results"
+ props = list({prop for g in granules for prop in g.properties.keys()})
+ tile_ids = map(lambda granule: granule.id.split('_')[3], granules)
+ rows = (([g.properties.get(k, None) for k in props] + [a, g.assets[a].href, t])
+ for g, t in zip(granules,tile_ids) for a in g.assets )
+ df = pd.concat(map(lambda x: pd.DataFrame(x, index=props+['asset','href', 'tile_id']).T, rows),
+ axis=0, ignore_index=True)
+ assert len(df), "Empty DataFrame"
+ return df
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# utility to remap pixel values to a sequence of contiguous integers
+def relabel_pixels(data, values, null_val=255, transparent_val=0, replace_null=True, start=0):
+ """
+ This function accepts a DataArray with a finite number of categorical values as entries.
+ It reassigns the pixel labels to a sequence of consecutive integers starting from start.
+ data: Xarray DataArray with finitely many categories in its array of values.
+ null_val: (default 255) Pixel value used to flag missing data and/or exceptions.
+ transparent_val: (default 0) Pixel value that will be fully transparent when rendered.
+ replace_null: (default True) Maps null_value->transparent_value everywhere in data.
+ start: (default 0) starting range of consecutive integer values for new labels.
+ The values returned are:
+ new_data: Xarray DataArray containing pixels with new values
+ relabel: dictionary associating old pixel values with new pixel values
+ """
+ new_data = data.copy(deep=True)
+ if values:
+ values = np.sort(np.array(values, dtype=np.uint8))
+ else:
+ values = np.sort(np.unique(data.values.flatten()))
+ if replace_null:
+ new_data = new_data.where(new_data!=null_val, other=transparent_val)
+ values = values[np.where(values!=null_val)]
+ n_values = len(values)
+ new_values = np.arange(start=start, stop=start+n_values, dtype=values.dtype)
+ assert transparent_val in new_values, f"{transparent_val=} not in {new_values}"
+ relabel = dict(zip(values, new_values))
+ for old, new in relabel.items():
+ if new==old: continue
+ new_data = new_data.where(new_data!=old, other=new)
+ return new_data, relabel
+```
+
+***
+
+## Identificación de los parámetros de búsqueda
+
+
+
+Identificaremos un punto geográfico cerca de la orilla norte del [lago Mead](https://es.wikipedia.org/wiki/Lago_Mead), haremos un cuadro delimitador y elegiremos un intervalo de fechas que cubra marzo y parte de abril del 2023.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+lake_mead = (-114.754, 36.131)
+AOI = make_bbox(lake_mead, 0.1, 0.1)
+DATE_RANGE = "2023-03-01/2023-04-15"
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Optionally plot the AOI
+basemap = gv.tile_sources.OSM(width=500, height=500, padding=0.1)
+plot_bbox(AOI) * basemap
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+search_params = dict(bbox=AOI, datetime=DATE_RANGE)
+print(search_params)
+```
+
+***
+
+## Obtención de los resultados de búsqueda
+
+
+
+Como siempre, especificaremos el _endpoint_ de búsqueda, el proveedor y el catálogo. Para la familia de productos de datos DSWx son los siguientes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+ENDPOINT = 'https://cmr.earthdata.nasa.gov/stac'
+PROVIDER = 'POCLOUD'
+COLLECTIONS = ["OPERA_L3_DSWX-HLS_V1_1.0"]
+# Update the dictionary opts with list of collections to search
+search_params.update(collections=COLLECTIONS)
+print(search_params)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+catalog = Client.open(f'{ENDPOINT}/{PROVIDER}/')
+search_results = catalog.search(**search_params)
+```
+
+
+
+Convertimos los resultados de la búsqueda en un `DataFrame` para facilitar su lectura.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = search_to_dataframe(search_results)
+display(df.head())
+df.info()
+```
+
+
+
+Limpiaremos el DataFrame `df` de maneras estándar:
+
+- eliminando las columnas `start_datetime` y `end_datetime`,
+- renombrando la columna `eo:cloud_cover`,
+- convirtiendo las columnas a tipos de datos adecuados, y
+- asignando la columna `datetime` como el índice.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.datetime = pd.DatetimeIndex(df.datetime)
+df = df.drop(['start_datetime', 'end_datetime'], axis=1)
+df = df.rename({'eo:cloud_cover':'cloud_cover'}, axis=1)
+df['cloud_cover'] = df['cloud_cover'].astype(np.float16)
+for col in ['asset', 'href', 'tile_id']:
+ df[col] = df[col].astype(pd.StringDtype())
+df = df.set_index('datetime').sort_index()
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+display(df.head())
+df.info()
+```
+
+***
+
+## Exploración y refinamiento de los resultados de la búsqueda
+
+
+
+Podemos observar la columna `assets` para ver cuáles bandas diferentes están disponibles en los resultados devueltos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.asset.value_counts()
+```
+
+
+
+La banda `0_B01_WTR` es con la que queremos trabajar posteriormente.
+
+También podemos ver cuánta nubosidad hay en los resultados de nuestra búsqueda.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+df.cloud_cover.agg(['min','mean','median','max'])
+```
+
+
+
+Podemos extraer las filas seleccionadas del `DataFrame` usando `Series` booleanas. Específicamente, seleccionaremos las filas que tengan menos de 10% de nubosidad y en las que el `asset` sea la banda `0_B01_WTR`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+c1 = (df.cloud_cover <= 10)
+c2 = (df.asset.str.contains('B01_WTR'))
+b01_wtr = df.loc[c1 & c2].drop(['asset', 'cloud_cover'], axis=1)
+b01_wtr
+```
+
+
+
+Por último, podemos ver cuántos mosaicos MGRS diferentes intersecan nuestro AOI.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+b01_wtr.tile_id.value_counts()
+```
+
+
+
+Hay cuatro mosaicos geográficos distintos que intersecan este AOI en particular.
+
+***
+
+
+
+## Procesamiento de los datos para obtener resultados relevantes
+
+
+
+Esta vez, utilizaremos una técnica llamada _creación de mosaicos_ para combinar datos ráster de mosaicos adyacentes en un conjunto único de datos ráster. Esto requiere la función `rasterio.merge.merge` como antes. También necesitaremos la función `rasterio.transform.array_bounds` para alinear correctamente las coordenadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import rasterio
+from rasterio.merge import merge
+from rasterio.transform import array_bounds
+```
+
+
+
+Ya hemos utilizado la función `merge` para combinar distintos conjuntos de datos ráster asociados a un único mosaico MGRS. En esta ocasión, los datos ráster combinados provendrán de mosaicos MGRS adyacentes. Al llamar a la función `merge` en la siguiente celda de código, la columna `b01_wtr.href` se tratará como una lista de URL ([Localizadores Uniformes de Recursos](https://es.wikipedia.org/wiki/Localizador_de_recursos_uniforme) (URL, por su siglas en inglés de _Uniform Resource Locator_)). Para cada URL de la lista, se descargará y se procesará un archivo GeoTIFF. El resultado neto es un mosaico de imágenes, es decir, un ráster fusionado que contiene una combinación de todos los rásteres. Los detalles del algoritmo de fusión se describen en la [documentación del módulo `rasterio.merge`](https://rasterio.readthedocs.io/en/latest/api/rasterio.merge.html).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+mosaicked_img, mosaic_transform = merge(b01_wtr.href)
+```
+
+
+
+La salida consiste de nuevo en un arreglo de NumPy y una transformación de coordenadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f"{type(mosaicked_img)=}\n")
+print(f"{mosaicked_img.shape=}\n")
+print(f"{type(mosaic_transform)=}\n")
+print(f"{mosaic_transform=}\n")
+```
+
+
+
+Las entradas de `mosaic_transform` describen una [_transformación afín_](https://es.wikipedia.org/wiki/Transformaci%C3%B3n_af%C3%ADn) de coordenadas de píxel a coordenadas UTM continuas. En particular:
+
+- la entrada `mosaic_transform[0]` es el ancho horizontal de cada píxel en metros, y
+- la entrada `mosaic_transform[4]` es la altura vertical de cada píxel en metros.
+
+Observa también que, en este caso, `mosaic_transform[4]` es un valor negativo (es decir, `mosaic_transform[4]==-30.0`). Esto nos dice que la orientación del eje vertical de coordenadas continuas se opone a la orientación del eje vertical de coordenadas de píxeles, es decir, la coordenada continua vertical disminuye en dirección descendente, a diferencia de la coordenada vertical de píxeles.
+
+Cuando pasamos el objeto `mosaic_transform` a la función `rasterio.transform.array_bounds`, el valor que se devuelve es un cuadro delimitador, es decir, una tupla de la forma `(x_min, y_min, x_max, y_max)` que describe las esquinas inferior izquierda y superior derecha de la imagen en mosaico resultante en coordenadas continuas UTM.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+bounds = array_bounds(*mosaicked_img.shape[1:], mosaic_transform)
+
+bounds
+```
+
+
+
+La combinación de toda la información anterior nos permite reconstruir las coordenadas UTM continuas asociadas a cada píxel. Computaremos arreglos para estas coordenadas continuas y las etiquetaremos como `longitude` y `latitude`. Estas coordenadas serían más precisas si se llamaran `easting` y `northing`, pero utilizaremos las etiquetas `longitude` y `latitude` respectivamente cuando adjuntemos los arreglos de coordenadas a un Xarray `DataArray`. Elegimos estas etiquetas porque, cuando los datos ráster se visualizan con `hvplot.image`, la salida utilizará coordenadas longitud-latitud.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+longitude = np.arange(bounds[0], bounds[2], mosaic_transform[0])
+latitude = np.arange(bounds[3], bounds[1], mosaic_transform[4])
+```
+
+
+
+Almacenamos la imagen en mosaico y los metadatos relevantes en un Xarray `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = xr.DataArray(
+ data=mosaicked_img,
+ dims=["band", "latitude", "longitude"],
+ coords=dict(
+ longitude=(["longitude"], longitude),
+ latitude=(["latitude"], latitude),
+ ),
+ attrs=dict(
+ description="OPERA DSWx B01",
+ units=None,
+ ),
+ )
+raster
+```
+
+
+
+Necesitamos adjuntar un objeto CRS al objeto `raster`. Para ello, utilizaremos `rasterio.open` para cargar los metadatos relevantes de uno de los gránulos asociados a `b01_wtr` (deberían ser los mismos para todos estos archivos).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(b01_wtr.href[0]) as ds:
+ crs = ds.crs
+
+raster.rio.write_crs(crs, inplace=True)
+print(raster.rio.crs)
+```
+
+
+
+En el código de investigación, podríamos agrupar los comandos anteriores en una función y guardarla en un módulo. No lo haremos aquí porque, para los propósitos de este tutorial, es preferible asegurarse de que podemos analizar la salida de varias líneas de código interactivamente.
+
+Con todos los pasos anteriores completados, estamos listos para producir una visualización del mosaico. Reetiquetaremos los valores de los píxeles para que la barra de colores del resultado final sea más prolija.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster, relabel = relabel_pixels(raster, values=[0,1,2,253,254,255])
+```
+
+
+
+Vamos a definir las opciones de imagen, las opciones de diseño, y un mapa de color en los diccionarios como lo hicimos anteriormente para generar una única visualización.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ crs=raster.rio.crs
+ )
+layout_opts = dict(
+ xlabel='Longitude',
+ ylabel='Latitude',
+ )
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Define a colormap using RGBA values; these need to be written manually here...
+COLORS = {
+0: (255, 255, 255, 0.0), # No Water
+1: (0, 0, 255, 1.0), # Open Water
+2: (180, 213, 244, 1.0), # Partial Surface Water
+3: ( 0, 255, 255, 1.0), # Snow/Ice
+4: (175, 175, 175, 1.0), # Cloud/Cloud Shadow
+5: ( 0, 0, 127, 0.5), # Ocean Masked
+}
+
+c_labels = ["Not water", "Open water", "Partial Surface Water", "Snow/Ice",
+ "Cloud/Cloud Shadow", "Ocean Masked"]
+c_ticks = list(COLORS.keys())
+limits = (c_ticks[0]-0.5, c_ticks[-1]+0.5)
+
+c_bar_opts = dict( ticker=FixedTicker(ticks=c_ticks),
+ major_label_overrides=dict(zip(c_ticks, c_labels)),
+ major_tick_line_width=0, )
+
+image_opts.update({ 'cmap': list(COLORS.values()),
+ 'clim': limits,
+ })
+
+layout_opts.update(dict(colorbar_opts=c_bar_opts))
+```
+
+
+
+Definiremos el mapa base como un objeto separado para superponerlo usando el operador `*`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.ESRI(frame_width=500, frame_height=500, padding=0.05, alpha=0.25)
+```
+
+
+
+Por último, podemos utilizar la función `slice` de Python para extraer rápidamente las imágenes reducidas antes de tratar de ver la imagen completa. Recuerda que reducir el valor de `steps` a `1` (o `None`) visualiza el ráster a resolución completa.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+steps = 1
+view = raster.isel(longitude=slice(0,None,steps), latitude=slice(0,None,steps)).squeeze()
+
+view.hvplot.image(**image_opts).opts(**layout_opts) * basemap
+```
+
+
+
+Este ráster es mucho más grande de los que analizamos anteriormente (requiere aproximadamente 4 veces más espacio de almacenamiento). Este proceso podría ser iterado para hacer un deslizador que muestre los resultados fusionados de mosaicos vecinos en diferentes momentos. Esto, por supuesto, requiere que haya suficiente memoria disponible.
+
+***
+
+


 +
+ +
+