情報・システム研究機構 データサイエンス共同利用基盤施設 ライフサイエンス統合データベースセンター
客員教授 坊農 秀雅 [email protected]
2021年7月15日 13:40-15:20
これは統合データベース講習会AJACSオンライン7「ゲノムデータベース/ゲノムブラウザを使って配列解析と遺伝子機能解析を行う」の資料です。
© 2021 DBCLS, licensed under Creative Commons Attribution 4.0 International license (CC-BY-4.0)
本講習は、だれでも自由に使うことができる公共DBやウェブツールを活用した、ゲノムデータベースやゲノムブラウザを使って配列解析と遺伝子機能解析を行う方法と基礎知識について学びます。 とくに、ゲノムブラウザとBLASTの実際の使い方をデモンストレーションしながら詳しく説明、実習します。
今回の講習では、以下の内容について順番に説明します。 【実習】は、講習会でデモンストレーションする内容を、【応用】は全ては講習会ではデモンストレーションしない発展的な内容となっています。
- 研究現場で頻繁に使われるDBやツールを知る: 統合TV
- ゲノムデータベースとゲノムブラウザ
- NCBI
- Genome Data Viewer
- Genome Information by Organism
- 【実習1】UCSC Genome Browser
- 【応用1】Ensembl Genome Browser
- NCBI
- 塩基配列データベース
- 国際塩基配列データベース共同研究(INSDC)
- DDBJ/EMBL/GenBank
- Sequence Read Archive (SRA)
- RefSeq
- 国際塩基配列データベース共同研究(INSDC)
- 配列解析入門
- BLAST概説
- 【実習2】BLAST
- 【応用2】BLAT
- 入門者向け
- 生命科学データベース・ウェブツール 坊農秀雅、小野浩雅 (2018)
- Dr. Bonoの生命科学データ解析 第2版 坊農秀雅 (2021)
- 初中級者向け
- 生命科学者のためのデータ解析実戦道場 坊農秀雅 (2019)
- みんなで同時にアクセスするとサイトにつながりにくくなることが予想されます。
- 資料を見ながら自力で進められそうな方はどんどん先に、そうでない方は動画と一緒にすすめていきましょう。
- サイトの反応が悪い時はタイミングをずらして実行してみてください。
- 反応が無いからと言って何度もクリックするとますます繋がらなくなってしまいます。おおらかな気持ちで臨みましょう。
- わからないことがあったらslidoにてお知らせください。
- 遠慮は無用です(そのための講習会です!)。おいてけぼりは楽しくありません。
- 生命科学分野の有用なデータベースやツールの使い方を動画で紹介するウェブサイト
https://togotv.dbcls.jp/- ウェブサイトへのアクセスから結果の見方まで、操作の一挙手一投足がわかります。
- 講義・講習などの参考資料や後輩指導の教材として利用できます。
- 今回の講習に関連する内容の多くは、「塩基配列の検索、取得、比較を行う」にあります。
- 過去の講習会の内容はそのほとんどが統合TVに収録されており、いつでもどこでも繰り返し復習できるようになっています。
- お探しの動画が見つからない or 統合TV未掲載の場合は、統合TV番組リクエストフォームへどうぞ!
- 統合TVを作ってくれる方、募集中!!
- お探しの動画が見つからない or 統合TV未掲載の場合は、統合TV番組リクエストフォームへどうぞ!
- ゲノム配列を始めとした(遺伝)情報を生物種ごとにまとめたデータベース
- 狭義にはゲノム配列のデータベースをいう
- NCBI (National Center for Biotechnology Information)
- Genome Data Viewer
- 例: Naked mole-rat
- 生物種ごと(Browse by Organism)
- Genome Data Viewer
- 各種モデル生物種ごとに色々と
- 例: ハエのFlyBase、ヒト病原体を媒介する無脊椎動物のVectorBaseなどなど
- それ以外の代表的なものは以下のゲノムブラウザのセクションにて
- 塩基配列解読したゲノム配列とそこに付与(アノテーション)された情報を見るための仕組み
- オンライン型とローカル型
- オンライン型:ウェブブラウザ上で。サーバにあるゲノムデータベースから必要な情報を取り出してこれる
- UCSC Genome Browser
https://genome.ucsc.edu/ - Ensembl Genome Browser
https://www.ensembl.org/ - NCBI Genome Data Viewer
https://www.ncbi.nlm.nih.gov/genome/gdv/
- UCSC Genome Browser
- ローカル型:手元のコンピュータにインストールして使用
- Integrative Genomics Viewer(IGV)
https://software.broadinstitute.org/software/igv/
- Integrative Genomics Viewer(IGV)
- オンライン型:ウェブブラウザ上で。サーバにあるゲノムデータベースから必要な情報を取り出してこれる
- "UCSC Genome Browser" でググって、そのトップページを開く(もしくは直接
https://genome.ucsc.edu/にアクセス)。 - トップページにはツール名がリストされている。Our toolsの一番上にある "Genome Browser" をクリックする。
- 最寄りのミラーサイトに接続するか訊いてくるので、指示に従う。
- Genome Browserのページが開くので、生物種(Human)とアッセンブリ(Dec.2013/(GRCh38/hg38))を選んで、検索語を入力する。ここでは、
ACE2と入力。 - ACE2遺伝子のゲノム領域が表示される。
- "Regulation"のENCODE Regulation Trackをクリック。"TF Clusters"を"hide"から"pack"に変更して、"submit"ボタンを押す。
- 転写因子結合サイトの情報が追加される。
- 転写開始点領域を拡大して表示する、情報をたくさん出るように"pack"を"full"に変更するなど、いろいろ変更して表示してみましょう。
わからなくなったら、図の下に並んでいるボタンの"default tracks"を押すと最初の状態に戻せます。
- 表示できるアノテーションを調べる 2018
https://doi.org/10.7875/togotv.2018.124 - 様々な目的の配列を取得する
https://doi.org/10.7875/togotv.2017.098
- 様々な組織、細胞における遺伝子発現データをゲノムブラウザで表示する
https://doi.org/10.7875/togotv.2017.111 - UCSC Track Hubs を使って大規模な公共データをゲノムブラウザで閲覧する
https://doi.org/10.7875/togotv.2019.174 - UCSC Table Browserを使って多様なアノテーショントラックからデータを絞り込み閲覧・取得する
https://doi.org/10.7875/togotv.2021.051
- Ensembl Genome Browser
https://www.ensembl.org/- 脊椎動物のゲノムを対象としたゲノムブラウザ
- 比較ゲノム、進化、配列変異、転写制御などの研究をサポート
- Ensembl Genome Browser でも上記のACE2を検索してみよう。
- EnsemblGenomes
https://ensemblgenomes.org/- 脊椎動物以外のゲノムDB
- EnsemblPlants (植物)
- EnsemblMetazoa (後生動物)
- EnsemblProtists (原生生物)
- EnsemblFungi (菌類)
- EnsemblBacteria (細菌)
- 脊椎動物以外のゲノムDB
- 配列を取得する
https://doi.org/10.7875/togotv.2017.046 - 塩基配列のアラインメントを作成し生物種間で比較する 2019
https://doi.org/10.7875/togotv.2019.106 - 過去のバージョンのゲノムアノテーションを調べる
http://doi.org/10.7875/togotv.2017.088 - 遺伝子の場所や周辺情報を調べる
http://doi.org/10.7875/togotv.2017.082
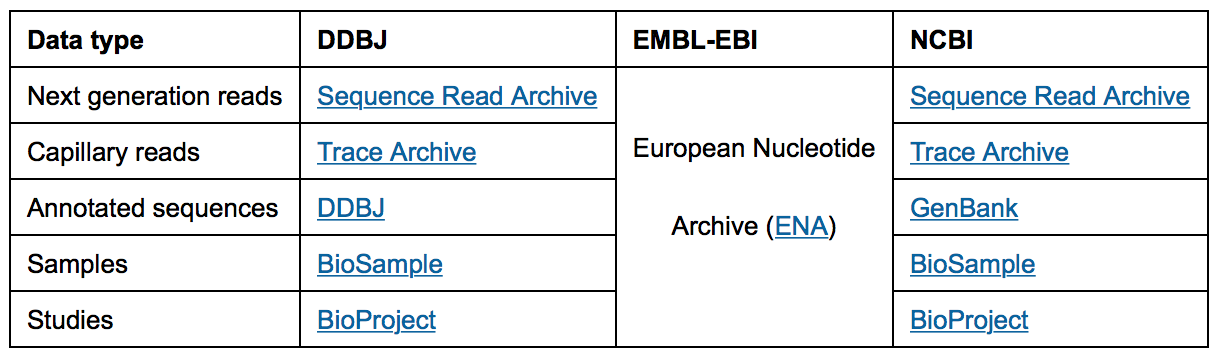
塩基配列(DNAやRNA)のデータベースで、上述のゲノムデータベースやゲノムブラウザの元になっているデータがここに一次ソースとしてバンク(アーカイブ)されている。
-
International Nucleotide Sequence Database Collaboration (INSDC) によって、1987年より日欧米の3箇所で塩基配列の登録、データの交換、維持管理を行っている。
- つまり、どこに登録してもデータは他の2つに反映される
-
日本では、国立遺伝学研究所のDDBJセンターに
- 日本語での対応をしてもらえる
-
INSDC DBのTable (http://www.insdc.org/ より)
-
アクセッション番号
- INSDの登録(accession)番号(例:
AB016472.1)- 論文掲載の必須条件
- データを他の研究者に再利用してもらうことが研究の価値を高める上でとても大事
- 末尾の
.数字はバージョン情報
- 日本だとDDBJへ。日本語でのやりとり可
- INSDの登録(accession)番号(例:
- 今はAnnotated sequencesと呼ばれている昔(1990年ごろ)からあるDB
- SRAデータも交換されている
- DDBJにもあるので、そこから取ると早くダウンロードできる。
- DDBJではDDBJ Sequence Read Archive (DRA)と呼ばれることも。
- 次世代シークエンサーのデータは以下の3つに収められている
NCBIの上述以外のDB(GEOやPubMedなど)はINSDCの枠外=データ交換などがなされていないので、個別に検索を実行する必要がある。
- DDBJ/EMBL/GenBankに登録されたデータを NCBIが独自に curationした二次データベース
- 例:
NM_001043619.1 - RefSeqはINSDには入ってないが、DDBJからもリンクしているDBCLS謹製のGGRNAを使うと検索可能
- 例:
- 【統合TV】 遺伝子のRefSeq IDを調べ、そのmRNA、アミノ酸配列を取得する
http://doi.org/10.7875/togotv.2017.086
- 2021年7月8日公開
- 生命科学分野におけるデータベース(DB)のID間の対応関係を検索および変換することができるウェブアプリケーション
- 例: 生命科学者のためのデータ解析実戦道場 p76にあるIDリスト
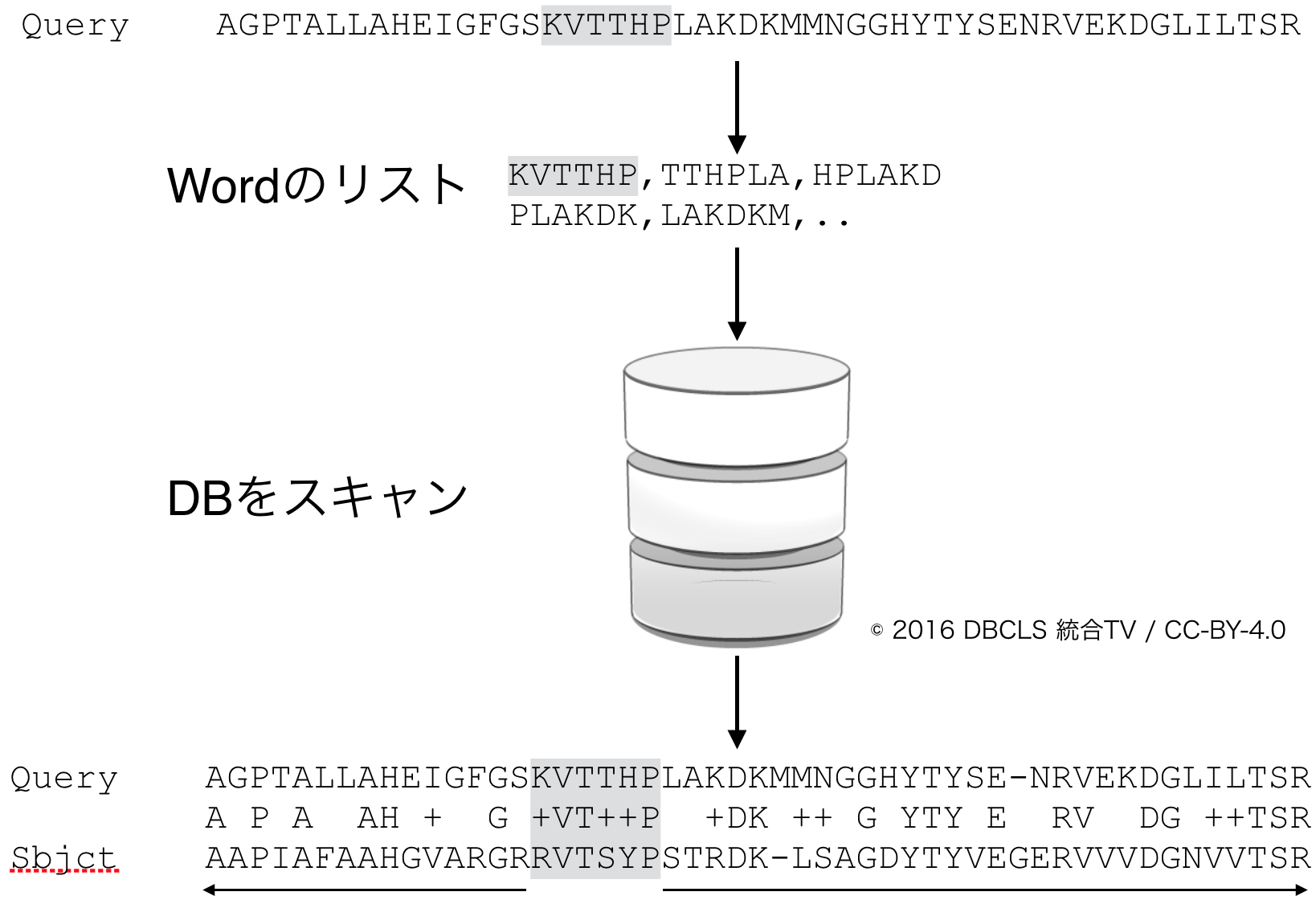
配列解析の基本である配列アラインメントについて、BLASTを例にその検索アルゴリズムを解説する。
色々あるが多くがテキストファイル。
| ファイルフォーマット | ファイル拡張子 | 用途など |
|---|---|---|
| FASTA | .fa .fasta | 塩基配列、アミノ酸配列 |
| FASTQ | .fq .fastq | NGSからの塩基配列とそのquality |
| DDBJ(Genbank) | .dbj (.gbk) | メタデータを含んだ塩基配列やアミノ酸配列の記述 |
| SRA | .sra | FASTQを圧縮したファイル形式 |
| SAM/BAM | .sam .bam | リファレンスゲノム配列へのアラインメント |
| GFF(GTF) | .gff .gtf | ゲノムアノテーション |
| BED | .bed | ゲノムアノテーション |
| VCF | .vcf | バリアントの記述 |
詳細は、Dr. Bonoの生命科学データ解析 第2版 坊農秀雅 (2021) の3.3 生命科学分野で使われたきたデータ形式(p96-122) などを参照
- 大域的アラインメント(Global alignment)
- 配列中の全塩基(アミノ酸)がアラインされるようにしたもの
- Needleman-Wunsch algorithm
- 配列中の全塩基(アミノ酸)がアラインされるようにしたもの
- 局所的アラインメント(Local alignment)
- 部分的な類似が見つけられるようにしたもの
- Smith-Waterman algorithm
- Goad-Kanehisa algorithm
- 配列類似性検索へ応用
- 部分的な類似が見つけられるようにしたもの
- なぜ相同性じゃなく類似性か
- BLASTとは
- Basic Local Alignment Search Tool
- 配列類似性検索のデファクトスタンダード
- BLASTの動作原理
- 質問配列(Query)
- 検索対象DB(Sbjct)
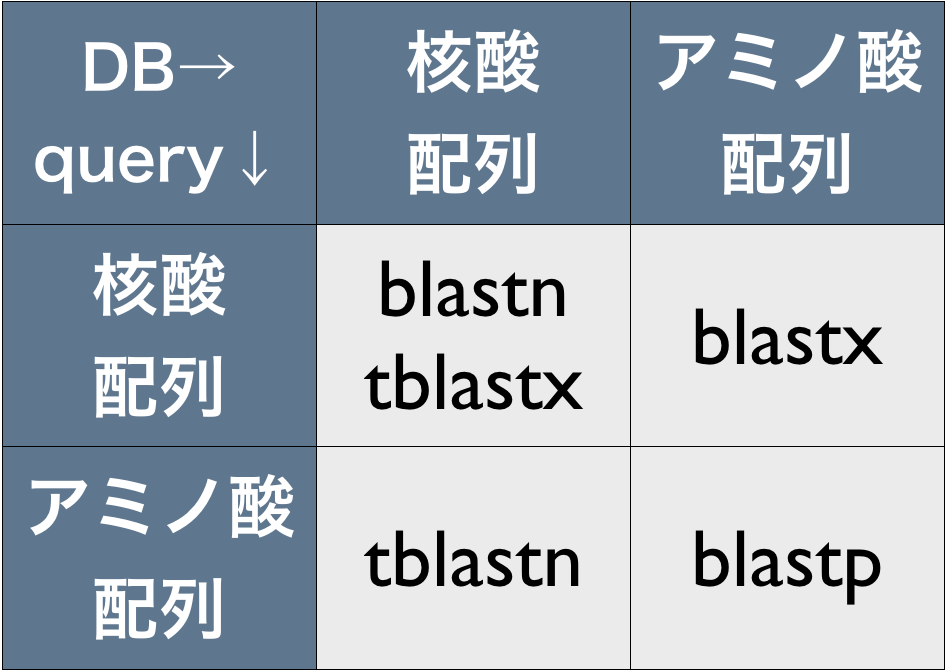
- 質問配列とDBの組み合わせ→使うプログラム名が異なる
- blastnだけが核酸配列レベルでの比較
- 残り全てはアミノ酸配列レベルの比較
- "NCBI BLAST" でググって、そのトップページを開きます
- Nucleotide BLASTを選びましょう
- 検索する配列(query)をFASTA形式で入力する。興味ある配列がない場合は、例1(カイコのエノラーぜ塩基配列)を使ってみましょう。
- 検索対象DBを選びます。まずは、デフォルトのまま(nr/nt)で。
- BLASTボタンを押すと、検索開始。混んでる時は時間がかかりますが、しばらく待つと結果が得られますので、忍耐強く待ちましょう。
- BLASTの結果の見方。Graphical SummaryやTaxonomy。Other reportsとして、Distance tre e of results と MSA viewer。
- 検索対象DBを'Human RefSeqGene Sequence (RefSeq_Gene)'にするなど、パラメータを変えて検索してみよう。
- 検索配列(query)にタンパク質(アミノ酸)配列を使ってみましょう。興味ある配列がない場合は、例1(カイコのエノラーぜアミノ酸配列)を使ってみましょう。
- 検索対象DBはnrかUniProtKBを選んでBLASTしましょう。
- 結果が得られたらじっくり精査して、考察しましょう。DBを変えるとどう変わるでしょうか?
- NCBI BLASTの使い方〜基本編〜2017
https://doi.org/10.7875/togotv.2017.023
- 大量にBLAST検索する際には自分のパソコンにインストールして使う(localBLAST)
- Local BLASTの使い方〜導入・準備編(MacOSX版)〜
https://doi.org/10.7875/togotv.2020.094 - Local BLASTの使い方〜検索実行・オプション(MacOSX版)〜
https://doi.org/10.7875/togotv.2021.019
- Local BLASTの使い方〜導入・準備編(MacOSX版)〜
- UNIXのコマンドライン操作が必要に。参考図書のデータ解析実戦道場(道場本)の3.2章参照。
- BLASTの誤表記ではない。The BLAST-like alignment toolで、BLAT
- 検索対象DBがゲノム配列に特化した配列類似性検索
- それゆえ、genome landing toolとも呼ばれる
- 企業には有償のライセンス
- 依然としてBLASTを使う例も多く
- BLASTと同じ検索を、BLATを用いてやってみよう。統合TVを参考に。
- 【統合TV】UCSC BLATを使って、ウイルスの持ち出した宿主の遺伝子配列がコードされている領域をアミノ酸配列レベルでゲノム中から探し当てる
https://doi.org/10.7875/togotv.2017.124
- 【統合TV】UCSC BLATを使って、ウイルスの持ち出した宿主の遺伝子配列がコードされている領域をアミノ酸配列レベルでゲノム中から探し当てる